Professional Documents
Culture Documents
Lotta Biologica in Agricoltura
Uploaded by
pedro41Copyright
Available Formats
Share this document
Did you find this document useful?
Is this content inappropriate?
Report this DocumentCopyright:
Available Formats
Lotta Biologica in Agricoltura
Uploaded by
pedro41Copyright:
Available Formats
I
CAPITOLO 1
1.1. - Introduzione La popolazione umana ha superato, nel 1999 i sei miliardi di individui e considerando il tasso odierno di crescita annuale, si prevede il suo raddoppiamento nei prossimi cinquantanni (Weber, 1993). Laumento incontrollato del numero mondiale di abitanti ha avuto delle ripercussioni molto serie sul problema dellalimentazione e sulla necessit di aumentare le rese produttive delle colture agrarie al fine di garantire a tutti il fabbisogno minimo giornaliero di calorie. Stimolata da tale esigenza, lagricoltura si modificata abbandonando progressivamente le antiche tecniche di coltivazione per abbracciare un insieme di mezzi pi moderni in grado di ottimizzare le produzioni agricole. Le nuove tecniche colturali, i fertilizzanti chimici, i fitofarmaci rappresentarono per i coltivatori strumenti agricoli fondamentali. Ma labuso di tali innovazioni cre non pochi problemi. Leccessivo impiego di fertilizzanti per aumentare le rese (Palti, 1981), la somministrazione dei fitofarmaci sistemici, finalizzato alleradicazione dei patogeni (Risch, 1987), lomogeneit delle cultivar utilizzate (Altieri e Letourneau, 1982), e una non controllata gestione dellirrigazione (Matta et al., 1996) portarono, ben presto, a nuovi e pi gravi problemi di tipo fitopatologico e ad un aumento dellinquinamento ambientale. Per combattere le malattie fu pressoch scontato il ricorso massivo a fitofarmaci. Per esempio luso ripetuto di fungicidi a meccanismo dazione specifico, in particolare benzimidazoli, port negli anni 60 alla comparsa di popolazioni di patogeni resistenti (Gullino, 1987). Ci spinse, in generale, i coltivatori ad aumentare le dosi di trattamento con evidenti effetti sullambiente: inquinamento delle falde acquifere per lisciviazione dei composti, effetti sulla salute legati alla tossicit dei principi attivi presenti come residui negli alimenti e lo squilibrio della microfauna utile del terreno. Le organizzazioni mondiali, lopinione pubblica, le unioni di consumatori aiutati da dati sempre pi sconfortanti riguardanti la situazione del pianeta terra, hanno avuto in passato e continuano ad avere,
oggi, un ruolo cruciale nel denunciare tali pericoli. Non senza passaggi intermedi, si passati, da un modello di agricoltura che mirava alleradicazione del patogeno con una lotta tout court, ad un modello dinamico globale basato sulla conoscenza, il pi possibile completa, della coltivazione Lagricoltura contemporanea si cosi evoluta verso un modello di tipo gestionale integrato (Fry, 1982). Gli agricoltori hanno a disposizione oggi programmi di gestione integrata complessi. Nei disciplinari di difesa sono affiancati ai mezzi chimici, i mezzi agronomici, genetici e biologici (Van Lenteren et al., 1992). La lotta biologica si inserisce nellottica di unagricoltura che salvaguardi maggiormente lambiente. 1.2. - Lotta biologica In passato la definizione di lotta biologica (biological control) era riferita al contenimento biologico delle malattie; cio limitata al solo organismo utilizzato per combattere un patogeno. La definizione corrente pi complessa e si estende a tutte le misure biologiche attuabili per limitare lo sviluppo di parassiti animali e vegetali (Weber, 1993). Riguarda, escludendo luomo, limpiego di qualsiasi organismo, compresi i suoi prodotti: geni, enzimi, metaboliti, componenti cellulari (Chet, 1987). In questo capitolo tratter la lotta biologica sottolineando maggiormente gli aspetti legati alla difesa dalle malattie delle piante e alla lotta biologica condotta con antagonisti microbici, ma opportuno considerare che essa ha caratteristiche pi generali, che esplorano molteplici settori. Quindi occorrerebbe, per chiarezza espositiva, riferirla e descriverla, di volta in volta, in relazione ai differenti campi di interesse. In patologia vegetale la lotta biologica , pi specificamente, rivolta al contenimento dellazione parassitaria di agenti batterici e fungini delle piante. La malattia si pu formalizzare come risultato interattivo tra pianta o suscetto (S), patogeno (P) e ambiente (A) (Matta et al., 1996). Ma pi opportuno includere nellinterazione anche il concetto di tempo, che rende il modello dinamico. Gli antagonisti possono essere considerati come parte dellambiente
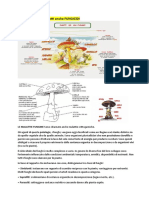
e quindi capaci di influenzare la pianta e il parassita. Per sviluppare validi agenti di lotta biologica dunque importante, conoscere in modo completo il sistema in cui si sviluppa la malattia. Occorre per cui conoscere non solo la biologia del patogeno e della pianta, ma anche i meccanismi di azione dei microrganismi antagonisti. Storicamente, sono stati evidenziati tre meccanismi di interazione che sono basilari per la comprensione della lotta biologica: lantagonismo diretto, l'antagonismo indiretto e lipovirulenza (Gullino, 1995). Nellantagonismo diretto un microrganismo si rapporta direttamente con un altro. Sono tipiche di tale modello le interazioni di antibiosi, competizione, predazione e iperparassitismo. Un microrganismo pu utilizzare una o pi modalit di antagonismo a seconda del sistema in cui si trova (Chet, 1987). Lantibiosi, fenomeno che si pu osservare spesso in natura, la produzione di metaboliti di origine microbica, capace di influenzare negativamente lo sviluppo di un altro microrganismo. Tali metaboliti possono essere molecole solubili in acqua, enzimi secreti, composti volatili (Fravel, 1988; Weller, 1988). Possono essere selettivamente tossici o avere uno spettro di tossicit molto ampio ed i siti di azione, di norma, possono essere molto eterogenei. E noto che molti filtrati colturali di origine microbica, siano in grado, quando addirittura non li prevengano, di attenuare i sintomi di una malattia. Pythium ultimum e Rhizoctonia solani sono contenuti da P. fluorescens e da Gliocladium virens (Howie e Suslov, 1986). Lantibiotico gliovirina sembra avere un ruolo preminente in tale fenomeno. Infatti, mutanti gliovirina negativi perdono la loro capacita antagonistica (Howell e Stipanovic, 1983). Sono stati individuati molti antibiotici in Pseudomonas spp. aventi un ruolo attivo nel contenimento di agenti patogeni (Fravel, 1988). Ceppi di P. fluorescens isolati da terreni repressivi verso Gaeumannomyces graminis f. sp. tritici riescono a contenere questultimo grazie alla produzione di fenazina (PCA) (Thomashow e Weller, 1988; Thomashow et al., 1990). Altri antibiotici giocano un ruolo cruciale e sinergico, come loomycin A (Gutterson et al., 1986 e Howie e Suslov, 1986), la pyoluteorina e il 2,4-diacetil fluoroglucinolo (Haas et al., 1991 e Keel et al., 1992). B. subtilis utilizzato nel contenimento di Monilinia
fructicola produce le iturine, peptidi ad attivit antifungina, attivi contro il marciume bruno delle pesche (Gueldner et al., 1988). P. cepacia produce fenilpirroli, nel contenimento della muffa grigia delle mele (Roitman et al., 1990). Lantibiosi un meccanismo di contenimento molto interessante e capace di conferire un elevato grado di contenimento nei confronti di determinati patogeni, ma la capacit di produrre antibiotici , per motivi legislativi, una caratteristica negativa, discriminante in fase di autorizzazione al rilascio nellambiente. Il secondo meccanismo di antagonismo, osservabile in natura, la competizione. Essa pu riguardare lo spazio, i siti di ingresso della pianta (ferite), e le sostanze nutritive nel senso pi ampio possibile. Un microrganismo patogeno per svilupparsi deve poter occupare i siti di infezione e prendere dallesterno le molecole di cui necessita. Durante le prime fasi di infezione, se presente un microrganismo che occupa tali siti e compete con il parassita per accaparrarsi le sostanze utili, lo sviluppo del patogeno e quindi il progredire della malattia rallentano. Normalmente i microrganismi antagonisti si adattano meglio dei patogeni alle variate condizioni ambientali. Si sviluppano pi rapidamente e in condizioni ambientali pi critiche (temperatura, umidit, pH) occupando pi rapidamente i siti dove avviene la malattia. Ceppi di Fusarium saprofiti isolati da terreni repressivi presenti nella piana di Albenga sono altamente competitivi nel terreno con diverse forme speciali di Fusaria patogeni, grazie alla grande capacit di colonizzare la rizosfera (Garibaldi et al., 1990) In terreni americani sono stati isolati delle pseudomonadi che agirebbero contro alcune forme speciali di F. oxysporum in virt della capacit di chelare il ferro grazie alla produzione di siderofori; in questo modo la germinazione delle clamidospore dei patogeni rallentata (Scher e Baker, 1982 ; Rovira et al., 1992) Nella lotta contro specie diverse di Penicillium, negli agrumi, Candida oleophila sembra agire competendo per le sostanze nutritive (Droby e Chalutz, 1994). Il ceppo US-7 di Pichia guilliermondii attivo contro Penicillium digitatum grazie allo stesso tipo di competizione. Nelle ferite sulle mele, Trichoderma compete
per i substrati nutritivi con B. cinerea (Dubos, 1997), mentre Metschnikowa pulcherrima compete specificamente per i nitrati (Piano et al., 1997). Tra i fenomeni di antagonismo diretto, non meno diffusi, ma a cui si guarda, per il futuro, con minore interesse c la predazione. Si tratta di un contenimento biologico operato da organismi animali, quali insetti, nematodi, protozoi e amebe. Nel pabulum alimentare di tali organismi rientrano a volte i parassiti delle piante. Pythium spp, agente di marciumi radicali limitato naturalmente dal nematode Aphelenchus avenae (Rhoades e Linford, 1959). Lameba Arachnula impatiens si ciba del micelio di molti funghi fitopatogeni mentre esistono funghi, quali Arthrobotrys, Dactylaria e Dactylella, che predano di nematodi grazie alla capacit di intrappolare con il micelio o di formare cappi costrittivi (Baker e Cook, 1974). In generale si pu, tuttavia, affermare che i fenomeni di predazione sono risultati poco sfruttabili dal punto di vista commerciale e che difficilmente troveranno un efficiente applicazione pratica. Una forma sui generis di predazione il micoparassitismo, detto anche iperparassitismo. Esistono micoparassiti biotrofi e necrotrofi: i primi instaurano rapporti trofici tramite strutture austoriali senza uccidere la vittima, altri uccidono la vittima e penetrano le ife del fungo fitopatogeno per cibarsi dei succhi cellulari. Tra i primi sono conosciuti alcuni funghi appartenenti agli Zygomycota (Piptocephalis, Dispira e Dimargalis spp.). Tra i micoparassiti necrotrofi sono noti Trichoderma spp. e G. virens (Papavizas, 1992), Sporidesmium sclerotivorum (Adams, 1990), Coniothyrium minitans (Whipps e Gerlagh, 1992), Pythium spp. (Deacon e Berry, 1992) e Ampelomyces quisqualis (Sztejnberg et al., 1989). Gliocladium roseum che per sembra agire sia per contatto sia per antibiosi. Talaromyces flavus invade gli sclerozi melanizzati di Verticillium dahliae presenti nelle radici malate e poi sporula sulla superficie di queste strutture. T. flavus produce lantibiotico talarone ma la sua azione parassita sembra essere promossa dallenzima secreto glucoso-ossidasi, che genera perossido di idrogeno (Kim et al., 1988). La maggior parte delle prove sperimentali, sul meccanismo di parassitismo riguardano per Trichoderma spp.. Il micoparassitismo pu essere suddiviso in fasi successive: individuazione,
riconoscimento, contatto, penetrazione, parassitismo e degenerazione della vittima. Allinizio le ife del fungo si sviluppano orientandosi verso la vittima, prendono contatto e formano degli appressori e si avvolgono allifa. In un secondo momento vengono prodotte chitinasi e (1,3)-glucanasi che permettono la penetrazione attiva nellifa.
2. - STRATEGIE DI LOTTA BIOLOGICA Una volta osservati i fenomeni naturali di contenimento biologico si possono applicare diverse strategie perch questi risultino utili per la difesa delle colture. E possibile, per interferire minimamente sullambiente, attuare strategie di gestione dei fenomeni naturali di contenimento delle malattie allinterno dellagroecosistema. Si pu sfruttare il fenomeno della resistenza indotta. Laddove ci sia una malattia si pu introdurre una componente microbica antagonistica e aiutarne linsediamento. 2.1. - Gestione dellagroecosistema La gestione degli equilibri presenti tra patogeni e microflora antagonistica consente di limitare i danni seppure presenti di una malattia. I fenomeni naturali, pi studiati, di contenimento delle malattie sono: i terreni repressivi e lipovirulenza. I terreni repressivi sono terreni inospitali nei confronti di uno o alcuni patogeni nonostante la coltivazione di ospiti suscettibili, le condizioni pedologiche e ambientali siano favorevoli allo sviluppo della malattia (Garibaldi e Gullino, 1989). Il concetto di repressivit opposto a quello di conduttivit: i terreni conduttivi non presentano fenomeni naturali di contenimento biologico. Gi dal 1892, negli USA era noto che la fusariosi del cotone portata da F. oxysporum f.sp. vasinfectum si manifestava con gravit maggiore negli stati con terreni sabbiosi piuttosto che in quelli con terreni argillosi.
Il fenomeno della repressivit venne inizialmente trascurato e solo allinizio del secolo successivo si incomici a intravedere un possibile sfruttamento. I primi studi furono fatti su terreni resistenti alla fusariosi del banano, presenti in America centrale, mettendo in correlazione la tessitura del terreno, la popolazione del patogeno e la repressivit. Il fenomeno del take-all decline la diminuzione della gravit degli attacchi di G. graminis f. sp. tritici sul grano, dopo alcuni anni di monocoltura fu messo in relazione con le componenti microbiologiche presenti nel terreno (Cook, 1981). Il primo approccio verso lo sfruttamento del fenomeno della repressivit rappresentato dalla possibilit di rendere repressivo un terreno condutttivo. Solo negli ultimi anni del secolo sono stati fatti studi mirati per identificare le cause specifiche che danno luogo alla repressivit. Le tipologie di terreni repressivi sono molteplici ma, di base essa pu essere legata a fenomeni biotici o abiotici. Riferendosi allazione dei microrganismi presenti nel terreno si pu definire una repressivit di tipo generale, operata dallazione della biomassa presente nel terreno e una repressivit di tipo specifico legata ad una componente microbica specifica presente nel terreno. La repressivit indotta da fattori abiotici pu essere legata dai valori di pH: in terreni della brughiera francese vicino a Paimpout lacidit del terreno correla con la riduzione dei danni da Phytophthora infestans. La repressivit di tipo biotico pu essere causata dallalta concentrazione dellantagonista: nella savana di Bogot lalto titolo di propaguli di Trichoderma spp. (108/g) responsabile della repressivit verso la rizottoniosi del garofano (Chet e Baker, 1981). Le iniziali applicazioni pratiche dei terreni repressivi erano basate sulla aggiunta di quote di terreno repressivo a terreni conduttivi. Tale pratica non si rivel di fatto perseguibile per la presenza relativamente limitata di terreni repressivi. E pi attuabile lisolamento dal terreno dei microrganismi responsabili della repressivit ed il loro inserimento in preparati commerciali allo scopo di arricchire i terreni conduttivi di popolazioni microbiche utili. Lipovirulenza un fenomeno naturale osservato in ambito forestale. Essa la ridotta capacit di parassitizzare la pianta ospite da parte di alcuni funghi fitopatogeni.
Cryphonectria parasitica lagente del cancro del castagno che colpisce in Italia, Francia e Usa. Ad una esplosione endemica della malattia che aveva decimato le popolazioni di castagno in Italia segu una drastica diminuzione della malattia. Il fenomeno fu in seguito spiegato grazie alla presenza di un ipovirus (CHV1) a RNA a doppio filamento. Lazione del virus potrebbe agire sulla subunit delle G-protein eterotrimeriche dal momento che in seguito allinfezione dellipovirus CHV1-EP713 si ha una diminuzione dei livelli di CPG1, una subunit G-protein cruciale per il ruolo della cellula e in seguito a mutazioni nel locus BDM-1 (beta disruption mimic factor-1),si ha la riduzione postrascrizionale di CPG-1 (Kasahara, et al. 2000). Ma la presenza di ipovirus non il solo fattore coinvolto nellipovirulenza. Recenti studi fatti su ceppi di C. parasitica esenti da virus hanno evidenziato il trasferimento orizzontale sia tramite contatto ifale sia conidico tra ceppi compatibili e incompatibili, seppure in maniera minore, di un DNA mitocondriale plasmidico pCRY1, capace di dare sintomi di senescenza, diminuzione di crescita, diminuzione della produzione conidica e alti livelli di respirazione tramite la via ossidativa cianuro-resistente (Baidyaroy et al., 2000) Lipovirus si trasmette tra ceppi di C. parasitica infetti ai sani tramite contatto. Le applicazioni tradizionali prevedevano linoculo nelle ferite di ceppi di ipovirus purificati. Tale strategia ha avuto effetto solo quando lipovirus utilizzato proveniente da gruppi di compatibilit europei veniva usato su funghi compatibili: esso non aveva alcuna utilit su ceppi americani appartenenti a diverso VCG. Le future applicazioni prevedono luso di ceppi chimerici che abbiano un pi ampio spettro dazione anche laddove intervenga il fenomeno dellincompatibilit vegetativa tra funghi (Chen e Nuss, 1999).
3. - STRUMENTI PER LA PRODUZIONE Quando un produttore possiede un microrganismo utile per la lotta biologica ha due possibilit concrete per trarne profitto: la brevettazione e la registrazione. Affinch possa vendere il microrganismo come prodotto fitosanitario obbligato
a registrarlo. Mentre si pu avvalere dellistituto brevettuale quando voglia proteggere il microrganismo come propriet intellettuale. Le due vie non sono alternative e la registrazione una conditio sine qua non la vendita del prodotto non sarebbe legalmente possibile.
4. - BREVETTAZIONE Il brevetto un documento tecnico-legale che descrive il contenuto di un'invenzione della quale si intende proteggere l'utilizzazione finale. Tramite il brevetto si concede un diritto esclusivo di utilizzo dell'invenzione e si assegna allinventore il privilegio di usufruire del profitto che ne deriva. Chi voglia utilizzare tali diritti privativi brevettando un microrganismo pu seguire due strade: il Brevetto Nazionale (italiano, francese, tedesco, etc.) o il Brevetto Europeo. I due brevetti sono molto simili, rifacendosi alle stesse normative ma presentano differenze per quanto riguarda lestensione territoriale della protezione e i costi. 4.1. - Cenni storici sui brevetti riguardanti microrganismi LIstituto brevettuale classico riguarda la tutela giuridica della propriet intellettuali e delle invenzioni. Occorre precisare la differenza tra invenzione e scoperta. Linvenzione qualcosa di nuovo alla cui identificazione partecipa lattivit inventiva delluomo, mentre la scoperta si riferisce al rinvenimento di qualcosa di nuovo che gi esiste in natura. Allo stato dellarte giuridico per brevettare un microrganismo si deve fare riferimento alla Convenzione di Monaco sul Brevetto Europeo (CBE) sancita il 5 ottobre 1973. La concessione del brevetto pu riguardare prodotti e procedimenti. La convenzione di Monaco stabilisce, dallarticolo 52 al 57 loggetto del brevetto. Esso riguarda ogni nuovo prodotto o procedimento in qualsiasi settore della tecnica, esclusi procedimenti terapeutici sul corpo umano o animale e nuove variet animali e vegetali ottenute con metodi essenzialmente biologici.
L'invenzione deve avere i caratteri di novit, attivit inventiva, industrialit e riproducibilit: -NOVITA: Essa deve essere assoluta, poich ogni divulgazione anteriore alla data di deposito della domanda di brevetto o alla data di priorit pu rendere nullo il brevetto -ORIGINALITA: il requisito che considera lattivit inventiva; essa deve sussistere al momento del deposito della domanda di brevetto a giudizio di un esperto del ramo che a conoscenza dello stato dellarte. -INDUSTRIALITA: il risultato dell'invenzione deve poter essere oggetto di applicazione industriale, cio utilizzabile nell'industria, inclusi i settori dei servizi e dell'agricoltura -RIPRODUCIBILITA e DESCRIZIONE SUFFICIENTE: un qualsiasi tecnico medio deve essere in grado di riprodurre il trovato, partendo dalla sua descrizione. Le invenzioni vengono suddivise in due categorie: invenzioni microbiologiche ed invenzioni macrobiologiche. Prendendo spunto dalla Convenzione di Strasburgo (24 marzo 1971) vengono dichiarati brevettabili i prodotti e i procedimenti microbiologici (art.53). Linterpretazione di tale articolo cambiata col tempo, grazie anche allinfluenza di decisioni brevettuali assunte negli USA, aprendo la strada al brevetto su tecniche biotecnologiche, microrganismi isolati in natura e microrganismi modificati geneticamente. Il trattato di Budapest del 28 aprile 1977, stabilisce in modo non equivoco il riconoscimento internazionale del deposito dei microrganismi, presso enti di deposito internazionali. 4.2. - Modalit per la costituzione di un brevetto Ogni persona fisica o giuridica italiana o straniera, inventore o suo cessionario, ha diritto a richiedere un brevetto europeo, presentando la domanda per la concessione, indipendentemente dall'esistenza di un precedente brevetto nazionale. La domanda va depositata presso una delle sedi dell'Ufficio Brevetti Europeo di Monaco (RFT) o presso l'Ufficio Brevetti nazionale di uno Stato aderente alla Convenzione Europea. Le lingue ufficiali, da utilizzare nella
10
procedura di fronte all'Ufficio Brevetti Europeo sono il francese, l'inglese e il tedesco. L'Ufficio Europeo effettua una ricerca di novit sull'invenzione che viene messa a disposizione del titolare e che serve di base per la successiva fase di esame, attivata dallo stesso titolare. L'Ufficio Europeo pu concedere o rifiutare la concessione del brevetto e la sua decisione ha effetto in ciascuno Stato membro. Contro il rifiuto di concessione si pu fare ricorso ad una Commissione di Appello interna all'Ufficio Europeo. Il Brevetto Europeo valido per ventanni e ha validit territoriale negli Stati membri a cui si vuole estendere la domanda. Il titolare della domanda dopo la pubblicazione della domanda europea, cio dopo 18 mesi dalla data di deposito o di priorit, pu conseguire la protezione provvisoria, negli Stati membri designati, attivando la relativa procedura. Conseguentemente il titolare della domanda pu agire nei confronti di terzi secondo le leggi dei rispettivi paesi e ha comunque diritto ad un equo compenso per l'attivit di tali terzi che, nel periodo tra la pubblicazione e la concessione del brevetto, comporti la violazione dei diritti di brevetto. Contro la concessione pu essere presentata un'opposizione da parte di terzi che viene discussa di fronte alle divisioni di opposizione dello stesso Ufficio Europeo. Alla fine della procedura, l'Ufficio pu confermare integralmente il brevetto concesso, modificarne la portata, annullare il brevetto. Contro tale decisione pu essere proposto appello ad una Commissione di Appello interna all'Ufficio Europeo. Dopo la concessione del brevetto, il titolare deve procedere alla sua convalida nazionale nei singoli Stati designati. Tale operazione in generale richiede una traduzione nella lingua di tale stato. Dopo la convalida il brevetto europeo regolato in ciascuno stato dalla legge nazionale. I documenti da presentare riguardano il testo della domanda di brevetto nazionale di cui si rivendica la priorit e la notifica dei paesi che si vogliono designare nella domanda di brevetto. Nel caso si tratti di un primo deposito si devono presentare la bozza di descrizione dell'invenzione preparata dall'inventore, lindicazione dello stato della tecnica a partire dal quale
11
l'invenzione stata realizzata, la bozza dei disegni, se previsti, il nome e dati anagrafici e fiscali del richiedente il brevetto e lidentit dell'inventore. 4.3. - Considerazioni Il limite del Brevetto Europeo quello di essere fondamentalmente un insieme di brevetti valevoli nei 19 stati membri firmatari della Convenzione di Monaco. Infatti tale istituto giuridico non offre una protezione omogenea in tutti gli stati. Il brevetto, adottato nei singoli paesi, rientra nei diversi quadri normativi e genera sperequazioni sulla protezione finale. Ad esempio, pu essere diverso il trattamento giuridico di chi trasgredisca alle norme di protezione, qualora alcuni Stati membri abbiano, in materia, norme penali diverse. In pi il procedimento molto costoso perch richiede la traduzione di tutto il fascicolo nelle lingue dei paesi in cui si vuole estendere la validit del brevetto. Gi da 1975 si sentita lesigenza di creare uno strumento nuovo, pi completo e pi efficiente, per disciplinare i brevetti a livello europeo. La Convenzione sul Brevetto Comunitario o Convenzione di Lussemburgo del 15 dicembre 1975, prevede la concessione di un brevetto unico avente identica efficacia sul territorio di tutti gli Stati membri dell'Unione Europea. Questa Convenzione nacque dall'esigenza di creare un unico brevetto comunitario e per aiutare la costituzione del mercato unico europeo, il quale, attraverso l'abbattimento di qualsiasi barriera doganale, potesse aumentare il livello di concorrenza e rendesse necessario tutelare le invenzioni al di l delle frontiere nazionali. Oggi lunione economica dellEuropa una realt ma la Convenzione di Lussemburgo non ancora entrata in vigore, n si possono fare previsioni attendibili circa i tempi necessari per la sua costituzione. In sua mancanza le procedure previste dalle Convenzioni di Monaco e dal PCT acquistano quindi una rilevanza ancor pi significativa. Ci soprattutto alla luce del sempre pi intenso processo di globalizzazione dell'economia che ormai coinvolge in modo rilevante anche le piccole e medie imprese.
12
5. - REGISTRAZIONE Uno dei maggiori problemi da superare per commercializzare un prodotto biologico la fase di registrazione. Alla base della registrazione la necessit di identificare in modo univoco il prodotto cos da differenziarlo da altri e per poterne seguire il destino nellambiente una volta rilasciato. Le tecniche utilizzabili sono molteplici e normalmente i criteri delezione sono la economicit, rapidit di esecuzione e la complessit intrinseca della tecnica. Ad oggi sono disponibili metodiche basate sullanalisi del DNA, delle proteine, degli acidi grassi della membrana e di tutte quelle caratteristiche fisiologiche e morfologiche che possano aiutare nella distinzione di un individuo microbico. Ma per la registrazione devono esser tenute in conto requisiti di tipo tossicologico. La registrazione di un microrganismo deputato a svolgere il ruolo di antagonista microbico segue differenti legislazioni a seconda che il prodotto venga certificato in Europa o negli USA. 5.1.- Registrazione negli USA Negli Stati Uniti, si attua una politica case by case, in cui cio le analisi richieste per la registrazione variano in base al microrganismo da immettere nellambiente e allo scopo con cui viene rilasciato. Il primo requisito da stabilire, per un prodotto fitosanitario la cui sostanza attiva sia un microrganismo, la unicit del ceppo utilizzato al fine di poter seguire il suo destino nello spazio e nel tempo una volta rilasciato nellambiente. La carta di identit del microrganismo pu essere creata con strumenti eterogenei e che forniscono, in ordine decrescente, un numero di informazioni inferiore: -lanalisi del DNA -lanalisi proteica -la tipizzazione degli acidi grassi della membrana
13
-lo studio dei metaboliti secondari (antibiotici, terpeni, fenoli, alcaloidi) -la caratterizzazione fisiologica e morfologica Verranno trattate successivamente le diverse metodologie accennate per tipizzare un microrganismo. Liter del prodotto durante la registrazione scandito da diverse fasi la cui importanza valutata in base a criteri tossicologici. Ogni prodotto che manifesti caratteristiche negative in fase 1 viene bloccato. I dati tossicologici, prodotti da laboratori indipendente dal gruppo richiedente la registrazione, vengono inviati allEPA (U.S. Enviromental Protection Agency) per una valutazione complessiva. Per quanto riguarda la valutazione dei potenziali effetti dannosi sullambiente lEPA sottopone il prodotto a 4 fasi di studio. Tali prove comprendono gli effetti tossici aspecifici e gli effetti patogenetici sulle piante e sulluomo. E importante sottolineare che in base al microrganismo da registrare il numero e il tipo di analisi cambia. Tale procedura, particolarmente snella, ha portato ad un grande divario tra il numero di microrganismi registrati negli USA e quelli registrati in Europa. 5.2. - Registrazione nellUE Per la registrazione di un prodotto fitosanitario contenente microrganismi con capacit antagonistiche, ci si deve riferire alla Direttiva Comunitaria 91/414/CEE, applicata in Italia con il Decreto Legislativo numero 194 del 17/5/95. Tale decreto disciplina: a) l'autorizzazione, l'immissione in commercio, l'utilizzazione ed il controllo dei prodotti fitosanitari presentati nella loro forma commerciale; b) l'immissione in commercio ed il controllo delle sostanze attive per luso cui sono destinate c) l'autorizzazione all'immissione in commercio di prodotti fitosanitari contenenti o costituiti da organismi geneticamente modificati, per i quali l'emissione
14
deliberata nell'ambiente sia gi stata approvata in base al decreto legislativo 3 marzo 1993, n. 92. Il decreto precisa la definizione del termine generale prodotto fitosanitario in cui vengono comprese: le sostanze attive ed i preparati contenenti una o piu' sostanze attive, presentati nella forma in cui sono forniti all'utilizzatore e destinati a: proteggere i vegetali o i prodotti vegetali da tutti gli organismi nocivi o a prevenirne gli effetti, favorire o regolare i processi vitali dei vegetali, con esclusione dei fertilizzanti, conservare i prodotti vegetali, con esclusione dei conservanti disciplinati da particolari disposizioni, eliminare le piante indesiderate, eliminare parti di vegetali, frenare o evitare un loro indesiderato accrescimento (art 2). Vengono definite sostanze attive: le sostanze o i microrganismi, compresi i virus, aventi un'azione generale o specifica sugli organismi nocivi o su vegetali, su parti di vegetali o su prodotti vegetali. Cos pure il decreto precisa che per preparati si intendono: le miscele o le soluzioni composte da due o piu' sostanze, delle quali almeno una sostanza attiva, destinate ad essere utilizzate come prodotti fitosanitari. Vengono elevati al ruolo di prodotto fitosanitario anche i microrganismi, permettendo il loro lutilizzo e disciplinando la loro registrazione e il controllo dopo la loro immissione. 5.3. - Autorizzazione alla sperimentazione La richiesta di autorizzazione alla sperimentazione deve essere inoltrata al Ministero della sanit, almeno 90 giorni prima dellinizio delle prove. Tale richiesta necessaria nel caso in cui le prove, comportino limmissione di un prodotto fitosanitario non ancora autorizzato o allo scopo di estendere lautorizzazione a nuovi impieghi di prodotti gi autorizzati. Lautorizzazione viene concessa, sentito il parere del Servizio Fitosanitario Centrale e dellAgenzia Nazionale per la protezione dellambiente, che devono dare il loro parere entro 45 giorni. Dopo tale periodo il Ministero della sanit puo procedere autonomamente. Il Ministero determina tutte le condizioni necessarie
15
per la prevenzione dei rischi per luomo, gli animali e lambiente, fissando i quantitativi massimi per lapplicazione sperimentale. Le derrate derivate dalle sperimentazioni devono essere mantenute separate dalle altre, smaltite secondo le normative vigenti o nel caso in cui si sia ottenuta lautorizzazione allimmissione al commercio, potenzialmente destinate allalimentazione umana. Tale decreto non si applica direttamente agli organismi geneticamente modificati se quelli che abbiano ottenuto un provvedimento formale di assenso per la sperimentazione e per lautorizzazione allimmissione in commercio (parte B e C della Direttiva 90/220 recepita con il D.Leg n.92 del 3/3/1993). 5.4. - Autorizzazione allimmissione in commercio Il decreto presenta nella sua parte finale sei allegati a cui il testo rimanda per la specifica dei singoli articoli della legge. I prodotti devono sottostare a determinate condizioni per lautorizzazione. Un prodotto fitosanitario pu essere autorizzato solo se: (A) le sostanze attive in esso contenute sono iscritte nellallegato I e se soddisfa i principi uniformi di cui allallegato VI; (B) se sufficientemente efficace; non produce effetti inaccettabili sui vegetali o sui prodotti vegetali; non provoca sofferenze e dolori inaccettabili ai vertebrati da combattere; non produce effetti nocivi in maniera diretta o indiretta, sulla salute dell'uomo o degli animali o sulle acque sotterranee; non produce effetti inaccettabili sull'ambiente, in particolare per quanto riguarda il suo destino e la sua distribuzione ambientale, con riferimento particolare alla contaminazione delle acque, comprese quelle potabili e sotterranee, nonche' l'impatto sulle specie non bersaglio; (C) se e' possibile determinare la natura e la quantita' delle sostanze attive in esso contenute e, ove occorra, delle sue impurezze e degli altri componenti significativi dal punto di vista tossicologico ed ecotossicologico, con adeguati metodi stabiliti in sede comunitaria o, in mancanza, riconosciuti dal Ministero della sanita'; (D) se possibile, con adeguati metodi di uso corrente, determinarne i residui di rilevanza tossicologica ed ambientale derivanti da un impiego autorizzato; (E) se le sue proprieta' fisico-chimiche sono state determinate e giudicate
16
accettabili per garantire un'utilizzazione ed un magazzinaggio adeguati; (F) se per i prodotti agricoli previsti dall'autorizzazione, i suoi residui non superano i limiti massimi stabiliti ai sensi dell'articolo 19 (art. 4). Il decreto disciplina il riconoscimento degli enti preposti allo svolgimento delle prove necessarie. Le condizioni viste sono accertate dai centri di saggio secondo lapplicazione dei principi di buona prassi di laboratorio(GLP) descritti dal decreto legislativo n120 del 27 gennaio 1992. Le prove ed analisi compiute dai centri di saggio devono ottemperare all norme previste nellallegato III del decreto. Lautorizzazione viene rilasciata dal Ministero della sanit ed ha una durata di dieci anni. Essa prescrive i requisiti di commercializzazione, di utilizzazione e di quelli necessari per ottemperare alle condizioni previste dallarticolo 4. Per richiedere lA.I.C. per un prodotto fitosanitario un produttore deve presentare la domanda di autorizzazione al Ministero della Sanit, un dossier che soddisfi i requisiti richiesti dallallegato III e un dossier per ogni sostanza attiva presente nel preparato, redatto secondo le modalit precisate nellallegato II. La domanda e i fascicoli allegati devono essere redatti in lingua italiana. La commissione consultiva (art. 20), lo strumento che il Ministero utilizza per verificare la conformit del prodotto con le condizioni stabilite dallarticolo 4. Tale commissione valuta avvalendosi della cooperazione dei richiedenti la domanda e procede al rilascio dellautorizzazione entro e non oltre un periodo di12 mesi dal momento in cui disponibile un dossier completo dal punto di vista tecnico. LA.I.C. viene notificata al titolare con il relativo numero di registrazione e vengono pubblicate sulla Gazzetta Ufficiale della Repubblica Italiana le etichette dei prodotti fitosanitari autorizzati. Presso il Ministero della Sanit viene conservata tutta la documentazione consegnata cos da renderla disponibile agli Stati membri e alla Commissione Europea. LA.I.C. pu essere rinnovata al termine della sua durata se la domanda di rinnovo viene presentata almeno un anno prima della sua scadenza, purch il
17
Ministero della Sanit accerti che non siano venute a mancare le condizioni imposte dallarticolo 4 del decreto. LA.I.C. non unautorizzazione statica ma pu essere modificata, con o senza il parere della commissione e sulla base della importanza delle modificazioni richieste (art.5 comma 13, 14, 15, 16). Essa pu essere riesaminata e ritirata, anche su richiesta del titolare quando vengano a mancare le condizioni viste allarticolo 4. Linformazione riguardante la riesamina e il ritiro sono di competenza del Ministero che si deve occupare di darne ampia pubblicit la titolare, agli organi di vigilanza e alle principali organizzazioni professionali di rivenditori e agricoltori affinch tutti gli operatori ne vengano a conoscenza. Dopo laccettazione della domanda di autorizzazione allimmissione in commercio, il prodotto fitosanitario autorizzato viene iscritto nellallegato I (art. 6, comma1) per un periodo di dieci anni e ogni modifica viene stabilita dal Ministero della Sanit. Liscrizione a tale allegato rimane vincolata a: (A) assenza di effetti nocivi sulluomo, sugli animali, sullambiente e sulle acque sotteranee, (B) rispetto della dose giornaliera per luomo, (C) livello ammissibile desposizione delloperatore, (D) stima del destino e della distribuzione nellambiente nonch dellimpatto sulle specie non bersaglio Ottenuta lAIC possibile per il titolare vendere il prodotto che prevedibilmente verr immesso nellambiente. Durante le fasi di registrazione la legislazione si occupa di regolamentare le modalit di accertamento dei rischi. 5.5. - Valutazione dellimpatto ambientale Il rilascio deliberato o accidentale nellambiente di un microrganismo ha delle implicazioni ambientali molto importanti. Dal punto di vista giuridico il decreto legislativo n.194 del 17/5/1995 definisce, allarticolo 4, le caratteristiche generali che un microrganismo deve possedere per salvaguardare lambiente, mentre nellallegato II, con riferimento allorganismo e nellallegato III, con riferimento al
18
prodotto fitosanitario, precisa le prove da effettuare prima che lAIC venga concessa. La legge si occupa di definire quali siano il destino e il comportamento nellambiente del prodotto, ponendo lattenzione sulla sua diffusione, mobilit, moltiplicazione e sulla persistenza nellaria, nellacqua e nel suolo. Dal punto ecotossicologico si occupa delle possibili interazioni tossiche con pesci, uccelli, lombrichi e insetti, cos come i possibili effetti negativi sulle api da miele sulle alghe, gli effetti su organismi utili, su altri organismi non bersaglio e sulla microflora del suolo. Tuttavia, il decreto legge non definisce, in modo preciso, un microrganismo mentre il precedente decreto legislativo n.91 del 3 marzo 1993, dava questa definizione di microrganismo: ogni entit microbiologica cellulare o non cellulare capace di replicarsi e di trasferire materiale genetico; se da una parte tale legge ammette che ci possa essere uno scambio di materiale genetico la legislazione sulla registrazione di microrganismi come prodotti fitosanitari non ne fa menzione, nel caso di microrganismi non modificati geneticamente, prevedendo tale eventualit solo per i MOGM. Dal punto di vista della stabilit genetica e del trasferimento orizzontale di geni non viene precisato nulla, considerando il fatto che ci sia un rischio effettivo per l'ambiente solo quando del materiale esogeno venga introdotto in un ospite o quando siano applicate tecniche che creino nuove combinazioni di materiale genetico ereditabile Nel caso della registrazione, le prove di valutazione del rischio sono quindi inferiori di numero e molto meno onerose perch ricadono sotto una legge, da questo punto di vista, pi permissiva. Ci ha una ricaduta pratica sulle strategie di ricerca che le aziende mettono a punto per sviluppare un microrganismo utile. Il produttore che si trovi di fronte la possibilit di migliorare un ceppo antagonista, modificandolo geneticamente, deve quindi sottostare ad una prima legge per limpiego in ambiente confinato e quindi ad una seconda che si occupa della sua immissione sul mercato. Le procedure tecniche diventano onerose e il prodotto perde in economicit e di competitivit. Esistono tuttavia delle scappatoie legislative per migliorare lattivit antagonistica senza ricadere
19
nella normativa sul rilascio in ambiente confinato di MOGM (Decr.leg. n.91 del 3/3/93). Si possono creare varianti genetiche tramite coniugazione, trasduzione, trasformazione o qualsiasi altro processo naturale; e permessa induzione della poliploidia, la mutagenesi per creare mutanti con caratteristiche nuove e la fusione cellulare (compresa la fusione dei protoplasti) di cellule di piante che possono essere ottenute mediante metodi tradizionali di riproduzione. 5.6. - Etichettature e controlli Il problema delletichettatura ha assunto oggi una rilevanza cruciale. Il decreto legislativo 194/95 regolamenta tale materia per i prodotti fitosanitari. Esso prevede che siano apposte delle etichette, in lingua italiana, su tutti gli imballaggi e contenitori. In generale letichetta deve portare notazioni riguardanti il prodotto e il produttore. In modo specifico le etichette devono contenere la denominazione commerciale del prodotto fitosanitario, con il nome e l'indirizzo del titolare dell'autorizzazione, il numero e la data di registrazione, nonche' i dati anagrafici del responsabile dell'imballaggio dell'etichettatura e della distribuzione, qualora sia diverso dal titolare dell'autorizzazione, nonche' l'indicazione del nome e della sede dello stabilimento di produzione. Letichetta deve inoltre contenere notazioni molto precise riguardanti il nome e la concentrazione di ciascuna sostanza attiva presente nel prodotto fitosanitario, (come da decreto del Presidente della Repubblica 24 maggio 1988, n. 223); i nomi delle sostanze attive devono essere indicate secondo la nomenclatura di cui alla legge 29 maggio 1974, n. 256, (e successive modifiche), oppure, se la sostanza non vi figura, essa pu essere indicata con il suo nome comune ISO o, in assenza, con la sua denominazione chimica IUPAC. Letichetta deve essere corredata anche dai dati sul tipo di azione del prodotto fitosanitario, il tipo di preparazione, la quantita' netta del prodotto fitosanitario, espressa in unita' di misure legali, il numero di partita del preparato o una indicazione che ne permetta l'identificazione. Con Decreto del Ministro della Sanit vanno indicate le frasi di rischio (R) riferite all'uomo, gli animali o l'ambiente (allegato IV) e le complementari frasi di
20
sicurezza (S) per la tutela dell'uomo, degli animali o dell'ambiente (allegato V) e le indicazioni riguardanti i primi soccorsi da prestare. Il comune utilizzatore del prodotto deve poter comprendere dalletichetta le possibilit di uso del fitofarmaco. A tale scopo devono essere indicati gli usi autorizzati le condizioni agricole, fitosanitarie ed ambientali specifiche nelle quali il prodotto fitosanitario puo' essere utilizzato o, al contrario, deve essere escluso (art.16, comma 1, lettera k). Sono fondamentali le istruzioni per l'uso e la dose, espressa in unita' metriche, per ogni diverso impiego autorizzato. I prodotti fitosanitari, seppure autorizzati, possono presentare effetti fitotossici o effetti tossici verso luomo; per tal motivo va indicato in etichetta, nel caso sia necessario, l'intervallo di sicurezza, da rispettare, tra l'applicazione e la semina, tra l'impianto della coltura da proteggere o di quelle successive, tra l'applicazione e l'accesso dell'uomo o degli animali, tra l'applicazione ed il raccolto e tra l'applicazione e l'uso o il consumo dei vegetali trattati. Letichetta appare molto simile a quella di un farmaco ad uso umano ed in fatti contiene la dicitura "Prima dell'uso leggere le istruzioni sul foglio illustrativo" qualora sia allegato un foglio illustrativo, la data di scadenza in normali condizioni di conservazione del preparato, se la durata dello stesso e' inferiore a due anni. Non si possono apporre sulletichetta notazioni quali innocuo o non tossico, in riferimento, ad esempio, alleffetto verso insetti pronubi. Nel caso di trattamento previsto in concomitanza di periodi critici per la presenza di api e permesso indicare sulletichetta frasi del tipo utilizzabile in presenza di specie non bersaglio". I titolari hanno il compito di adeguare gli imballaggi, le etichette fatto salvo per le scorte di prodotti etichettati ed imballati prima dellentrata in vigore di tale decreto. I controlli effettuati dal legislatore prevedono misure a breve e media scadenza. Il controllo ufficiale dei prodotti fitosanitari viene attuato con ladozione di piani nazionali annuali per verificare la rispondenza ai requisiti per cui i diversi prodotti sono stati messi in commercio e la loro utilizzazione conforme alle indicazioni annotate sulletichetta nel rispettto delle dei principi di buone
21
pratiche fitosanitarie e di lotta integrata. E compito delle regioni e delle provincie autonome trasmettere, annualmente, i risultati ispettivi al Ministero della sanit che deve presentare successivamente agli Stati membri e alla Commissione europea una relazione in materia. Il Ministero della sanit deve approntare, in accordo con il Ministero dellambiente e sentito il parere della Conferenza permanente Stato-Regioni dei piani nazionali triennali (art.17, comma 4) In analogia con la Farmacopea Ufficiale dei farmaci destinati alluso stata creata la Fitofarmacopea ufficiale. Il Ministero della sanit tramite il Servizio Informativo Sanitario ne cura la pubblicazione. Essa comprende le monografie delle sostanze attive autorizzati, il prontuario dei prodotti fitosanitari autorizzati e i metodi di analisi per la verifica dei requisiti sanciti dallarticolo 4. Il decreto stabilisce di adottare, riferendosi anche alla legge n.283 del 30/4/1962, i limiti massimi residuali definiti in sede comunitaria e qualora essi non esistano di stabilirli, in via provvisoria con il parere della Commissione consultiva. 5.7. - Considerazioni sulle differenti legislazioni E evidente la grande differenza normativa che esiste tra Unione Europea e USA in materia di registrazione di prodotti fitosanitari. In Europa lobbligatoriet di molte analisi anche quando il microrganismo sia palesemente innocuo, fa si che le richieste di autorizzazione di prodotti fitosanitari presentate siano inferiori di numero a quelle presentate negli USA. La laboriosit delle prove necessarie nonch la difficolt economica della registrazione, che in se non avrebbe tempi di accettazione eccessivamente lunghi ha come effetto non solo la depressione del mercato ma la comparsa di fenomeni di frode. E nota agli operatori lesistenza in commercio di prodotti non registrati come prodotti fitosanitari, bens come ammendanti, aventi come denominazione: "Additivi per terreni e substrati di radicazione a base di sostanze naturali e attivanti biologici E quasi inevitabile che accada ci, vista la grande massa critica, talora non essenziale, che un produttore deve presentare per la registrazione. Sarebbe
22
quindi auspicabile una riforma della legge che presti pi attenzione al singolo prodotto, intendendo ogni prodotto fitosanitario come unico cos da esigere un insieme di prove ed analisi adeguate e non standardizzate come numero. Lo stesso si potrebbe dire per gli OGM e gli MOGM, campo in cui il divario con gli USA ancora pi ampio. 6. - PRODUZIONE e COMMERCIALIZZAZIONE Dopo la fase di isolamento di un microrganismo e lo studio delle sue caratteristiche utili come agente di contenimento biologico, la tappa fondamentale per il successo di un prodotto biologico la cosiddetta fase industriale. Essa studia la formulazione del prodotto e la sua produzione su vasta scala. Spesso si ritrovano in fase di ricerca microrganismi utili e promettenti, ma il passaggio alla produzione e alla commercializzazione non sempre facile e comunque di esito non scontato. La carenza sul mercato di prodotti microbiologici frequentemente da ascrivere ai costi di produzione elevati e dai margini di profitto inadeguati, se riferiti allinvestimento iniziale. Per capire le ragioni di tale fenomeno occorre analizzare gli elementi caratterizzanti la produzione di biomasse microbiche. 6.1. - Produzione su vasta scala In una biofabbrica i costi che pi incidono sono lacquisto dei substrati e lenergia elettrica richiesta per far funzionare gli impianti; a questi due fattori primari si collegano lefficienza del processo fermentativo, collegato alle diverse esigenze di sviluppo dei microganismi e dal punto di vista del marketing, la ridotta economia di scala che tali prodotti per la difesa riescono a raggiungere sul mercato. Lapprovvigionamento di substrati economici per le fermentazioni un fattore cruciale. Le melasse di scarto, derivate dalla lavorazione della barbabietola da zucchero e della canna da zucchero, gli scarti derivati dalla trasformazione industriale del mais rappresentano un materiale di partenza assai economico
23
ma qualitativamente eterogeneo nel tempo. In questo ambito si deve ricercare il compromesso tra economicit dei fattori produttivi e qualit costante del prodotto finale. La produzione in grandi fermentatori , di solito, il mezzo pi comune per la produzione di biomasse ingenti. Si utilizzano due tipi di colture: la coltura su mezzo liquido e quella su mezzo solido. Sono molte di pi le conoscenze sulla fermentazione in liquido che non in solido, dal momento che essa stata il mezzo di produzione delezione di attinomiceti produttori di antibiotici. Tuttavia occorre considerare ogni microrganismo come a se' stante e con caratteristiche di sviluppo e ed esigenze nutrizionali differenti. Non sembra corretto privilegiare un tipo di coltura rispetto ad un altro in modo aprioristico. La coltura in liquido necessita alla del fine processo due fasi fondamentali: la separazione dellinoculo dal mezzo di coltura mediante filtrazione e la fase di concentrazione dellinoculo tramite processi di centrifugazione e di disidratazione. Questultima pu alterare la capacita funzionale del microrganismo, specie se condotta rapidamente tramite riscaldamento. Al contrario una coltura solida non richiede separazione dellinoculo n la disidratazione del prodotto; il micoparassita Sporidesmium sclerotivorum viene prodotto, di routine, con una fermentazione solida (Ayers e Adams, 1983) e anche per Trichoderma spp. stata messa a punto una strategia per la coltura solida su vasta scala (Roussos et al., 1991) Per produrre una grande quantit di biomassa si devono ricercare i valori adeguati non solo per il microrganismo di interesse ma per lo stadio vitale desiderato (conidio, ascospora, clamidospora, spora). La temperatura, lossigenazione, il pH, lumidit, la fonte di carbonio nel substrato, i micromeso- e macro-elementi del substrato devono essere considerati per raggiungere due obiettivi tra loro contrastanti: massimizzare le rese e contenere i costi. La temperatura influenza direttamente lo sviluppo dei microrganismi, influenzando la cinetica delle reazioni enzimatiche. Avere a disposizione un ceppo microbico che cresca a temperature relativamente basse rappresenta un vantaggio economico poich fa risparmiare energia. Il pH e la fonte di carbonio influenzano la qualit dei metaboliti secondari prodotti nel mezzo: la produzione
24
di fenazina in P. fluorescens altamente influenzata dalla composizione del mezzo (Slininger e Shea-Wilbur, 1995). La necessit di ossigenare il mezzo di coltura richiede energia per aumentare la velocit delle pale del fermentatore. Talvolta la scarsa aerazione pu' incrementare la formazione di clamidospore in ceppi micoerbicidi di F. oxysporum (Hebbar et al., 1997). Variare le condizioni di coltura per promuovere la formazione di strutture di resistenza porta ad indubbi vantaggi di tipo commerciale. Un prodotto microbiologico, infatti, richiede, oltre alle caratteristiche finali di elevato titolo, una buona efficacia ed unelevata stabilit commerciale (shelf life). La vita commerciale del prodotto tanto pi elevata quanto pi si commercializzano le forme di resistenza del microrganismo. Ceppi di Fusarium saprofiti vengono prodotti in forma di clamidospore (Hebbar et al., 1997); le spore di Bacillus subtilis rappresentano la forma di resistenza pi efficace per la conservazione del prodotto. Talaromyces flavus presenta ascospore molto resistenti preferibili ai conidi per la commercializzazione (Fravel et al., 1985); alcuni batteri, come Agrobacterium radiobacter K84, sono facilmente disidratabili (Kerr, 1980), altri, tra i batteri gram negativi, sono pi difficili da conservare, mancando di forme fisiologiche di resistenza. 6.2. - Formulazione La formulazione di un prodotto fitosanitario lequivalente della forma farmaceutica dei farmaci destinati alluso umano che rende la sostanza attiva, utilizzabile per le applicazioni. Spesso una corretta formulazione il fattore discriminante tra il successo e linsuccesso di un prodotto. E pressoch inutile avere a disposizione un microrganismo utile ed efficace se non si sono compiuti studi adeguati sulla formulazione da adottare: il microrganismo rimane uno strumento utile in potenza. Gli obiettivi della formulazione di prodotti fitosanitari contenenti microganismi riguardano il mantenimento dellefficacia, la durata commerciale e la facilit duso, distribuzione completa sul sito da raggiungere, la compatibilit con le normali pratiche agricole e i bassi costi di produzione. Un fitofarmaco tradizionale rispetto ad un fitofarmaco microbiologico presenta
25
problemi peculiari che riguardano la stabilizzazione e la durata del prodotto; spesso i produttori di formulati tendono a trascurare queste componenti rispetto ai problemi, di pi facile soluzione tecnica, di distribuzione, di assorbimento e di sicurezza per loperatore. Queste non devono mancare, ma dal momento che il prodotto fitosanitario contiene microrganismi che devono svolgere una funzione in campo diventa primario ed essenziale che essi arrivino vivi al sito di applicazione. Unadeguata formulazione deve rendere stabile la sostanza attiva, sia durante la conservazione, sia nellesposizione agli agenti abiotici, una volta applicata alla coltura: anche in questo caso esistono formulati specifici e pi idonei al differente utilizzo finale di un prodotto. Sono molto usate paste a base di talco, pirofillite, kaolinite utilizzati come veicoli della sostanza attiva; altre volte sono usati substrati veicolanti quali torba, vermiculite, lignite (Kloepper e Schroth, 1981; Vidhyasekaran et al., 1997). Gli alginati si prestano molto alla veicolazione del microrganismo nellambiente ma dal momento che occorre studiare, prodotto per prodotto, ladatta formulazione hanno un costo finale eccessivo. Il Pesta (Connick et al., 1991) un formulato granulare che costituito utilizzando la tecnologia della pasta fatta in casa. Si prepara un in impasto con la farina di semola e la kaolinite cui viene aggiunta la sospensione conidica di interesse; si lavora limpasto fino a creare una palla umida di pasta, si tira in sfoglia e si producono degli spaghetti. Gli spaghetti seccati vengono poi frammentati in granuli di 3-4 mm di lunghezza e 1-2 mm di diametro. Tale formulato viene usato per prodotti micoerbicidi, per G. virens e per Trichoderma spp. pur essendo molto efficace molto costoso applicato a produzioni su vasta scala, per lelevato costo della kaolinite. Un ulteriore miglioramento verr apportato sostituendo la kaolinite con substrati pi a buon mercato come la bentonite. Gli amidi pregelatinizzati vengono utilizzati per formulare G. virens e T. hamatum (Lewis et al., 1995); in tal caso lamido viene gelatinizzato aggiungendo 10 ml ogni 100 g di farina di mais creando delle maglie in cui vengono intrappolati i microrganismi. Tale sistema garantisce una buona sopravvivenza allinoculo, facile da produrre ed economico. I biopolimeri sfruttano il medesimo principio degli amidi gelatinizzati per incapsulare il
26
microrganismo (Bok et al., 1996). Alcuni formulati sono costituiti da emulsioni invertite in cui il microrganismo nella fa se dispersa, circondata dalla fase continua oleosa: tale forma garantisce una buona protezione dagli UV. Si pu influire sullefficacia di un prodotto microbiologico grazie a additivi che alterino lo stato nutrizionale della sostanza attiva. Lazione di Trichoderma e Gliocladium spp. migliore se si aggiunge crusca di grano al formulato (Elad et al., 1980; Elad e Hadar, 1981; Lewis e Papavizas, 1985). Sono noti casi, come per T. flavus, in cui una formulazione povera garantisce risultati migliori (Fravel et al., 1995) ed altri in cui una formulazione con sostanze nutritive specifiche come additivi avvantaggiano lantagonista sul patogeno (Chun et al., 1997). La durata di un prodotto essenziale per garantire la qualit del prodotto nel tempo. E importante ridurre il tenore di ossigeno nel preparato aggiungendo delle sostanze adsorbenti lossigeno, o mediante formulati sotto vuoto. E utile ridurre il tenore di acqua (Connick et al., 1996) cos come si possono aggiungere agenti osmoprotettori e UV protettori (Shapiro, 1992; Shapiro e Robertson, 1992). Labbassamento della temperatura non sembra essere la giusta via da perseguire, dal momento che occorrerebbe creare sistemi quali catene del freddo nei canali distributivi e ci porterebbe, con tutta probabilit, a problemi logistici specie in un sistema a basso profilo tecnologico come quello agricolo italiano, rappresentato da una moltitudine di piccole aziende.
7. - GENERALITA SU Fusarium oxysporum 7.1. Tassonomia Il genere Fusarium rappresenta un gruppo ubiquitario ed eterogeneo di patogeni responsabili di numerose malattie di piante, agenti di marciumi in numerosi ospiti e di micosi in uomini ed animali (Liddell, 1991). Il genere Fusarium conosciuto in tutto il mondo da fitopatologi e micologi come uno dei generi pi difficili da distinguere in specie (Snyder e Hansen, 1939) per la elevata instabilit dei caratteri morfologici e fisiologici influenzati dallambiente e
27
della scarsa corrispondenza tra caratteristiche morfologiche e patogenetiche (Goidanich, 1964). La prima pubblicazione riguardante Fusarium risale al 1809 (Link) e da allora le classificazione sono state molte. La prima classificazione prevedeva la divisione delle circa mille specie di Fusarium allora conosciute, in 16 sezioni (Wollenweber e Reinking, 1935). I criteri di classificazione usati per suddividere le sezioni riguardano la presenza o assenza dei microconidi e la loro forma, la presenza o assenza di clamidospore, la loro disposizione sull'ifa (intercalare o terminale), la forma dei macroconidi e la forma della cellula basale dei macroconidi. Inoltre in ogni sezione si distinguono: specie, variet e forme sulla base del colore dello stroma, dell'assenza o presenza di sclerozi, del numero di setti nei macroconidi, e delle dimensioni dei macroconidi. Il sistema di Wollenweber e Reinking (1935) risulta oltremodo complesso se si pensa che vengono considerati anche le differenti modalit di crescita su differenti substrati (pura influenza ambientale) come fattore per evidenziare differenze significative. Sono stati successivamente sviluppati dei sistemi tassonomici alternativi quali quello di Gerlach e Nirenberg (1982, 78 specie), di Raillo (1950, 55 specie), di Bilai (1955, 26 specie) e di Joffe (1974, 33 specie) di Snyder e Hansen (1940, 9 specie), Messiaen e Cassini (1968, 9 specie), Matuo (1972, 10 specie) di Gordon (1952, 26 specie), di Booth (1971, 44 specie) e di Nelson et al. (1983, 30 specie). Attualmente si fa riferimento al sistema tassonomico proposto da Snyder e Hansen ed illustrato da Toussoun e Nelson (1976) basato sulla suddivisione in una decina di specie. In ogni specie la cultivar rappresenta lentit morfologicamente distinta e in ogni specie vengono raggruppate insieme in forme speciali gli individui che hanno una capacit comune di aggredire il medesimo ospite. Nellambito delle forme speciali si possono ancora distinguere le razze fisiologiche o patotipi distinguibili per la selettivit cultivarietale. I Fusaria, di cui sia nota la forma perfetta, appartengono alla sottodivisione Ascomycotina e allordine Hypocreales. Possiedono dei periteci a parete soffice, flessibile o membranosa, e di solito brillantemente colorata. I periteci
28
hanno inoltre un ostiolo ben definito (Pyrenomycetes). Sono assenti vere parafisi libere, ma parafisi apicali che crescono verso il basso dal centro sono state osservate in varie specie nei primi stadi di sviluppo; gli aschi sono unitunicati, tipicamente clavati e monoamiloidi; le ascospore sono di solito mono- o trisettate. Le specie hanno spesso due stati conidici, conosciuti come microconidi e macroconidi; le loro modalit di formazione sono prevalentemente enteroblastiche (o fialidiche), ma i macroconidi possono essere generati anche in modo oloblastico. Una terza forma conidica rappresentata dalla clamidospora: una spora tallica dalla parete poco ispessita, presente in molte specie e avente funzione di resistenza nellambiente. Nella classificazione di Wollenweber e Reinking (1935) una delle 16 divisioni la Elegans in cui si ritrova la specie chiamata F. oxysporum Schlect: Fr. (Snyder e Hansen, 1939). Il F. oxysporum presente in natura come saprofita e come agente di fitopatie (tracheomicosi). Tra i F. oxysporum sono state descritte pi di 120 formae speciales, ognuna delle quali specifica per una specie vegetale. Nellambito delle diverse forme speciali si distinguono numerose razze fisiologiche o patotipi (Armstrong e Armstrong, 1981). La divisione in forme speciali e patotipi (Snyder e Hansen, 1940) ha un origine fisiologica basata su criteri patogenetici e non evidenzia in modo corretto i rapporti genetici tra isolati patogeni diversi. Sono entrambe valide le teorie secondo cui un patogeno si evolva da una precedente forma speciale patogena o da un insieme di funghi ad attivit saprofitica. In natura si possono trovare isolati che possiedono la medesima specificit dospite ma distanti geneticamente; il caso di un Fusarium patogeno del banano che presenta molte similitudini con F. oxysporum f. sp. lycopersici: il fenotipo virulento, che viene acquisito in maniera indipendente dai due isolati evidenzia unorigine polifiletica. Diversamente sono noti casi in cui i Fusaria patogeni delle cucurbitacee sembrano derivare tutti da un unico progenitore (Gordon e Martyn, 1997). Per studiare lorigine monofiletica o polifiletica di una forma specializzata o di una nuova razza si devono valutare i diversi casi con metodiche differenti.
29
La comparsa di una nuova infezione in una nuova area, o di un nuovo patotipo (razza fisiologica), pu essere spiegata con il fatto che il patogeno sia stato introdotto o che sia generato localmente. L'introduzione, non deliberata, di un patogeno in una nuova area disciplinata da regole di quarantena severe che in un momento di libero scambio come quello odierno spesso vengono superate. Spesso, l'ampia distribuzione di un isolato patogeno da ricondurre alla dispersione clonale di un singolo genotipo, accidentalmente introdotto; tale ipotesi facilmente verificabile tramite l'analisi comparativa dei VCGs: se il nuovo patogeno stato introdotto accidentalmente o deriva da uno preesistente in quellarea allora, verosimilmente, non sar compatibile con le specie indigene. La seconda ipotesi, invece, trova la sua spiegazione nella derivazione da una forma preesistente o nella selezione di un individuo proveniente dalle popolazione di isolati non patogeni di F. oxysporum. Le analisi filogenetiche condotte con tecniche di ibridazione dimostrano, a questo riguardo, che le varie razze di F. oxysporum f. sp. conglutinans possono essere ricondotte ad un comune antenato (Kistler et al., 1991). Le razze fisiologiche 1, 2 e 3 di F. oxysporum f. sp. lycopersici, tutte appartenenti al VCG 0030 (Elias e Schneider, 1991), sottoposte ad analisi con RFLP sul DNA nucleare permettono di confermare l'associazione al VCG 0030 e di ipotizzare una derivazione delle diverse razze, luna dallaltra. Per F. oxysporum f. sp. dianthi lapplicazione delle diverse tecniche di caratterizzazione molecolare (VCG, RFLP, cariotipi elettroforetici, distribuzione degli elementi trasponibili impala e Fot1, PCR) ha permesso di ipotizzare l'evoluzione della razza 8 a partire dalle razza 1 per la perdita di un fattore di avirulenza (Chiocchetti et al., 1999). 7.2. - Malattie causate da Fusarium oxysporum Sotto il nome di tracheomicosi si annoverano malattie caratterizzate dalla prevalente localizzazione del patogeno di natura fungina nel sistema vascolare legnoso delle piante. Molte forme speciali di F. oxysporum sono i pi comuni e diffusi agenti di tracheomicosi delle piante (tracheofusariosi). Le forme speciali
30
di F. oxysporum diffondono i loro propaguli per via aerea e penetrano nell'ospite di solito attraverso il sistema radicale. F. oxysporum non presenta la forma perfetta, ma produce tre tipi di spore asessuate: macroconidi, microconidi e clamidospore. Queste ultime sono la forma di resistenza del patogeno che, in presenza degli essudati radicali delle piante ospiti e delle condizioni ottimali, rompono la quiescenza e germinano. I sintomi tipici delle malattie vascolari includono: nanismo, appassimento, clorosi, scolorimento vascolare, necrosi e morte della pianta. I diversi sintomi possono comparire contemporaneamente, in successione o prevalere gli uni sugli altri in funzione dell'ospite, del patotipo del parassita e delle condizioni ambientali. La caratteristica pi evidente e costante l'imbrunimento dell'apparato vascolare legnoso, che deve essere assunto come indizio diagnostico di primo ordine. Possono essere interessate da tracheofusariosi piante in qualsiasi stadio di sviluppo, ma sono le infezioni contratte precocemente quelle che causano danni economici pi rilevanti. Il patogeno penetra nell'ospite attraverso le micro-ferite dellapparato radicale, provocate da nematodi o nelle operazioni di trapianto, i meristemi apicali e la zona di allungamento delle giovani radici, dove la suberificazione dell'epidermide ancora incompleta, e la sua diffusione di tipo intercellulare (Brandes, 1919; Smith e Walker, 1930; Ullstrup, 1937). Durante la colonizzazione dei tessuti il patogeno si sposta verso i vasi legnosi e una volta raggiunto l'interno del sistema vascolare, il patogeno rimane confinato negli elementi dei vasi xilematici, e si diffonde verticalmente nella pianta in queste cellule (Brandes, 1919; Pennypacker e Nelson, 1972; Tisdale, 1917; Ullstrupp, 1945). In un secondo momento, il fungo penetra nelle cellule del parenchima xilematico circostante, attraversando le punteggiature areolate sulla parete dei vasi stessi (Pennypacker e Nelson, 1972; Phipps e Stipes, 1976). La colonizzazione del sistema vascolare dell'ospite da parte del fungo rapida e si osserva la produzione di conidi liberi di muoversi nella corrente xilematica, fino a che non incontrano i setti trasversi punteggiati, posti fra due elementi dei vasi. Non potendo superare lostacolo a causa delle loro dimensioni, i conidi germinano, superando il setto con i sottili tubuli germinativi. Oltre lelemento xilematico il
31
fungo produce conidiofori e conidi che vengono trasportati al setto successivo dal flusso xilematico. Il processo di colonizzazione dellospite verso lalto procede a salti. I Fusaria agenti di tracheomicosi, nel processo di colonizzazione dell'ospite producono enzimi degradativi (Deese e Stahmann, 1962 e 1962, Gothoskar et al., 1953 e 1952). Gli enzimi prodotti da questi patogeni sono in prevalenza di tipo pectolitico con effetti di macerazione cellulare e di ostruzione dei vasi. Le piante ospiti generalmente hanno approntato sistemi di difesa nei riguardi della maggior parte delle forme speciali di F. oxysporum patogene. Sono in grado di produrre gel, gomme e tille allinterno degli elementi dei vasi xilematici (Pennypacker, 1981) e nelle cellule dell'epidermide, ipodermide e cortex delle radici, la presenza del patogeno stimola lispessimento della parete in corrispondenza dei punti di penetrazione, che possono evolvere in papille, caratteristiche formazioni coniche, con base poggiante sull'interno della parete cellulare, che crescono tra parete e plasmalemma in seguito a deposito di calloso (-1,3-glucano) e di lignina attorno all'ifa del patogeno. Il deposito di gel e gomme di natura pectica ed emicellulosica, che accompagna la colonizzazione dello xilema, inizia dapprima a livello dei setti trasversi e procede, quindi, sulle pareti laterali dei vasi. La produzione di tille nei vasi legnosi da parte delle cellule parenchimatiche annesse ai vasi stessi, un ulteriore ostacolo alla diffusione del parassita vascolare. La loro formazione sembra essere ospite-dipendente, ed state rilevata solamente durante l'infezione del banano (Beckman, 1969; Beckman e Zaroogian, 1967), melone e patata dolce (Matta et al., 1996). Nel caso del banano la formazione delle sostanze gelatinose incomincia dalla superficie superiore dei setti trasversi ed causata dall'ingrossamento degli stessi setti e della porzione terminale della parete dei vasi. Le gomme, le tille e le pareti dei vasi possono essere impregnate sia da composti antibiotici, come i tannini, sia da altri composti fenolici rapidamente ossidati in chinoni: questi ultimi, rilasciati dalle cellule associate ai vasi, rendono i vasi impermeabili all'acqua e, frapponedosi negli spazi intercellulari,
32
impediscono gli scambi di tossine e nutrienti tra il patogeno ed i tessuti circostanti (Matta et al., 1996). La formazione di gomme pu dipendere da gelificazione dei prodotti di degradazione della parete di vasi, operata dagli stessi enzimi pectolitici del patogeno, o da enzimi della stessa pianta, attivati in risposta all'infezione. Si ritiene che possano giocare un ruolo importante nel contenimento del patogeno le cellule annesse ai vasi xilematici con ipertrofie, che si traducono in tilosi, con iperplasie, in grado di delimitare e sostituire vasi o interi gruppi di vasi infetti e tramite fenomeni degenerativi irreversibili come imbrunimenti e gommificazioni (Matta et al., 1996). Nella comparsa della malattia fondamentale la velocit relativa con cui avvengono i fenomeni di colonizzazione del patogeno e di difesa della pianta. Se le difese sono attivate rapidamente e in modo persistente i conidi si ritrovano in sacche d'invasione: gel e gomme rilevate nel garofano (Pennypacker e Nelson, 1972), nella mimosa (Phipps e Stipes, 1976), nella patata dolce (McClure, 1950), nel cotone (Bugbee, 1970) e nell'anguria (Nishimura, 1971), giocano un importante ruolo nel limitare la diffusione del patogeno, intrappolando i conidi fra gli elementi dei vasi. Tuttavia se il gel persiste solamente per un limitato lasso di tempo, se le tille ritardano a formarsi o se non si formano affatto, i conidi sono liberi di diffondere oltre l'occlusione vascolare, e l'infezione si traduce in blocchi via via pi estesi del trasporto xilematico (Beckman, 1966). In virt delle molteplici anastomosi tra vasi, che consentono il facile aggiramento delle occlusioni, la disfunzione di una parte anche rilevante dell'apparato vascolare del fusto non necessariamente seguita da sintomi esterni evidenti di appassimento e di avvizzimento. Essi diventano irrimediabili ed evidenti quando il patogeno colonizza il sistema xilematico dei piccioli e delle nervature fogliari, non ridondante e privo di anastomosi laterali (Matta et al., 1996).
33
7.3. - Tecniche di lotta contro le fusariosi Le tracheomicosi ad opera di F. oxysporum arrecano danni gravi, legati allavvizzimento della porzione epigea della pianta, in numerosissime colture, tra cui basilico, garofano e ciclamino. Le principali strategie di lotta alle tracheofusariosi prevedono luso di fungicidi sistemici quali i benzimidazoli (Garibaldi et al., 1986). Tale approccio porta a due tipologie di problemi che ne limitano lefficacia: i residui, dannosi per lambiente, e linsorgenza di ceppi resistenti, come peraltro gi evidenziato per Botrytis cinerea su di versi ospiti (Gullino e Garibaldi, 1987), a causa della specificit del meccanismo dazione di questi fungicidi. La fumigazione con il bromuro di metile il mezzo pi utilizzato per leradicazione dei patogeni agenti di tracheomicosi e malattie telluriche fungine. Il Protocollo di Montreal e le sue successive modificazioni recepite dallUE, in materia di limitazioni di impiego, hanno sancito lanno 2005 come data ultima, nei paesi industrializzati, per attuare leliminazione di tale pratica, dannosa per lo strato di ozono atmosferico (Garibaldi e Gullino, 1995; Ristaino e Thomas, 1997). La solarizzazione o pacciamatura riscaldante del terreno stata messa a punto nel 1976 in Israele (Katan, 1987). Essa si basa sul riscaldamento del terreno nei periodi pi caldi, mediante copertura con un film plastico trasparente, cos da raggiungere temperature letali per i parassiti. Luso della resistenza genetica (verticale e orizzontale) rappresenta lo strumento pi sicuro ed economico. Esso si basa sullimpiego di cultivar resistenti in cui il patogeno non si insedia o non riesce a completare il ciclo di patogenicit (Johnson, 1992). I mezzi biologici rappresentano un ausilio, in una logica di lotta integrata, per il contenimento delle malattie. Gli agenti di tracheofusariosi possono essere limitati sfruttando il fenomeno naturale dei terreni repressivi. Tali fenomeni di repressivit, verso patogeni diversi, sono naturalmente presenti in terreni italiani (Garibaldi et al., 1980) e in terreni francesi (Alabouvette et al., 1980). Lattivit antagonistica di isolati antagonisti stata dapprima riprodotta in vitro, con lisolamento degli agenti antagonisti (Callegarin e Cugudda, 1983) e poi saggiata in vaso dando risultati incoraggianti (Garibaldi et al., 1985). E ormai
34
noto che trattamenti come la concia dei semi con Fusarium spp. antagonisti o lapplicazione, al trapianto, a livello dellapparato radicale di preparati a base di clamidospore di Fusarium spp. serva per contenere, entro le soglie di danno, le fusariosi vascolari (Minuto et al., 1997). Studi sul contenimento delle formae speciales dianthi (Garibaldi et al., 1985), cyclaminis (Minuto et al.,1995) e basilici (Minuto et al., 1997) di F. oxysporum sono stati compiuti dando risultati soddisfacenti, se pure non paragonabili a quelli ottenuti con la lotta chimica. Le strategie future sono comunque proiettate verso lapplicazione e della lotta biologica integrandola con le altre risorse di difesa delle colture.
8. - CARATTERIZZAZIONE DI Fusarium spp. ANTAGONISti Lidentificazione univoca di un microrganismo al fine di differenziarlo da altri una condizione essenziale per la registrazione, per la brevettazione e per poterne seguire il destino una volta rilasciato nellambiente. Le tecniche utilizzabili sono molte e, per essere efficaci ed efficienti, devono rispondere ai requisiti di: economicit, rapidit di esecuzione e la facilit di esecuzione. Tra i metodi utilizzati in diagnostica fitopatologica e in particolare per lanalisi dei Fusarium spp. saprofiti sono state sviluppate: lanalisi dei gruppi di compatibilit vegetativa, lanalisi di proteine, lanalisi isoenzimatica, la sierodiagnosi e lanalisi degli acidi nucleici. I Fusarium spp. possono essere suddivisibili in gruppi di compatibilit vegetativa (VCG). La compatibilit vegetativa la capacit dei funghi di formare eterocarion, in seguito ad anastomosi ifale. La base genetica normalmente monogenica poliallelica o regolata da pi loci (Leslie, 1993). Il fenomeno della compatibilit si verifica, tra due funghi se possiedono i medesimi alleli vic (vegetative incompatibility). Per identificare i diversi VCG possibile usare la diretta identificazione della formazione dell'eterocarion tramite la complementazione fra marcatori auxotrofi, ovvero si pu verificare lincapacit formare l'eterocarion, grazie alla formazione di linee di "barrage" o verificando modalit di crescita caratteristiche di isolati
35
parzialmente diploidi ed eterozigoti per uno o pi loci vic. Nel primo caso isolati auxotrofi, compatibili, formano un eterocarion prototrofico. Se gli isolati appartengono a VCG differenti, non si avr crescita prototrofica. In genere, si usano mutanti nit spontanei isolati su substrato a base di clorato. Tale tecnica si rivelata particolarmente efficiente per la caratterizzazione del genere Fusarium. Al contrario, si pu utilizzare il barrage come criterio di individuazione di non appartenenza ad uno specifico VCG. La metodica prevede laccrescimento in piastra di funghi appartenenti a VCG diversi e la formazione di una linea di demarcazione tra i due miceli, in cui le ife, nel punto di contatto delle due colonie, crescono una all'interno dell'altra. La comparsa del fenomeno richiede comunque la fusione ifale, e nella zona interessata al fenomeno ci saranno tracce della fusione letale. Il "barrage" consiste quindi in una zona centrale di contatto fra ife contenente cellule morte o morenti; si pu evidenziare una linea di pigmento che si deposita in tale zona (Newhouse e MacDonald, 1991). La determinazione di parentela tra isolati esclusivamente tramite VCG si presta ad errori se non viene affiancata da altre analisi. Spesso si evidenziato che due isolati non sono chiaramente collocabili in un VCG se questi non sono identici. Sono stati fatti studi complementari affiancando i VCG ad analisi biometriche (Butcher et al., 1972) a marker molecolari (Bosland e Williams, 1987; Rosewich et al., 1999). Si quindi evidenziato che isolati appartenenti allo stesso VCG tendono ad essere pi simili fra loro di altri, appartenenti a VCG differenti. Nellambito della specie F. oxysporum sono state distinte 122 formae speciales e per ognuna diverse razze (patotipi) in base alla capacit di infettare diverse specie di piante. Affiancate alle formae speciales, ne esistono molte, non patogenetiche, che non sono in grado di causare fitopatie; si tratta in genere di isolati saprofiti, oppure di formae speciales avirulente, o isolati scarsamente patogeni (Gordon e Okamoto, 1992; Gordon e Martyn 1997). Se per gli isolati patogeni si riscontra una grandissima variabilit per differenza di ospite, lo stesso non si verifica per gli isolati saprofiti difficilmente distinguibili su base morfologica. Per quanto riguarda i VCG i Fusarium spp. saprofiti
36
possono essere considerati entit microbiche ecologicamente distinte in cui la differenziazione con i VCGs sostituisce, per mancanza di specificit dospite il criterio di distinzione basato sulle formae speciales. Isolati non patogeni di F. oxysporum, colonizzanti le radici del sedano, appartengono a soli due VCG principali (Correll et al., 1986). In base allanalisi correlata tra capacit patogenetica e VCG si ipotizzato che: isolati patogeni vegetativamente compatibili possano avere unorigine monofiletica, anche se provenienti da aree geografiche distinte e, di contro, che isolati con capacit patogeniche simili appartenenti a VCG diversi possano avere unorigine polifiletica, cio si siano evoluti indipendentemente luno dallaltro. E' questo il caso, ad esempio, della f. sp. dianthi, per la quale sono stati individuati ben 6 VCG diversi, tutti patogeni su garofano (Baayen et al., 1997). Lapplicazione dei VCG e lanalisi dei relativi loci vic hanno dei risvolti negli studi di popolazione e servono come strumento ausiliario dellanalisi molecolare. Lipotesi di studio che sta alla base della validazione dei VCG come strumento diagnostico riguarda levidenza che isolati dello stesso gruppo di patogenicit, forma speciale, razza appartengano ad uno o pochi VCG. Utilizzati come strumento unico possono non essere completamente esaustivi, ma affiancati a studi di variabilit genetica con marcatori RFLP o RAPD possono concorrere a precisare le correlazioni tra patogenicit e filogenesi (Correll, 1991). 8.1.- Analisi delle proteine Lanalisi delle componenti biochimiche di organismi fungini stata allinizio condotta sulla matrice proteica per evidenziare differenze specifiche e subspecifiche considerando che isolati fungini coltivati in vitro producono, in condizioni standard e in assenza di stress, le medesime proteine, a meno della variabilit indotta a livello somaclonale. Le analisi vengono fatte normalmente sulle proteine totali e sulle diverse frazioni proteiche presenti nei diversi scomparti cellulari e nei diversi ambiti analizzare proteine strutturali o funzionali (Hawksworth, 1988). Lanalisi delle proteine procede per fasi distinte riassumibili in a) concentrazione del campione fungino di partenza, b) lisi del
37
materiale meccanica e mediante detergenti, c) separazione delle diverse componenti proteiche, d) separazione elettroforetica o mediante diversi tipi di cromatografie, e) colorazione e confronto. a) Normalmente bene partire da micelio liofilizzato e sminuzzato per avere una buona quantit di materiale su cui lavorare. b) Si procede alla lisi in presenza di detergenti SDS e mediante laggiunta di inibitori di proteasi come la pepstatina, laprotinina e la leupeptina. c) Si effettua una prima separazione precipitando con soluzioni saline le proteine dalle altre frazioni cellulari (membrane, pareti etc.). d) Si carica su matrici di composizione opportuna a seconda dello scopo il campione proteico e lo si separa in base alla carica, al pH, al peso molecolare. e) Si colora con coloranti come il Blue Coomassie, il nitrato dargento o si evidenziano le proteine con reazioni immuno-colorimetriche. Le tappe cruciali una volta ottenuto lestratto proteico sono quelle di separazione. La cromatografia stata storicamente la prima tecnica di separazione di macromolecole anche se inizialmente veniva utilizzata per separare i pigmenti, di natura saccaridica, estratti dai vegetali. Esistono parecchi tipi di cromatografie ed esse sono divisibili, sulla base dei diversi principi di separazione e sulla base dei diversi supporti. Si pu utilizzare il principio della ripartizione delle molecole in diversi solventi (polari, apolari) tra queste si possono utilizzare supporti quali la carta o matrici composte da gel di silice su strato sottile (TLC). Ma possibile sfruttare i principi della cromatografia a scambio ionico e di affinit. Normalmente le matrici per questi due tipi di separazione vengono impaccate allinterno di colonne di cromatografia. Nella cromatografia su carta la fase stazionaria liquida legata alle fibre di cellulosa di un foglio di carta, la fase mobile passa sul foglio di carta per gravit oppure per capillarit (Lehninger et al., 1979). ll foglio fatto permeare, a partire da un bordo, da una miscela di solventi ed al passaggio del liquido, le molecole del campione proteico si separano secondo la loro solubilit relativa nei solventi. E' una delle forme pi vecchie di cromatografia e attualmente viene
38
utilizzata in biochimica per dimostrare i principi cromatografici pi che per vere applicazioni. Nella cromatografia su strato sottile la fase stazionaria, legata ad una matrice adatta, si trova stratificata su una superficie di vetro, plastica o metallo e la fase mobile liquida passa, per capillarit, sul sottile strato di materiale in posizione orizzontale o verticale. Questo metodo ha il vantaggio di poter analizzare simultaneamente pi campioni. Nella cromatografia su colonna, la fase stazionaria attaccata ad una matrice adatta (un supporto inerte ed insolubile), impaccata all'interno di una colonna di vetro o di metallo, e la fase mobile viene fatta passare nella colonna per gravit, tramite un sistema di pompe, oppure applicando gas sotto pressione. E' il metodo cromatografico pi usato. Le diverse proteine sono in questo caso ritardate in base alle loro interazioni con la matrice. Possono essere usati diversi principi in base alla matrice scelta, le proteine possono essere separate in base alla carica (cromatografia a scambio ionico), alle dimensioni (cromatografia su gel) e alle loro capacit di legarsi a gruppi chimici particolari (cromatografia per affinit; Dean et al., 1985). Il limite delle cromatografia era la risoluzione ma oggi, grazie anche ai nuovi materiali disponibili, possibile ottenere alte risoluzioni usando lHPLC (cromatografia liquida ad alte prestazioni; Gilbert, 1987). Si utilizzano per la ripartizione del campione elevate forze di pressione ottenute da pompe. Le colonne sono di metallo cos da resistere alla forze di pressione usate per risolvere i campioni. Rispetto alla cromatografia convenzionale presenta indubbi vantaggi, oltre che in risoluzione anche nei tempi di analisi, dal momento che la pressione velocizza il processo di separazione. Oltre alla cromatografia le proteine possono essere separate e analizzate mediante lelettroforesi. Questa metodica sfrutta la differente mobilit delle macromolecole allinterno di un campo elettrico. La tecnica elettroforetica pi utilizzata per analizzare le proteine lSDS-PAGE (Hames e Rickwood, 1981). La matrice in cui avviene la separazione un gel di poliacrilammide, derivato dalla polimerizzazione in presenza di catalizzatori di acrilammide e bis-acrilammide. Questo tipo di gel agisce come un setaccio
39
molecolare, rallentando la migrazione delle proteine proporzionalmente alla loro massa molecolare (Lehninger et al., 1979). I campioni proteici sono di norma preparati in un tampone contenente SDS (sodio-dodecil solfato) e mercaptoetanolo; il primo un detergente capace di legarsi alle porzioni idrofobiche delle proteine in maniera proporzionale alla massa, in un rapporto di una molecola di SDS ogni due aminoacidi. Tale legame conferisce una carica netta negativa alla proteina rendendo trascurabile la carica propria della proteina. Il -mercaptoetanolo un agente riducente in grado di scindere tutti i legami S-S, intra e inter-molecolari. LSDS e il -mercaptoetanolo contribuiscono a denaturare le proteine alterandone la conformazione nativa. In definitiva le proteine, tutte cariche allo stesso modo e linearizzate migreranno, poste in un campo elettrico, solo in base alla differenza di peso molecolare e verso lanodo. Il risultato la separazione in bande nette del campione proteico. Le proteine principali sono visualizzate tramite la colorazione con blu di Coomassie, mentre le proteine meno rappresentate vengono evidenziate con il nitrato dargento (silver staining), che permette di visualizzare una banda contenente soltanto 10 ng di proteina. E possibile in tal modo confrontare i profili proteici di isolati fungini diversi e discriminarli sulla base della presenza differenziale di proteine specifiche. Possono emergere profili identificativi di una specie e si possono valutare affinit e differenze a fini tassonomici e diagnostici. Lanalisi fine delle proteine pu essere migliorata purificando mediante immunoprecipitazione con anticorpi (monoclonali o policlonali). Si effettua, quindi, un SDS-PAGE il trasferimento su una membrana di nitrocellulosa (Western blot) e si rileva con sistemi di chemioluminescenza. Tale metodologia usata di routine in ricerca di base quando si vuole studiare una proteina e le sue associazioni con altre allinterno della cellula. Essa presuppone la produzione a monte di un antisiero di tipo policlonale. E una tecnica che richiede qualche giorno di tempo e come tale non utilizzabile per analisi diagnostiche massali.
40
Le prime applicazioni dell'analisi elettroforetica delle proteine miceliari per l'identificazione di funghi risalgono agli anni '60. Clare (1963) analizz gli estratti miceliari di sei specie di Pythium dimostrando che ciascuna di esse aveva un profilo proteico caratteristico. Lo stesso metodo fu usato per distinguere isolati di Phytophthora cinnamomi, P. citrophthora e P. palmivora e si evidenzi isolati diversi della stessa specie mostravano profili identici o simili tra loro, indipendentemente dell'area geografica o dall'ospite di provenienza (Clare e Zentmyer, 1966). La analisi esterasica dei medesimi profili elettroforetici su P. cinnamomi, P. citrophthora e P. palmivora, confermati essere molto simili, dimostr un'elevata variabilit intraspecifica (Hall et al., 1969). Sono stati eseguiti numerosi studi comparativi su organismi dello stesso genere o di generi strettamente correlati sulle proteine ribosomali (Adouvette-Panvier et al., 1980; Delaunay et al., 1973; Gotz e Arnold, 1980), dimostrando che, generalmente le proteine strutturali presentano un alto grado di conservazione (Delaunay et al., 1973). Numerosi estratti ribosomiali di specie di Fusarium sono stati sottoposti a SDSPAGE, evidenziando la presenza di circa 40 bande distinte per ogni estratto proteico ribosomiale (Partridge, 1991). I ribosomi eucarioti, oltre gli acidi ribonucleici, sono costituiti da 70 proteine strutturali, alcune con diversi gradi di fosforilazione. Tali dati hanno permesso di concludere che esistono proteine apparentemente comuni a tutte le specie di Fusarium e che un numero maggiore di bande in comune rilevabile in quelle specie che presentano fasi sessualmente compatibili. La ricerca di proteine specifiche utili ai fini diagnostici ha dato esiti positivi in F. moniliforme. dove stato possibile caratterizzare proteine ribosomiali specifiche (Marshall e Partridge, 1981). Lanalisi di proteine solo sulla base del peso molecolare non sempre permette di evidenziare differenze utili ai fini diagnostici. Lanalisi di proteine funzionali di tipo isoenzimatico ha permesso di superare controversie tassonomiche, si rivelata utile per lidentificazione di patogeni, per analizzare la variabilit genetica di popolazioni o la segregazione di loci genetici, in studi epidemiologici
41
per monitorare la diffusione dei patogeni ed identificarne la condizione nucleare o il livello di ploidia (Burdon e Marshall, 1983; Tooley et al., 1989) Gli isoenzimi sono proteine con identica funzione enzimatica, ma aventi struttura primaria differente perch codificate da alleli diversi di uno stesso locus (allozimi), da loci genetici diversi che controllano la produzione dello stesso enzima, oppure in seguito a modifiche post trasduzionali. Le differenze nella struttura primaria si riflettono sulla struttura secondaria, terziaria e quaternaria e possono essere sfruttate per discriminare sulla base della carica e dimensione i diversi isoenzimi. Il profilo elettroforetico di molti isoenzimi per campione definisce una impronta digitale (fingerprint) e la differenziazione di un organismo da un altro si basa, sul confronto dei fingerprint ottenuti (Micales et al., 1986). Si pu esemplificare considerando che, nellanalisi isoenzimatica, la funzione catalitica, per ogni enzima considerato, sia la costante mentre la struttura sia la variabile. Sulla base di differenze strutturali, direttamente correlata con la sequenza genica, si riesce a collocare un individuo incognito in una specie, di cui siano noti il fingerprint isoenzimatici. L'analisi isoenzimatica si rivelata utile per confermare l'appartenenza di isolati di F. oxysporum f.sp. lycopersici (Elias e Schneider, 1992) e per isolati di F. oxysporum f.sp. conglutinans (Bosland e Williams, 1987) a determinati gruppi di compatibilit vegetativa. In studi tassonomici gli isoenzimi sono stati usati per caratterizzare isolati delle razze 0, 1 e 2 di F. oxysporum f.sp. niveum, isolati di F. oxysporum f.sp. cucumerinum, isolati di F. oxysporum f.sp. vasinfectum e F. solani. Gli enzimi utilizzati sono stati: glucosio-6-fosfato deidrogenasi, shikimato deidrogenasi, esterasi e fosfoglucosio isomerasi, (Biles e Martyn, 1988). Lanalisi dei profili delle esterasi e della -D-glucosilasi stata utilizzata per diagnosticare la presenza di infezioni di Fusarium ff.spp. in piante di ciclamino e garofano, (Kerssies et al., 1994). La aril esterasi stata usata per, distinguere le razze fisiologiche di F. oxysporum f.sp. dianthi (Granada et al., 1999)
42
8.2. Sierodiagnosi La sierodiagnosi una tecnica diagnostica che prevede laccertamento della presenza/assenza in un campione di analisi di un patogeno, in base alla reazione di riconoscimento antigene-anticorpo. Lanticorpo una proteina con peso molecolare di circa 200 kD, chiamata immunoglobulina. Le Ig utilizzate in ricerca e a scopo diagnostico sono le IgG. Esse sono tetrameri omodimerici formati da due catene pesanti tra loro collegate da due ponti disolfuro e due catene leggere legate ognuna ad una catena pesante con un ponte disolfuro. Lantigene (anticorpo-generatore) una molecola che pu avere composizione chimica molto variabile e che in grado, se inoculata in un organismo dotato di sistema immunitario (uomo e animali), di stimolare una reazione di tipo immunitario che sfocia nella produzione massiva da parte dei linfociti B di IgG. Gli anticorpi hanno la caratteristica peculiare di legarsi in modo specifico con alta affinit di legame agli antigeni che li hanno indotti. Questa peculiarit che viene utilizzata per riconoscere determinanti specifici dei patogeni al fine di evidenziarli. La sierodiagnosi presenta diversi vantaggi: il basso costo, la rapidit di esecuzione, la specificit delle reazioni e l'assenza di rischi biologici. Tra gli svantaggi ci sono: la difficolt, in alcuni casi, di produrre antisieri di buona qualit, la sensibilit non sempre paragonabile a quella di altre metodiche e la necessit di decidere ancor prima di effettuare la prova quale patogeno cercare e di dover disporre a priori, di numerosi anticorpi diversi (Matta et al., 1996). Gli anticorpi contro microrganismi vengono prodotti iniettando estratti fungini in sistemi animali (topo, ratto, coniglio, gallina), con richiami ogni settimana per un periodo di un mese, (periodo variabile nei diversi animali). Al termine di tale trattamento si preleva il sangue, si purifica separa il siero mediante coagulazione e si utilizza crudo o si sottopone a purificazione mediante matrice cromatografica. Il siero cos ottenuto detto policlonale perch gli anticorpi in esso contenuti riconoscono molteplici epitopi dellantigene ed epitopi di molecole non di interesse. Questo grado di aspecificit stato superato migliorando la qualit e la specificit dellantigene utilizzato per linoculo; sono
43
stati utilizzati.precipitati proteici o estratti miceliari delle colture fungine (Gleason et al., 1987; Gerik et al., 1987; Barker e Pitt, 1987; Mohan, 1988), sono stati approntati antisieri contro specifiche frazioni del fungo, rappresentate da bande specie specifiche evidenziate tramite SDS-PAGE (Poupard et al., 1991; Sundaram et al., 1991) o da carboidrati solubili (Notermans et al., 1987; Notermans e Kamphuis, 1990; Newton e Reglinski, 1993). La scarsa uniformit e costanza qualitativa tra antisieri policlonali preparati a partire dallo stesso tipo di antigene ma purificato in momenti diversi rende i risultati di una sierodiagnosi non sempre ripetibili. Questo inconveniente pu essere superato con ladozione di sieri monoclonali. Un antisiero linsieme di molecole anticorpali derivate da diversi linfociti B. La tecnica messa a punto da Kohler e Milstein (1975) ha permesso di ottenere ibridomi, cio linee cellulari clonali produttrici di un singolo anticorpo specifico. Tale metodica si avvale della fusione mediata da polietilenglicole di linfocita B con cellule di plasmacitoma di topo mutate per la via di salvataggio dei nucleotidi incapaci di crescere su terreno HAT (ipoxantina, aminopterina, timidina). I linfociti B non si moltiplicano in vitro in maniera indefinita ma sopravvivono nel terreno HAT, le cellule di mieloma si moltiplicano indefinitamente ma non sopravvivono in terreno HAT. Gli ibridomi ottenuti con la fusione sopravvivono su terreno HAT e si moltiplicano a dismisura. Occorre poi selezionare, previa diluizione limite, in presenza dellantigene il clone desiderato utilizzando il sistema ELISA in micropiastra. Gli ibridomi ottenuti sono propagabili come cloni individuali, ciascuno dei quali in grado di produttore in maniera stabile e permanente un singolo tipo di anticorpo per un unico antigene. La specificit degli anticorpi monoclonali li rende molto pi utili degli antisieri convenzionali per la maggior parte degli scopi. Le prove sierologiche, eseguite con anticorpi monoclonali, possono essere usate sia per diagnosi di tipo massale sia per discriminare in modo fine sulla base di differenze proteiche intraspecifiche o tra sottospecie (Dewey e Thornton, 1995; Dewey, 1992). Sono state messe a punto molte tecniche usando il principio del riconoscimento antigene-anticorpo. Tra le pi datate vi sono i saggi di precipitazione o
44
agglutinazione che sono per stati messi a punto in patologia vegetale in un secondo momento rispetto al campo medico. Nella precipitazione gli anticorpi e gli antigeni, si legano e formano aggregati, dando luogo ad un precipitato opalescente, visibile ad occhio nudo. Per l'identificazione di ceppi di Aspergillus e Penicillium, sono state utilizzate sferette di lattice sensibilizzate, adsorbite con anticorpi specifici per polisaccaridi fungini extracellulari (Notermans e Kamphuis, 1990). L'immunomicroscopia e lanalisi immunoenzimatica sono pi recenti della precipitazione e pi utilizzate in diagnostica fitopatologica. Limmunomicroscopia si avvale delluso di anticorpi per localizzare un patogeno nei tessuti di una pianta colpita. Si pu applicare alla microscopia ottica e a quella elettronica. Si deve disporre di anticorpi coniugati molecole fluorescenti, quali la fluoresceina isotiocianato o la rodamina isotiocianato (Brayton e Colwel, 1987), o coniugati con particelle, come sfere di oro colloidale opache agli elettroni. Utilizzando l'immunofluorescenza, si riusciti a distinguere clamidospore, conidi e frammenti miceliari della razza 4 di F. oxysporum f.sp. cubense da quelli prodotti dalla razze 1 e 2 (Wong et al., 1988). Il limite delle tecniche immunologiche e legata allinterazione stechiometrica di un anticorpo con un epitopo dellantigene. Spesso il riconoscimento avviene ma non rilevabile: la metodologia presenta una scarsa sensibilit. Lavvento delle tecniche immunoenzimatiche ha aumentato molto la sensibilit dello strumento sierodiagnostico. Si utilizza un anticorpo secondario, coniugato con un enzima, che riconosca la porzione costante (Fc) dell'anticorpo primario usato per riconoscere lantigene. La reazione immunologica rivelata da una reazione enzimatica, associata alla prima. Viene comunemente utilizzato l'enzima fosfatasi alcalina che, in presenza di appropriati reagenti, produce fosfato inorganico e porta alla formazione locale di un precipitato colorato. Il precipitato rivela la posizione dell'anticorpo secondario accoppiato all'enzima e, quindi, la posizione del complesso antigene-anticorpo a cui l'anticorpo secondario complessato. Lamplificazione del riconoscimento dovuto alla
45
caratteristica dellenzima che agendo cataliticamente genera un eccesso di prodotto visibile. Il test ELISA (enzyme linked immunosorbent assay) stata la tecnica originale. Essa prevedeva lassorbimento dellantigene su un supporto e la sua rivelazione con il sistema dellanticorpo primario e del secondario coniugato. Sono state successivamente sviluppate varianti pi efficienti. Uno dei metodi pi usati il DAS-ELISA (double antibody sandwich - enzyme linked immunosorbent assay), che utilizza lanticorpo primario adsorbito su una micropiastra per legare nel pozzetto lantigene (se presente) e il sistema primario-secondario per la rivelazione (Vruggink e Van Vuurde, 1990). Luso delle micropiastre permette unanalisi dei dati mediante colorimetri e lelaborazione con software adeguati agevola nel caso di prove con molti campioni I primi anticorpi monoclonali prodotti nei confronti di spore di F. oxysporum non permettevano di distinguere specifiche formae speciales o razze fisiologiche (Iannelli et al.,1983). Grazie alla tecnica della tolleranza immunologica successivamente gli stessi ricercatori produssero un antisiero capace di distinguere, tramite ELISA, isolati di F. oxysporum f.sp. lycopersici da isolati di F. oxysporum f.sp. dianthi (Del Sorbo, 1993). Sono stati messi a punto due saggi, uno per il riconoscimento specifico di F. oxysporum f.sp. cucumerinum ed un altro per tutte le specie di Fusarium (Kitagawa et al., 1989). Unaltra applicazione dellELISA stata messa a punto sviluppando un antisiero verso F. culmorum che riconosce tutte le specie di Fusarium ma non reagisce con altre patogeni del colletto e pu essere usato per determinare la biomassa totale delle specie di Fusarium che attaccano la regione basale dello stelo (Beyer et al., 1993) Il DIBA (dot immunobinding assay) un saggio derivato dallELISA. Si utilizzano membrane di nitrocellulosa come matrice adsorbente su cui si pongono gocce dei campioni estratti. Gli antigeni contenuti nei campioni si legano allanticorpo legato alla matrice. Per la rivelazione si utilizzano reazioni enzimatiche che dopo lidrolisi di substrati opportuni liberano prodotti insolubili, solo nei campioni positivi (Alberts et al., 1995). Arie et al. (1993) ha messo a
46
punto un metodo che combina la coltura fungina con i saggi immunologici per l'identificazione dei patogeni nel terreno. Una membrana viene posta sul substrato nutritivo contenuto in una capsula Petri, quindi viene cosparsa da una diluizione del campione di terreno. Dopo 20 ore, si recupera la membrana che viene sottoposta messa in contatto con l'anticorpo specifico. I propaguli dei funghi che si sono sviluppati durante l'incubazione hanno rilasciato gli antigeni che si sono fissati alla membrana, e possono cos essere rilevati. Questa metodica di rilevamento indiretto stata utilizzata per rilevare F. oxysporum f.sp. narcissi (Dewey e Thornton, 1995) e altre specie di Fusarium (Arie et al., 1993) 8.3. - Analisi degli acidi nucleici Le metodiche descritte sino ad ora hanno lobiettivo di evidenziare differenze tra due o pi individui per lo studio filogenetico o per lidentificazione del microrganismo coinvolto nellinfezione e quindi presente in un determinato campione. La fonte di biodiversit per definizione il DNA e tutte le tecniche diagnostiche viste fino ad ora si basavano sullinterpretazione di differenze fenotipiche collegate in ultima analisi a differenze intrinseche del genoma. E evidente che disporre di metodologie di indagine che scrutino la fonte di informazione permette di fare delle analisi pi precise e maggiormente esaustive. Lanalisi degli acidi nucleici, di fatto, incominci il suo corso nel 1944, quando Avery e i suoi collaboratori individuarono nel DNA il materiale genetico universale e definitivamente quando Watson e Crick scoprirono la sua struttura tridimensionale. Da quelle prime analisi, lindagine del DNA ha avuto innumerevoli evoluzioni ed entrato in modo innovativo in ogni settore della ricerca. La diagnostica fitopatologica, con un certo ritardo, si adeguata adottando le moderne tecniche molecolari con rapidit crescente e affiancandole alle tecniche gi di uso corrente. I vantaggi della diagnostica mediante analisi degli acidi nucleici risiedono nelle caratteristiche di elevata sensibilit, specificit, nella rapidit relativa di esecuzione e nella capacit predittiva precoce. Gli svantaggi sono legati
47
fondamentalmente ai costi di sviluppo delle tecniche al settore di interesse e la mancanza di sistemi diagnostici applicabili in condizioni di campo. In un settore come la difesa delle colture queste qualit hanno un importanza superiore agli altri settori perch ci si trova ad operare in un ambiente fortemente condizionato da agenti interferenti (biotici ed abiotici), in cui la disponibilit finanziaria spesso limitata e in cui la conoscenza tecnica media degli operatori agricoli spesso non sufficiente. La ricerca e levoluzione delle tecniche molecolari sta progressivamente orientandosi verso la soddisfazione dei criteri ottimali di riduzione dei costi e facilit duso. Di seguito verranno considerati i principali sistemi di diagnosi fitopatologica basati sugli acidi nucleici e le loro applicazioni per il riconoscimento di funghi fitopatogeni. 8.3.1. - Cariotipizzazione elettroforetica Lanalisi del cariotipo negli organismi fungini possibile tramite lo studio al microscopio ma tecnicamente complessa per le piccole dimensioni dei cromosomi. La mappatura genica possibile ma presenta dei problemi legati alla disponibilit di marcatori molecolari adeguati allo studio con incroci e ricombinazione parasessuale (Skinner et al., 1991). Tuttavia oggi sono disponibili tecniche per lanalisi del cariotipo basate sulla separazione elettroforetica dei cromosomi. Lelettroforesi in campo pulsato (PFGE: Pulsed Field Gel Electrophoresis) hanno aperto nuove prospettive per l'analisi genetica dei microrganismi. Uno dei problemi principali, superato con il PFGE, stata la separazione di macromolecole (cromosomi) di dimensioni variabili da circa 100 kb a oltre 10 Mb, non separabili in un campo elettrico unidirezionale, in quanto la loro velocit non pi proporzionale alla lunghezza. Le notevoli dimensioni delle molecole, inoltre, non ne consentono la migrazione elettroforetica attraverso pori molecolari del gel di agarosio. Con l'elettroforesi in campo pulsato si riescono a separare le macromolecole sottoponendole a campi elettrici che si alternano in due o pi direzioni (Vollrath e Davis, 1987). Poich, in un campo elettrico, il tempo necessario per una molecola per
48
cambiare il suo orientamento spaziale in seguito proporzionale alla sua dimensione possibile, separare molecole di diversa dimensione variando l'intensit di campo la concentrazione di agarosio, il numero di vettori che si alternano, l'angolo tra essi compreso, ed altre variabili (Mills e McCluskey, 1990). Le evoluzioni tecniche del PFGE hanno dato origine all OFAGE (Orthogonal Field Alternation Gel Electrophoresis) di Carle e Olson (1985), alla FIGE (Field Inversion Gel Electrophoresis) di Carle et al. (1986) ed alla TAFE (Tranverse Alternating Field Electrophoresis) di Gardiner et al. (1986). La tecnica, derivata, pi utilizzata per lanalisi elettroforetica del cariotipo il CHEF (Contourclamped Homogenous Electric Field; Chu et al. 1986) che, utilizza 24 elettrodi disposti attorno al gel di agarosio a formare un esagono. Tale tecnica di migrazione permette la risoluzione delle grandi molecole cromosomiche variando l'orientamento dell'angolo di campo fra 60 e 120. I geli di agarosio possono essere colorati con bromuro di etidio e le bande di DNA, osservabili mediante lirraggiamento con UV formano per ciascun campione il cariotipo elettroforetico. Il CHEF stato inizialmente applicato per analizzare i cromosomi di Saccharomyces cerevisiae (Schwartz e Cantor, 1984), ma stata poi estesa allanalisi di numerosi altri funghi (Mills e McCluskey, 1990). Si potuta evidenziare la notevole variabilit nel numero e nel peso molecolare dei cromosomi fungini riscontrabile anche tra isolati appartenenti allo stesso genere, specie, forma specialis e razza (Mills e McCluskey, 1990). Gli svantaggi della tecnica sono principalmente legati ai tempi molto lunghi di separazione elettroforetica e alla esperienza richiesta per la preparazione dei campioni. Da un punto di vista diagnostico massale di fatto scarsamente applicabile. E pero una tecnica altamente informativa se accoppiata con sonde molecolari cromosoma-specifiche, utili per la caratterizzazione di funghi fitopatogeni.
49
8.3.2. - Analisi dei profili di restrizione Le endonucleasi di restrizione sono enzimi batterici purificati che riconoscono specificamente una sequenza, variabile da quattro a otto nucleotidi, sul DNA e la tagliano in due frammenti. Oggi sul mercato sono disponibili oltre cento tipologie di enzimi di restrizione ed possibile utilizzarli come ausilio nellanalisi a scopo diagnostico. Il numero e le dimensioni dei frammenti di restrizione originati dal taglio di un campione di DNA riflettono direttamente la sequenza del DNA. Si pu, analizzando i profili ottenuti, separati mediante elettroforesi, ottenere informazioni di tipo diagnostico (Magnano di San Lio et al., 1995). LRFLP (Restriction Fragment Length Polymorphism), si basa sul principio che mutazioni sulla sequenza del DNA possano alterare i siti di riconoscimento degli enzimi di restrizione e quindi generare profili elettroforetici diversi (Nathans e Smith, 1975). L'RFLP ha permesso lo studio dei polimorfismi a livello genomico solo nei batteri, dotati di genoma ridotto (E. coli.: 3 x 104 bp). Le elevate dimensioni del genoma fungino, al contrario, non consentono la risoluzione di tutti i frammenti di restrizione generati e si ottiene un profilo non informativo (smear). Per superare questo inconveniente si ricorso allanalisi di frammenti digeriti provenienti dal DNA mitocondriale o ribosomiale. Si devono comunque utilizzare enzimi a bassa frequenza di taglio (rare cutters), per creare un ridotto numero di bande, facilmente risolvibili per via elettroforetica. Il DNA mitocondriale dei funghi una molecola di DNA a doppio filamento circolare, lunga meno di 100 kb. E una molecola che presenta regioni altamente conservate e regioni altamente variabili. E', quindi una molecola che si presta facilmente allanalisi delle relazione filogenetiche tra isolati e popolazioni strettamente correlate (Kimura, 1968; Taylor, 1986). Nel caso specifico di F. oxysporum (Kistler et al., 1987; Kim et al., 1990, 1991 e 1992; Jacobson e Gordon, 1990) si potuto organizzare isolati provenienti dalla stessa area geografica o aventi lo stesso spettro di ospiti in gruppi geneticamente omogenei.
50
I limiti applicativi dellanalisi RFLP, seppure ristretta al mtDNA, risiedono nella scarsa sensibilit del metodo e nellassenza di predittivit: occorre sempre coltivare il fungo in purezza per disporre di DNA altamente purificato (Magnano di San Lio et al., 1995). 8.3.3. - Ibridazione degli acidi nucleici Libridazione degli acidi nucleici un fenomeno di interazione, con diversi gradi di specificit, tra due singoli filamenti di DNA o RNA, che costituiscono una doppia elica stabile. E un fenomeno influenzato da fattori, quali lomologia di sequenza (complementariet), la temperatura e la concentrazione salina: la formazione dellibrido promossa tra sequenze altamente complementari, dalle basse temperature e bassa concentrazione salina (Schildkraut et al., 1961; Wetmur, 1991). Il DNA bersaglio viene deposto direttamente su una membrana di nylon o di nitrocellulosa (dot blot), o in seguito a separazione elettroforetica su gel di agarosio di frammenti di restrizione viene trasferito alla membrana facendolo assorbire ad essa per capillarit (Southern blot; Southern, 1975). Il DNA, fissato alla membrana mediante irraggiamento con radiazioni U.V., denaturato e posto a contatto con una soluzione contenente la sonda nucleotidica marcata a singolo filamento. Dopo libridazione si eliminano con dei lavaggi i residui di sonda che non si legata al DNA bersaglio. La rivelazione condotta ponendo una lastra autoradiografica sulla membrana che si impressiona laddove avvenuta la formazione dellibrido. La sonda ideale deve essere particolarmente specifica con lorganismo da analizzare e non rilevare in maniera aspecifica campioni non bersaglio. In patologia vegetale si deve porre attenzione al tipo di sequenza scelta per costituire la sonda perch sequenze del patogeno potrebbero coesistere nelIa pianta, seppure a minor grado di omologia e questo potrebbe andare a detrimento del risultato finale di analisi. La scelta pu ricadere su frammenti clonati a caso dal DNA del microrganismo da identificare, come dimostrato in Fusarium spp. (Manicom et al., 1987), Pythium spp. (Martin, 1991) e Ophiosphaerella herpotricha (Sauer et al., 1993), o pu essere rappresentare
51
sequenze altamente ripetute nel genoma bersaglio, cos da permettere di evidenziare la presenza di un numero esiguo di propaguli fungini. E stata costituita una sonda costituita da un frammento di DNA mitocondriale di F. oxysporum f.sp. niveum per distingure in F. oxysporum agenti di fusariosi vascolari delle Cucurbitaceae, isolati appartenenti a cinque diverse formae speciales (Kim et al., 1993). La marcatura fredda delle sonde ha agevolato e reso pi sicuro il lavoro delloperatore: stato sviluppato un sistema di sonde marcate con fluorocromi per l'identificazione di alcune formae speciales di F. oxysporum (Nonomura et al. 1996). In base allo scopo si possono impiegare sonde di origine diversa: le sonde possono derivare dallo stesso organismo (sonde omologhe) o da organismi appartenenti a specie, o in senso lato, ad organismi diversi (sonde eterologhe; Michelmore e Hulbert, 1987). Possono essere usate come sonde per libridazione di sequenze di DNA genomico, rDNA, mtDNA e cpDNA. Le sonde specifiche per il DNA ribosomiale, sono particolarmente utili per lanalisi filogenetica fra gli eucarioti perch individuano sequenze altamente conservate e presenti in centinaia di copie. Esse sono in grado di distinguere individui a livello specifico o subspecifico (Kasuga e Mitchelson, 1994). I minisatelliti sono sequenze genomiche ripetute in tandem, impiegabili per differenziare isolati della stessa specie ma provenienti da diverse aree geografiche o aventi differente attitudine parassitaria (Jeffreys et al., 1985). Pur essendo una tecnica altamente specifica allatto pratico le tecniche di ibridazione molecolare si rivelano inadeguate per analisi diagnostiche rapide e su molti individui. Libridazione con sonde a DNA spesso collegata con la tecnica PCR PCR (Polymerase Chain Reaction). Mediante PCR possibile fornire sequenze nuove da utilizzare come sonde negli esperimenti di ibridazione: per l'identificazione del fungo ectomicorrizico Tuber magnatum, stata sviluppata una sonda, a partire da un frammento specifico di DNA, ottenuto con lamplificazione RAPD (Lanfranco et al. 1993). Recentemente stata messa a punto una sonda specifica per rilevare F.oxysporum f. sp. basilici agente di tracheofusariosi sul basilico (Chiocchetti et al., 1999).
52
8.3.4. - PCR e applicazioni La PCR (Polymerase Chain Reaction; Saiki et al., 1988) un metodo enzimatico per la sintesi in vitro di sequenze specifiche di DNA. La reazione enzimatica ricalca, in parte, il fenomeno della replicazione del DNA nucleare: un filamento di DNA viene ricopiato a partire da uno originale. Poich le DNA polimerasi, generalmente necessitano di inneschi oligonucletidici per iniziare la sintesi si utilizza questi per determinare la specificit della reazione. Nella miscela di reazione vengono posti a contatto in un tampone di reazione adeguato: il DNA bersaglio (target o template), due sequenze oligonucleotidiche di riconoscimento specifico (primer), i nucleotidi fosforilati (dNTPs), una DNA polimerasi termostabile (Taq, isolata dal batterio termofilo Thermophilus aquaticus). La reazione consiste in una successione di cicli composti da tre fasi distinte in base alla temperatura di reazione: 1) denaturazione. 2) ibridazione, 3) polimerizzazione. Essa viene condotta in un ciclatore termico capace di modificare la temperatura di reazione in modo programmato e molto rapidamente. 1) La fase di denaturazione permette di rendere le molecole a singolo filamento e rendere cos possibili le successive fasi; di norma la temperatura utilizzata 94C per un tempo variabile con le caratteristiche del DNA bersaglio e dello scopo di analisi. 2) L'ibridazione (annealing) la fase in cui gli oligonucletidi complementari si possono appaiare alle sequenze del DNA bersaglio, qualora esse siano presenti. La temperatura di questa fase critica e varia da 35 a 68 C nelle diverse tipologie di PCR. La temperatura di annealing viene calcolata sulla base della temperatura di fusione dei primer. 3) La polimerizzazione avviene a 72 C, temperatura alla quale la polimerasi in grado di sintetizzare, incorporando nucleotidi a stampo, una catena complementare al DNA bersaglio, creando un doppio filamento.
53
Queste tre fasi vengono ripetute per un numero di cicli successivi variabile tra i 30 e 45 (n) della durata ognuno di 2-5 minuti. Il risultato netto la sintesi di 2n copie (amplificazione) del DNA bersaglio delimitato come sequenza dai due primer: a partire da 1 pg-25 ng di DNA bersaglio si possono produrre masse di DNA pari a 1 g di prodotto finale (McPherson et al., 1992; Henson e French, 1993). La reazione condotta avvalendosi di primer specifici produce un prodotto specifico se presente la sequenza bersaglio nel campione analizzato se essa non presente non si ottiene alcun prodotto amplificato. La scelta dei primer fondamentale ai fini diagnostici e va effettuata seguendo gli stessi principi usati per scegliere le sonde molecolari (Oliver, 1993). Per la scelta dei primer oggi sono disponibili dei programmi appositi che tengono conto delle caratteristiche ottimali affinch un oligonucleotide sia adeguato dal punto di vista termodinamico. Il grado di omologia con regioni del DNA bersaglio inversamente proporzionale alle dimensioni del primer, quindi opportuno scegliere la lunghezza del primer adottando, nel caso di omologia massima, dimensioni di 18-30 bp. Il contenuto in guanina e citosina (G+C) deve essere pari ad almeno il 50% del totale e particolare riguardo va posto sulle interazioni intramolecolari e intermolecolari: le prime generano dei duplex interni mentre le seconde formano dei duplex tra i due primer ed entrambe sottraggono allequilibrio gli oligonucleotidi rallentando o bloccando la reazione (Henson e French, 1993). Le reazione di ibridazione specifiche e quelle intra- e intermolecolari sono caratterizzate da una energia di associazione/ dissociazione che influenzata dalla temperatura. Si pu quindi conosciuti tutte le variabili, determinare la temperatura di fusione del primer: la temperatura alla quale il 50% degli oligonucleotidi appaiata al DNA bersaglio. In letteratura si ritrovano moltissimi casi di applicazioni diagnostiche della PCR e come per tutte le tecniche sin qui discusse, anchessa ha fatto il suo ingresso in diagnostica fitopatologica in tempi pi recenti rispetto al suo utilizzo in campo biomedico. La PCR stata applicata allamplificazione di DNA genomico e proveniente da organelli. Nellambito dellamplificazione di frammenti genomici, gli studi filogenetici sui geni ribosomali hanno avuto grande successo per le loro
54
caratteristiche intrinseche di conservazione e variabilit nellambito degli stessi loci e dal punto di vista tecnico per la loro abbondanza: sono presenti in copie multiple nel genoma, il che li rende facilmente amplificabili (White et al., 1990; Bruns et al., 1991; Lee et al., 1992; Ward et al., 1993). I geni ribosomali negli eucarioti sono presenti in unit trascrizionali ripetute in tandem 60-200 volte per una lunghezza complessiva di 2-30 Kb (Bruns et al., 1991). Sono presenti nellordine una sequenza 18 S, una 5,8 S e una 28 S intervallate da regioni ITS (Internal Transcribed Spacer). Nelle regione trascritte si ritrovano regioni spaziatrici non trascritte NTS (Non Transcribed Spacer e tra le unit trascrizionali si ritrovano le regioni IGS (Inter Genic Spacer). Tutte queste tipologie presentano differenti gradi di conservazione, utilizzati per studi filogenetici. Si pu utilizzare la PCR, amplificando con primer universali designati sulla base del gene codificante lrDNA 16S nei procarioti e il 18S per gli eucarioti: in particolare, si sono dimostrati efficaci per la classificazione dei fitoplasmi (procarioti), patogeni del sistema floematico (Lee et al., 1993; Gundersen e Lee, 1996). Primer disegnati sulle sequenze del rDNA 18S ha permesso lamplificazione del DNA di funghi endomicorrizici vescicolo-arbuscolari (VAM) da estratti complessi, comprendenti DNA di piante e altri funghi (Simon et al. 1992). Le regioni ITS (Internal Transcribed Spacer) permettono di ottenere maggiori informazioni sulla variabilit genetica (White et al., 1990; O'Donnell, 1992). Le regioni NTS e le regioni spaziatrici intergeniche (IGS) presentano un grado di variabilit ancora maggiore. Quindi da un lato possibile amplificare con primer universali sequenze appartenenti a specie anche molto differenti e con le diverse regioni spaziatrici discriminare nellambito della stessa specie. Popolazioni patogene e non patogene di F. oxysporum associate alle radici sono state distinte grazie allanalisi mediante PCR applicata alle regioni IGS (Edel et al., 1997; Appel e Gordon, 1996). Luso della PCR applicata alle sequenze ribosomali stata di aiuto nella sistematica di Fusarium e numerose specie sono state catalogate con la PCR: F. sambucinum (O'Donnell 1992). Sono stati messi a punto sistemi di primer per la diagnosi in tempi brevi e il
55
riconoscimento di tre patogeni F. culmorum, F. graminearum e F. avenaceum, difficilmente distinguibili tra loro se presenti contemporaneamente sulla pianta infetta (Schilling et al., 1996). Partendo da precedenti sonde molecolari specifiche per F. oxysporum f.sp. canariensis, agente della fusariosi della palma da dattero, da cui sono stati disegnati primer specifici da utilizzare in analisi PCR (Plyler et al., 1999). Anche lo studio approfondito della distribuzione dei trasposoni in F. oxysporum ha permesso di trovare marcatori specifici. Usando le giunzioni degli elementi trasponibili, come marcatori amplificabili mediante PCR, sono stati sviluppati primer specifici per F. oxysporum f.sp. dianthi (Chiocchetti et al., 1999), che consentono la distinzione delle principali razze fisiologiche del patogeno e per F. oxysporum f.sp. albedinis, agente di fusariosi delle palme (Fernandez et al., 1999). La tecnica di base della reazione polimerasica a catena ha subito numerose varianti che ne hanno ampliato lutilizzo per ogni genere di studio. Vedremo nei prossimi paragrafi le principali varianti a questa tecnica. 8.3.4.a. - RAPD (Random Amplified Polymorphic DNA) Tale tecnica, con opportune variazioni, anche chiamata AP-PCR (Arbitrarily Primed PCR; Welsh e McClelland, 1990). Essa utile nel caso in cui non si abbiano informazioni preliminari sul polimorfismo genetico di un organismo e si vogliano confrontare molti individui (Williams et al., 1991). Si basa sull'impiego di un solo primer, in genere di 7-10 nucleotidi, per amplificare casualmente in corrispondenza di regioni omologhe nel DNA bersaglio. I prodotti di amplificazione che si ottengono sono imprevedibili nel numero e nel peso molecolare ma, ottenibili con notevole ripetitivit a parit di condizioni sperimentali. Gli amplimeri possono essere analizzati mediante elettroforesi su gel di agarosio o su gel di poliacrilammide. Le condizione di reazione sono meno stringenti che per una PCR tradizionale e prevedono: denaturazione a 94 C, annealing a 36 C ed estensione a 72 C. I singoli cicli sono di circa 1-2 minuti ma sono molto pi elevati di numero (Williams et al., 1990).
56
Nei funghi fitopatogeni, l'utilizzo dei RAPD ha consentito di differenziare razze diverse di Cochliobolus carbonum (Jones e Dunkle, 1993), C. orbiculare (Correll et al., 1993), P. herpotrichoides (Nicholson e Rezanoor, 1994), F. solani f.sp. cucurbitae (Crowhurst et al., 1991), F. oxysporum f.sp. pisi (Grajl-Martn et al., 1993), f.sp. dianthi (Manulis et al.,1994; Migheli et al., 1994 e 1996), f.sp. vasinfectum (Assigbetse et al., 1994), f.sp. ciceris (Kelly et al., 1994) e f.sp. basilici (Chiocchetti et al., 1999). La tecnica RAPD pu essere applicata nella sua variante SCARs (Sequence Characterized Amplified Region; Paran e Michelmore, 1993; Gressoff, 1995). Essa consente di risalire, tramite il clonaggio e le procedure di sequenziamento, alla sequenza di un amplimero valutato come specifico sulla base di confronti con altri individui. Sulla base della sequenza possono poi essere disegnati primer specifici, come stato fatto per la distinzione specie-specifica di isolati di F. culmorum, F. graminearum e F. avenaceum (Schilling et al., 1996) e per la distinzione della forma specialis basilici da altri isolati patogeni di F. oxysporum (Chiocchetti et al., in preparazione). 8.3.4.b. - DAF (DNA Amplification Fingerprinting) Si basa sull'uso di oligonucleotidi molto corti di 5-7 residui come primer per la reazione di amplificazione (Caetano-Anolls et al., 1995). La probabilit che questi primer incontrino regioni complementari sul DNA bersaglio molto alta e vengono quindi generate miscele di amplimeri assai pi complesse. Per la separazione dei frammenti consigliabile lelettroforesi su gel di poliacrilammide ed unanalisi con programmi analizzatori di immagine per lidentificazione delle bande. Per tale motivo questa variante della PCR non stata ancora applicata su larga scala per il riconoscimento di funghi fitopatogeni, ma rappresenta un sistema di identificazione ad alta qualit informativa, che nel caso delluomo capace di discriminare tra individui consanguinei (Caetano-Anolls et al., 1995).
57
8.3.4.c. - REP-PCR Essa utilizza primer specifici disegnati su sequenze ripetute e conservate nel genoma (Versalovic et al., 1991). Le sequenze usate sono palindromiche extrageniche ripetute (REP), le sequenze intergeniche di consenso ripetute degli enterobatteri (ERIC) e gli elementi BOX. I primer sono disegnati tra due elementi ripetuti adiacenti in modo da amplificare il DNA tra loro compreso. Non sono pero da escludere fenomeni di appaiamento con sequenze omologhe non bersaglio (Gillings e Holley, 1997). La collezione di primer (REP, ERIC e BOX) stata utilizzata per analizzare diversi specie di batteri e specificamente batteri delle piante Gram positivi e Gram negativi (Clark et al., 1998; deBruijn, 1992; Louws et al., 1994 e 1998; Sadowsky et al., 1996). La presenza di sequenze ERIC-simili stata ritrovata anche in funghi quali Aspergillus: ci ha permesso la differenziazione di isolati strettamente correlati. In F. oxysporum stata ritrovata la presenza di elementi ERIC e grazie allanalisi della sua distribuzione, 60 isolati non patogeni sono stati raggruppati in 27 tipi: risultati analoghi erano stati ottenuti con analisi RFLP e RAPD (Edel et al.,1995). 8.3.4.d. - AFLP (Amplified Fragment Length Polymorphism) Le potenzialit della PCR possono essere aumentate, laddove la specificit dei primer non sia sufficiente a distinguer due o pi isolati, accoppiando ad essa la digestione enzimatica con endonucleasi di restrizione (Louws et al., 1999). Questa tecnica stata utilizzata con successo nel fingerprint di batteri fitopatogeni (Bragard et al., 1997; Clerc et al., 1998; Janssen et al., 1996). La tecnica AFLP prevede tre passaggi: 1) la digestione del DNA genomico; 2) laggiunta alle estremit digerite di adattatori (linker) con sequenza complementare al primer della successiva reazione; 3) lamplificazione del frammento usando due primer complementari alladattatore e in parte, 1-2 bp, alla sequenza bersaglio (Vos et al., 1995).
58
8.3.4.e. - ARDRA (Amplified Ribosomal DNA Restriction Analysis) E analoga allAFLP ma viene utilizzata per amplificare regioni di rDNA, successivamente digerite con enzimi ad alta frequenza di taglio per determinare la diversit e per identificare isolati dello stesso genere o specie (Louws et al., 1999). utilizzata nello studio della filogenesi batterica (Manceau e Horvalis, 1997; Nesme et al., 1995; Ponsonnet e Nesme, 1994; Vaneechoutte et al., 1992) ed in diagnostica fitopatologica, dove viene usata per l'identificazione e la classificazione di fitoplasmi (Lee et al., 1993; Schneider et al., 1993). Alcune specie di Fusarium sono state identificate tramite lARDRA (Edel et al., 1996). Levoluzione delle tecniche di PCR ha portato ad integrare con buoni risultati le tecniche immunodiagnostiche con quelle molecolari. 8.3.4.f. - Immunocapture Essa utilizza anticorpi monoclonali per concentrare il potyvirus del vaiolo della prugna isolato da piante infette: in un secondo tempo il virus viene amplificato mediante primer specifici disegnati su sequenze di RNA delle particelle virali (Wetzel et al., 1992). Per lidentificazione di Salmonella in alcuni campioni sono state utilizzate particelle magnetiche adsorbite con anticorpi, al fine di concentrare il batterio e quindi stata eseguita lamplificazione con primer specifici (Widjojoatmodjo et al., 1991). 8.3.4.g. - Immuno-PCR Lindividuazione del patogeno legata allanticorpo. La sensibilit della reazione immunologica viene aumentata coniugando lanticorpo con una sequenza di DNA mediante un ponte molecolare proteina A-streptavidinabiotina (Sano et al., 1992). Viene effettuata una reazione di PCR sulla sequenza etichetta (Henson e French, 1993). La sensibilit della reazione aumentata anche di 250 volte rispetto ad unamplificazione diretta dei virus vegetali (Wetzel et al., 1992). Limmuno-PCR oltre alla potenzialit intrinseca della tecnica non richiede la conoscenza di una specifica sequenza del patogeno ma richiede solo che si disponga di un anticorpo specifico.
59
I vantaggi in termini di sensibilit della PCR non sono comparabili nessunaltra tecnica oggi a disposizione. La PCR presenta, per, determinati difetti che possono precluderne un uso corretto. Pur essendo altamente specifica si presta ad evidenziare dei falsi positivi, in genere legati a problemi di contaminazione da acidi nucleici derivate da precedenti manipolazioni nellambiente di lavoro. La presenza di inibitori della reazione pu generare falsi negativi. Per ovviare alle contaminazioni necessario seguire una serie di pratiche di laboratorio che tendano a minimizzare questi eventi e approntare sistemi di decontaminazione da DNA di tipo preventivo. Per il problema degli inibitori occorre mettere a punto metodi di estrazione del DNA bersaglio rapidi, puliti e di facile attuazione a partire da tessuti vegetali infetti o dal terreno. Il problema che forse pi limitava lutilizzo della PCR su vasta scala era la disponibilit di sistemi di automazione, come avvenuto per lELISA, per analisi di un gran numero di campioni. Sono noti da tempo ed ora reperibili sul mercato dei sistemi automatici di amplificazione che permettono lanalisi degli amplificati senza ricorrere allelettroforesi su gel dagarosio che mediante luso di fluorocromi. 8.3.4.h. - Automatizzazione della PCR Le tecnologie disponibili riguardano la polarizzazione in fluorescenza (Gibson et al., 1997), il sistena TaqMan (Holland et al., 1991; Livak et al., 1995) e i Molecular Beacons (Tyagi e Kramer, 1996; Tyagi et al., 1998). I molecular beacons sono sonde oligonucleotidiche coniugate alle due estremit con un fluoroforo ed una molecola "quencher". Quando la sonda non ibridata al DNA bersaglio, adotta, in soluzione, una conformazione a forcina; e cosi strutturata il fluoroforo ed il "quencher" vengono a trovarsi appaiati: questultimo assorbe la radiazione del fluoroforo. In questo modo la sonda incapace di emettere fluorescenza. Quando, invece, la sonda incontra la molecola bersaglio, si forma un ibrido pi stabile della forcina, la cui rigidit e lunghezza precludono l'appaiamento dei terminali ed il fluoroforo, non pi appaiato al quencher, pu
60
emettere la radiazione luminosa, che aumenta fino a 100 volte (Bonnet et al., 1999). I "molecular beacons" hanno finora trovato ampio utilizzo in campo biomedico e in genetica umana (Tyagi et al., 1998; Kostrikis et al., 1998 e 1998), nello studio di retrovirus (Vet et al., 1999), in ecologia (Schofield et al., 1997) e nella localizzazione di specifico RNA all'interno di cellule viventi (Matsuo, 1998). Il sistema antagonista del TaqMan il sistema LightCycler messo a punto dalla Roche. Esso sfrutta la fluorescenza emessa da un agente intercalante, il SYBR Green I, che in grado di legarsi al solco minore della doppia elica del DNA. Questa molecola, intercalandosi al DNA, emette un segnale che diviene sempre crescente finche la reazione di PCR prosegue e nuove molecole diventano disponibili per il marcatore. Permette quindi di seguire in tempo reale una reazione di PCR e di fornire, previa costituzione di curve di taratura, un risultato quantitativo.
9. - ELEMENTI TRASPONIBILI La prima prova di esistenza dei trasposoni risale al 1956. Barbara McClintock (1956), una studiosa statunitense, osservando delle pannocchie di mais, scopr che alcune cariossidi che erano prive di colore, o che erano colorate a macchie ritornavano al fenotipo iniziale dopo un certo periodo di tempo. Suppose che si il fenotipo revertante fosse indotto da geni che si spostavano, e li battezz jumping genes. Allinizio il fenomeno non venne compreso in termini molecolari e solo molti anni dopo, con lisolamento dellelemento Ac in mais nellallele wx-n7, si riusci a spiegare il ruolo dei trasposoni in quella bizzarra mutazione (Behrens et al., 1984). I trasposoni sono sequenze discrete di DNA capaci di spostarsi direttamente da un locus ad un altro del genoma. Il salto del trasposone in genere un evento raro ma pu aumentare di frequenza sotto stimoli ambientali che inducano stress nella cellula. Leffetto dello spostamento di un elemento mobile una mutazione che interessa grandi regioni del genoma. In seguito ad un salto si
61
possono creare inserzioni, delezioni, duplicazioni, inversioni, traslocazioni e riarrangiamenti genici e cromosomici. La mutazione pi frequente si verifica quando un trasposone viene escisso e si integra in un locus genico, alterandolo. Tale cambiamento pu portare ad attivazione o inattivazione di allelica a seconda del punto di inserzione dellelemento mobile. Tuttavia sono stati fatti studi che dimostrano un ruolo attivo dei trasposoni nel riarrangiamento genico e nellevoluzione (Berge e Howe, 1989). Dai tempi della prima scoperta (McClintock, 1956), i trasposoni sono stati oggetto intenso di studio per le potenziali capacit mutagene e di veicolazione di DNA. Fin'ora, sono stati ritrovati trasposoni in tutti gli organismi viventi in cui si sono cercati. 9.1. - Tipologie di elementi trasponibili Gli elementi trasponibili sono stati divisi in tre classi da Finnegan (1989) sulla base del loro differente meccanismo di trasposizione. La classe I comprende tutti gli elementi che si traspongono mediante un la trascrizione inversa di un intermedio a RNA. La classe II comprende tutti gli elementi che si traspongono mediante trasposizione con intermedio a DNA (trasposoni di tipo batterico). Esistono tuttavia elementi mobili che non rientrano in tali modelli e rientrano nellinsieme degli elementi non classificabili. Nei paragrafi seguenti se ne dar una breve descrizione. 9.2. -Elementi mobili di classe I Tali elementi si traspongono tramite un intermedio a RNA che viene trascritto in una copia di DNA (cDNA) tramite una trascrittasi inversa il cui gene spesso codificato dallo stesso elemento mobile. Per tale modalit sono chiamati pi semplicemente retrotrasposoni. Essi sono divisibili in base alla caratteristiche strutturali primarie in: retrotrasposoni con LTR (A) e retrotrasposoni sprovvisti di LTR (B). A) I retrotrasposoni con LTR si distinguono dagli altri perch portano sequenze alle estremit di 334 bp orientate nello stesso verso. Le sequenze
62
LTR (long terminal repeats) hanno la funzione di promotori forti. Sono note due tipologie di retrotrasposoni: la famiglia Ty3 e la famiglia Ty1/copia. Gli elementi della famiglia Ty3 presentano i domini codificanti per la poliproteina pol in un ordine simile a quello ritrovato nei retrovirus e generalmente presentano una cornice di lettura supplementare che codifica per una proteina simile a env dei retrovirus (Finnegan, 1994). Tra i pi noti v il retrotrasposone gipsy, oggi considerato un retrovirus. Gli elementi Ty1/copia prendono il loro nome dai due retrotrasposoni identificati in per la prima volta in lievito e presentano, rispetto a Ty3, una diversa organizzazione dei domini del gene pol B) I retrotrasposoni sprovvisti di LTR non possiedono sequenze promotori forti alle estremit ma presentano al 3' di una sequenza di poly A o poly A/T. Sono stati ritrovati nella maggior parte degli organismi eucarioti e sono presenti nel genoma in migliaia di copie. Tra i retrotrasposoni privi di LTR ci sono il gruppo dei SINEs, dei LINEs e gli pseudogeni I SINEs (Short Interspersed Elements) sono elementi mobili di piccola taglia che hanno perso la di codificare proteine (Deininger, 1989). Tra i SINEs le sequenze AluI sono quelle pi presenti nel genoma dei primati (Houck et al., 1979). I LINEs (Long Interspersed Elments) sono sequenze identificate per la prima volta nei mammiferi (Hutchinson, 1989). Contengono frequentemente due cornici di lettura aperte simili ai geni gag e pol dei retrovirus. Gli pseudogeni sono geni non funzionali con sequenza strettamente correlata ai normali geni funzionali. Essi mancano di sequenze regolatrici non presentano introni e presentano una coda di polyA al 3. Sono probabilmente legati allintegrazione nel genoma di mRNA, ricopiato in cDNA, per trascrizione inversa. 9.3. - Elementi di classe II Si traspongono con un meccanismo DNA/DNA e possiedono alle estremit 5 e 3 sequenze terminali ripetute e invertite di lunghezza variabile a seconda dellelemento (ITR). Tra le sequenze ripetute terminali si ritrova un gene per
63
necessario la trasposizione, codificante per lenzima trasposasi. Sono stati i primi trasposoni ad essere scoperti e sono tipici degli organismi procarioti. Esistono le sequenze di inserzione (IS) identificate analizzando mutazioni spontanee e i trasposoni Tn implicati nella trasmissione di resistenza agli antibiotici. La struttura delle sequenze IS formata da una regione centrale codificante (trasposasi), e due regioni terminali ove sono poste due sequenze ripetute e invertite (ITR). I trasposoni batterici posseggono o una struttura semplice simile a quella delle IS ad eccezione della presenza di geni supplementari (ad esempio Tn3; Sheratt, 1989), oppure una struttura pi complessa (trasposoni composti) dove la regione centrale codificante fiancheggiata da due IS. Il Tn10 presenta alle estremit due copie di IS10 (Kleckner, 1989). Negli eucarioti sono presenti un gran numero di trasposoni di classe II e la loro struttura simile a quella delle IS dei batteri. Possono essere suddivisi in due sottoclassi: gli elementi contenenti il dominio DDE o non contenenti il dominio DDE (Capy et al., 1998). Il dominio DDE la sequenza codificante per una proteina indispensabile allo svolgimento della funzione catalitica della trasposasi. Il dominio presenta tre aminoacidi invarianti, due acidi aspartici (D) distanti 90 aminoacidi e un acido glutammico (E) separato dall' ultimo aspartato di 34 o 35 residui aminoacidici. E un dominio abbastanza conservato e lo si ritrova in alcune famiglie di IS e nelle integrasi dei retroelementi (Fayet et al., 1990; Kulkosky et al., 1992). Nellambito degli elementi provvisti di dominio DDE si ritrova la superfamiglia degli elementi Tc1-mariner. Ad essa appartengono gli elementi di tipo Tc1 (TLE) scoperti in Caenorhabditis elegans (Emmons et al., 1983) e gli elementi mariner (MLE), ritrovati per la prima volta in Drosophila mauritiana (Jacobson et al., 1986). Sono elementi ubiquitari e rappresentati in tutti i tipi di organismi nei protozoi, nell'uomo, nei funghi e recentemente sono stati identificati in una pianta (Jarvik e Lark, 1998). Hanno una lunghezza limitata e si inseriscono in sequenze in cui presente un dinucleotide specifico (TA). Codificano generalmente per la sola trasposasi di circa 350 aminoacidi. Gli elementi
64
mariner hanno motivi terminali variabili ma la lunghezza delle ITR costante (24-30 bp). Tra gli elementi con dominio DDE sono state ritrovate sequenze raggruppabili nella superfamiglia Pogo: Sulla base della similitudine delle sequenze nella regione del dominio DDE, Smit e Riggs (1996) hanno recentemente raggruppato in una sola famiglia gli elementi pogo di D. melanogaster (Tudor et al., 1992), tiggers1 e 2 nell' uomo (Smit e Riggs , 1996; Robertson 1996), Tc4 e Tc5 dei nematodi (Moermann e Waterson, 1989) e l' elemento Fot1 del fungo F. oxysporum (Daboussi et al., 1992), La seconda categoria di elementi di Classe II sono quelli senza DDE. Tra questi, la superfamiglia hAT che proviene dal raggruppamento degli elementi hobo di Drosophila, Ac di mais e Tam3 della bocca di leone (Calvi et al., 1991). Sono elementi ubiquitari presenti nelle piante, negli insetti, nei pesci, nei nematodi, nei funghi (Kempken e Kuck, 1996). Tutti hanno delle sequenze ITR corte e motivi subterminali ripetuti, un sito di inserzione duplicato di 8 bp, e omologia di sequenza nella regione C-terminale della trasposasi (Calvi et al., 1991). La superfamiglia degli elementi P prende il nome dal primo elemento identificato in Drosophila. E lungo 2,9 Kb, presenta ITR di 31bp e possiede 4 esoni codificanti per la trasposasi (Engels, 1989). L'elemento attivo solo nelle cellule della linea germinale dellinsetto. Attualmente sono stati trovati in drosofilidi e in qualche altro dittero (Kidwell, 1994) La superfamiglia CACTA presente solo nelle piante. Questi elementi hanno ITR che iniziano con la sequenza CACTA. Fino ad oggi sono stati ritrovati En (Spm) in mais (Gierl, 1996), Tam1 nella bocca di leone (Nacken et al., 1991), Tgm in soia (Vodkin et al., 1996), nel pisello Pis1 (Shirsat, 1988), Tnr3 nel riso (Motohashi et al., 1996). L'elemento Mu stato identificato in una linea particolarmente instabile di mais (Robertson, 1978), questa famiglia comprende pi sottofamiglie composte di elementi attivi e inattivi che presentano tra loro omologie delle ITR di circa 200 bp (Young et al., 1994).
65
9.4. - Elementi non classificabili Questi elementi non sono chiaramente classificabili perch non ci sono sufficienti informazioni sulla loro modalit di trasposizione. Possiedono ITR pi o meno lunghe e ci suggerisce una similitudine con gli elementi trasponibili di classe II. Tra i diversi gruppi ci sono i Foldback (FB) che, identificati in Drosophila, costituiscono una famiglia eterogenea nella struttura. Hanno lunghe ripetizioni terminali invertite, costituite da numerose ripetizioni in tandem di sequenze da 10 a 31 bp (Bingham e Zachar, 1989). Sono presenti nel riccio di mare (Liebermanne et al., 1983), nei nematodi (Yuan et al., 1991), nelle piante (Oosumi e Belknap, 1997). La grande diffusione suggerisce che siano ubiquitari tra gli eucarioti. Gli elementi MITE (Miniature Inverted Transposable Elements) sono stati identificati per la prima volta in pianta (Bureau e Wessler, 1992). Hanno una sequenza relativamente breve, non hanno capacit codificante e non esistono prove dirette e indirette della loro trasposizione. Presentano, tuttavia come gli elementi di classe II, delle ripetizioni terminali invertite di circa 20 bp, sono ricchi in TA (Bureau et al., 1996). Sono stati trovati nei funghi filamentosi (Yeadon e Catcheside, 1995), nell' uomo (Morgan, 1995) e nella mosca della febbre gialla (Tu, 1997).
10. - GLI ELEMENTI TRASPONIBILI IN FUSARIUM OXYSPORUM Nel genoma di F. oxysporum sono state trovate almeno sei famiglie diverse di elementi mobili appartenenti alla classe dei trasposoni batterici e dei retrotrasposoni. Sono stati identificati mediante il clonaggio in sequenze ripetute (elementi Foret1 e Palm) e mediante linserzione nel gene per la nitrato riduttasi (elementi Fot1, Fot2, impala e Hop) Foret1 un retrotrasposone provvisto di LTR e possiede sequenze interne alle LTR omologhe ai geni retrovirali pol. Lelemento isolato difettivo per la
66
trasposizione e sequenziato ha evidenziato una sequenza mutata con molti stop codon e mutazioni frameshift (Julien et al., 1992) Lelemento Palm un retrotrasposone con LTR di tipo LINE isolato da un F. oxysporum f.sp. elaeidis che attacca la palma da olio, durante un analisi di popolazioni per fingerprinting (Mouyna et al., 1996). Nel genoma di F. oxysporum sono presenti inoltre i trasposoni di tipo batterico Fot1, Fot2, impala e Hop. Hanno la stesa struttura formata da due ITR lunghe da 27 a 96 bp e una regione centrale che codifica in modo putativo per una trasposasi. Si possono distinguere per la modalit di duplicazione del sito di inserzione in due gruppi: Fot1, Fot2 e impala duplicano un dinucleotide TA mentre Hop si distingue dagli altri. Fot1 presente in circa 100 copie in alcuni genomi di isolati di campo. E lungo 1928 bp possiede ITR di 44 bp e la regione centrale codifica per una trasposasi il cui mRNA misura 1.6 kb. E una famiglia molto omogenea di trasposoni e fa parte della superfamiglia degli elementi Tc1 di Caenorhabditis elegans. Fot2 presente in circa 100 copie in alcuni genomi di isolati di campo. E lungo 2.1 kb e possiede ITR di 66 bp. Le ITR hanno 25 bp che sono omologhe con quelle di Fot1 e si verosimilmente tra loro correlati. Lelemento impala stato isolato, per la prima volta, in due mutanti niaD (Langin et al., 1995); E lungo 1280 bp, ha ITR di 27 bp ed ha una regione centrale contenente una ORF priva di introni. Il polipeptide dedotto dall'analisi dellORF rivela delle similitudine con le trasposasi degli elementi della superfamiglia Tc1-mariner, come pure le sequenze ITR. Le copie di impala presentano spesso polimorfismo di dimensioni e di profilo di restrizione, identificabili nelle tre sottofamiglie E, D, F che raggruppano al loro interno le differenti strutture degli elementi impala finora caratterizzati (Hua-Van et al.,1998). La famiglia di impala presenta poche copie sparse per il genoma (16). Lelemento Hop lungo 3.5bp ed ha ITR di 96 bp. Durante i fenomeni di escissione-integrazione produce una duplicazione di 7 bp, ritrovata nel gene niaD.
67
11. - SCOPO DEL LAVORO Sono ormai in aumento gli agenti di contenimento biologico messi in commercio, tra i quali anche isolati attivi di F. oxysporum. Come stato discusso nel capitolo precedente, un microrganismo, usato per la lotta biologica, assimilabile ad un prodotto fitosanitario e come tale deve essere sottoposto alla legislazione vigente valevole per i fitofarmaci (Decreto Legislativo numero 194 del 17/5/95). Uno dei requisiti principali per un microrganismo la sua identificabilit e distinguibilit da quelli presenti nellambiente o immessi dalluomo in maniera deliberata, nonch la sua rintracciabilit, cos da poter seguire il suo destino nello spazio e nel tempo. A tal fine stata provata la strada delletichettatura genica, con geni codificanti per la resistenza allantibiotico igromicina e al fungicida benomyl, inserite mediante processi di trasformazione. Il ricorso alla modificazione genica ha fornito le possibilit di individuare un isolato anche molti mesi dopo il suo rilascio (Gullino et al., 1995; Gullino e Migheli, 1999). I funghi cos modificati rientrano per sotto la disciplina del Decreto Legislativo 91/93 e sono, quindi, sottoposti a molteplici restrizioni che ne limitano lo sfruttamento economico. Si pensato, quindi, di adottare una strategia non modificativa per i ceppi antagonisti con pi elevata capacit di contenimento biologico. Per le sue potenzialit di sensibilit, economicit e rapidit si scelto di adottare la PCR (Saiki et al., 1988). In questo lavoro si voluto caratterizzare un insieme di isolati antagonisti di F. oxysporum per poterli distinguere, in maniera univoca, ai fini della registrazione o di un eventuale protezione brevettuale. Lidentificazione mediante lanalisi degli acidi nucleici rende pi efficienti e agevoli le procedure di valutazione del rischio, richieste dalla legge a seguito dellimmissione deliberata nellambiente di un microrganismo. A questo proposito, stata fatta, dapprima, unanalisi mediante la tecnica RAPD per caratterizzare tutti gli isolati antagonisti e successivamente si cercato di isolare una sequenza caratteristica per gli isolati 233/2 e 245 wt, dotati di buona attivit antagonistica.
68
Lanalisi RAPD non ha permesso lindividuazione di una banda unica e non si potuto applicare la strategia SCAR per gli isolati prescelti (vedi il paragrafo 8.3.4.a., cap.1). Si quindi optato per la costituzione di primer specifici disegnati sulle regioni fiancheggianti di inserzioni uniche degli elementi trasponibili impala e Fot1, come nei lavori precedenti su Fusarium oxysporum f. sp. albedinis (Fernandez et al., 1999) e su Fusarium oxysporum f.sp. dianthi (Chiocchetti et al., 1999). Si scelto di concentrarsi maggiormente sul trasposone impala analizzandone la distribuzione nei diversi isolati per evidenziare bande specifiche e distinte da quelle presenti nelle diverse forme speciali cos da usarle per costruire gli oligonucleotidi specifici.
69
II
CAPITOLO 2
2. - MATERIALI E METODI. 2.1. - Isolati fungini utilizzati Per lanalisi tramite Southern blot, la PCR inversa, le prove di PCR e i RAPD, stata usata una collezione di F. oxysporum saprofiti, isolati da terreni repressivi liguri e dotati attivit antagonistica verso diverse formae speciales di F. oxysporum. Sono stati inclusi nel lavoro anche i singoli rappresentanti di F. oxysporum ff.spp. basilici, canariensis, cepae, cyclaminis, gladioli, lilii, lycopersici, melonis, pisi, radicis-lycopersici e tulipae. La collezione mantenuta presso il Di.Va.P.R.A.- PatoIogia Vegetale, Facolt di Agraria di Torino su agar patata-destrosio (PDA) sotto olio minerale a 12C (Tabella 1). 2.2. - Coltura degli isolati fungini e liofilizzazione Con unansa sterile, lavorando sotto cappa a flusso laminare orizzontale, si preleva un frammento di micelio da un tubo contenente PDA (vedi Soluzioni e Terreni), e Io si trasferisce in capsule Petri contenenti idrolizzato di caseina. Le colture liquide vengono mantenute a temperatura ambiente o in incubatore a 24C senza agitazione per un periodo di 5-7 giorni. Il micelio viene successivamente filtrato, per eliminare residui di terreno di crescita e di acqua, trasferendolo in un filtro di porcellana del diametro di circa 10 cm, contenente sul fondo una membrana di nylon con pori del diametro di 100 m (entrambi precedentemente autoclavati) e adagiato su una beuta collegata ad una pompa a vuoto (BioRad). Dopo l'operazione il micelio ottenuto viene trasferito su carta bibula sterile e quando ben asciutto viene messo in una capsula Petri sterile e congelato mediante azoto liquido (-196C, vedi Soluzioni e Terreni). Le capsule vengono conservate a -80C per 15 minuti, quindi si trasferiscono nel
70
II
liofilizzatore (Heto) per 2-3 giorni. Il micelio liofilizzato viene conservato in tubi da 4 ml a -20C. 2.3. - Estrazione di DNA genomico 2.3.1. - Estrazione di DNA genomico da micelio liofilizzato Si preleva circa 0,5 g di micelio liofilizzato che viene frammentato tramite un pestello in microprovette eppendorf da 2 ml; si risospende in 1 ml di soluzione di lisi (vedi Soluzioni e Terreni), quindi viene incubato a 37C per 30 minuti in un bagnetto termostatico (VELP Scientifica). Dopo l'incubazione e l'inattivazione dell'enzima a 70 C per 15 minuti, vengono aggiunti 100 l di acetato di potassio 5M, miscelati con l'aiuto del vortex e posti in un bagno di ghiaccio per 30 minuti. I campioni vengono poi centrifugati a 13000 r.p.m. per 15 minuti in una microcentrifuga (Sanyo), il surnatante viene estratto con un volume di fenolo : cloroformio : isoamilalcool (25:24:1) (Sigma) e i tubi vengono posti su un agitatore basculante (Platform shaker STR6, Stuart Scientific) per almeno 15 minuti, centrifugati a 13000 r.p.m. per 15 minuti sotto cappa chimica; il surnatante prelevato viene posto in una nuova eppendorf cui viene aggiunto mezzo volume di acetato di ammonio 7,5 M (vedi Soluzioni e Terreni). Dopo 15 minuti in un bagno di ghiaccio e la centrifugazione a 13000 r.p.m. per altri 15 minuti, al sumatante prelevato viene aggiunto un volume di isopropanolo (Merck). Il DNA, sotto forma di gomitolo biancastro, viene trasferito in una nuova eppendorf contenente 1 ml di etanolo (Merck) 75% freddo e centrifugato per 5 minuti a 13000 r.p.m.. Viene aspirato il surnatante e lasciato asciugare il pellet, quest'ultimo viene risospeso in 200 l di TE pH 8 (vedi Soluzioni e Terreni) e incubato a 65C per una pi rapida risolubilizzazione. Il DNA viene poi conservato a 4 C per ulteriori manipolazioni. 2.3.2. - Estrazione di DNA rapida da impiegare in reazioni di PCR E' possibile ottenere un'amplificazione di DNA mediante la PCR partendo da poche copie di DNA stampo; perci utile disporre di un metodo di estrazione
71
II
rapido. Si preleva un cm quadrato di micelio da isolati cresciuti su PDA e si frantuma con un pestello in una microprovetta, cui era stata aggiunta un volume di 300 l di soluzione L (vedi Soluzioni e Terreni). La microprovetta viene poi passata in azoto liquido, quindi in acqua per alcuni istanti: tale procedimento viene ripetuto per tre volte, per facilitare lestrazione del DNA. Si procede alla bollitura per 10 minuti del campione e alla sua centrifugazione per 5 minuti a 13000 r.p.m. Vengono prelevati 200 l di surnatante che viene poi utilizzato come DNA-stampo per le reazioni di PCR. 2.3.3. - Preparazione di un gel di agaroso I DNA estratti vengono separati su un gel di agaroso (Sea Kem) 0,8% ottenuto sciogliendo, nelle adeguate proporzioni, agaroso in TAE 1X (vedi Soluzioni e Terreni) ed aggiungendo 1 l/100 mI di bromuro di etidio (10 mg/ml, Sigma)., Si utilizzano 5 l di 1 kb Ladder (GibcoBRL), avente bande di peso molecolare comprese tra 500 bp e 12000 bp, cos da quantificare visivamente il DNA estratto Si aggiunge ad ogni campione il tampone di carica 6X (vedi Soluzioni e Terreni), quindi si caricano i pozzetti del gel, gi posto nella vaschetta elettroforetica contenente TAE 1X, con 1l di campione. Si applica una differenza di potenziale tra gli elettrodi pari a circa 100 V e si effettua la migrazione del DNA. Alla fine della corsa elettroforetica le immagini sono rilevate con il Gel Doc 1000 Molecular Analyst (Bio-Rad). La quantificazione del DNA estratto avviene confrontando l'intensit della banda di DNA estratto con il segnale del Ladder di taglia 1.6 kb (50 ng) 2.4. - Southern blot 2.4.1. - Digestione del DNA genomico e precipitazione Vengono digeriti circa 10 g di DNA genomico con 50 U di Xho I o Bgl II a 37 C per 16 ore in un volume di 100-200 l. Un'aliquota di 5 l viene prelevata al termine del tempo di incubazione e controllata su gel di agaroso per verificarne lavvenuta digestione. Il DNA viene successivamente precipitato con 1/10 di
72
II
volume, della reazione di digestione, di sodio acetato 3M pH 5,2 (vedi Soluzioni e Terreni) e 2 volumi di etanolo 96% pre-refrigerato a 20 C. Le microprovette vengono mantenute a 80 C per 30 minuti e poi centrifugate a 13000 r.p.m. per 15 minuti a 4 C. Viene effettuato un lavaggio del pellet con 1 ml di etanolo 70% e di nuovo un passaggio in centrifuga a 13000 r.p.m. per 5 minuti a 4 C. Viene aspirato il surnatante e il pellet viene seccato allaria per qualche minuto. Il DNA viene risospeso in 10 l di TE. Il DNA cos preparato viene separato per elettroforesi su gel di agaroso allo 0.8% in tampone TAE 1X (vedi Appendice "Soluzioni e terreni). La separazione sulla base della taglia molecolare dei diversi frammenti di digestione viene eseguita lentamente per 12 ore a 35 V in una camera elettroforetica (Pharmacia). 2.4.2. - Trasferimento su membrana dei DNA genomici digeriti I campioni di DNA genomico digerito e separato per elettroforesi sono stati trasferiti su una membrana di del tipo Nylon Transfer Membrane "Hybond N" (Amersham): un supporto pi maneggevole per i successivi esperimenti di marcatura. Per il trasferimento si utilizzato un sistema a depressione (BioRad) e come protocollo quello fornito dalla ditta. Vengono preparati dei rettangoli di carta 3M (Whatman) dello spessore di 3 mm e la membrana di trasferimento entrambi tagliati delle dimensioni del gel e poi imbibiti in acqua milliQ (Millipore) sterile. Si dispongono nellordine sul supporto di trasferimento la carta, la membrana, la finestra con guarnizioni, il gel, il coperchio di tenuta. Viene poi applicata una depressione di 15 mm Hg mediante una pompa a vuoto (Vacuum pump, Biorad). Si ricopre il gel con una soluzione di depurinazione (vedi Appendice "Soluzioni e terreni) per 4 minuti ripetendo l'operazione per 2 volte. Si lava il gel con acqua sterile e poi lo si ricopre con una soluzione di denaturazione (vedi Appendice "Soluzioni e terreni) per 20 minuti. Si ricopre poi con una soluzione di neutralizzazione per 5 minuti (vedi Appendice "Soluzioni e terreni). Si termina ricoprendo con una soluzione di trasferimento (vedi Appendice "Soluzioni e terreni) per 30 minuti. Al termine la pompa a
73
II
vuoto viene spenta e smontato lapparato si preleva la membrana che viene mantenuta per 5 minuti su carta bibula imbibita di SSC 2X orientata verso lalto (vedi Appendice "Soluzioni e terreni). Viene fatta asciugare rapidamente e poi fissata mediante raggi UV a 260 nm per 5 minuti tramite un trans-illuminatore (2011 MACROVUE transilluminator, LKB). La membrana pu essere utilizzata subito per diverse manipolazioni o conservata a 20 C avvolta in una pellicola per alimenti (I.DI. Torino) 2.4.3. - Marcatura delle sonde a DNA con digossigenina Per la marcatura delle sonde si adottato kit DIG DNA Labeling Kit (Roche). Il DNA viene marcato usando la digossigenina-11-dUTP, usando come metodo di marcatura il "Random Primed DNA Labeling": la reazione di marcatura in grado di produrre una sufficiente quantit di sonda marcata in breve tempo. Durante La reazione di marcatura una molecola di digossigenina incorporata ogni 20-25 nucleotidi della sonda di interesse. Previa separazione e purificazione da gel di agaroso dei prodotti di PCR mediante kit Jet-quick (Genomed) 1 g DNA viene marcato secondo il protocollo della ditta: le provette contenenti il DNA da marcare vengono fatte bollire per 10 minuti per denaturarlo e repentinamente poste in ghiaccio per 5 minuti al fine di evitarne la rinaturazione. Vengono poi aggiunti i componenti della miscela di reazione nelle giuste proporzioni: in un volume finale di reazione di 20 l si utilizzano 2 U enzimatiche del frammento Klenow, esanucleotidi e dNTP. Viene fatta avvenire la reazione a 37C, in un bagnetto termostatico, per tutta la notte. Il risultato finale una sonda marcata non radioattiva. 2.4.4. - Precipitazione del DNA marcato con digossigenina Il DNA marcato viene precipitato aggiungendo 1/10 di volume di 200 mM EDTA pH 8 (vedi Soluzioni e Terreni), 1/10 di volume di 4 M cloruro di litio (Merck) e 3 volumi di etanolo 96% freddo, pre-refrigerato a 20C.Le microprovette vengono mantenute a 80 C per 30 minuti e poi centrifugate a 13000 r.p.m.
74
II
per 15 minuti a 4 C. Viene effettuato un lavaggio del pellet con 1 ml di etanolo 70% e di nuovo centrifugate a 13000 r.p.m. per 5 minuti a 4 C. Viene aspirato il surnatante e il pellet viene seccato allaria per qualche minuto. Il DNA marcato viene risospeso in 20 l di TE pH 8 (vedi Appendice "Soluzioni e terreni). Si procede poi alla quantificazione della sonda marcata mediante il kit di controllo annesso a quello di marcatura (Roche) I primer impiegati ed i relativi pesi molecolari dei frammenti, con cui sono state ottenute le sonde utilizzate, sono elencati nelle tabelle 2 e 4. 2.4.5. - Controllo del DNA marcato Per verificare una corretta marcatura delle sonde e per quantificarle si utilizzato il DNA di controllo fornito con il kit DIG DNA Labeling Kit (Roche). Vengono eseguite cinque diluizioni del DNA di controllo (stock [20 ng/l]): (A) 1:20, (B) 1:10 della diluizione A, (C) 1:100 della diluizione B, (D) 1:1000 della diluizione C, (E) 1:10000 della diluizione D. Il DNA marcato viene portato alla medesima concentrazione della diluizione A di controllo (1 ng/l); una volta ottenuta questa concentrazione, si effettuano le medesime diluizioni del DNA di controllo (A, B,C, D, E).Le diluizioni vengono fatte in un tampone fornito nel kit. Si pone un 1 l di ogni diluizione su una frammento di membrana Nylon Transfer Membrane "Hybond N" (Amersham), si lascia asciugare alI'aria e successivamente si pone per 5 minuti sul transilluminatore a U.V., per fissare il DNA all membrana. Il filtro viene reidratato con MBS 1X (vedi Appendice "Soluzioni e terreni), e poi incubato con BR 1% (vedi Soluzioni e Terreni) in agitazione a 37C per 30 minuti. In seguito vie mantenuto in agitazione su un piano basculante (Platform shaker STR6, Stuart Scientific) a temperatura ambiente alla presenza di anticorpo anti-digossigenina (1:10000 in BR 1%). Dopo due lavaggi di 10 minuti ciascuno in MBS 1X, si trasferisce la membrana in una vaschetta con il tampone AP (vedi Soluzioni e Terreni) e si lascia basculare per 5 minuti. Si sostituisce il tampone AP con il substrato lumigeno CSPD (Roche) (1:100 in tampone AP, (vedi Appendice Soluzioni e terreni), al buio, e si lascia basculare per 5 minuti. Si pone la membrana a contatto con
75
II
una lastra autoradiografica (Kodak) e si effettua una esposizione di per 30 minuti a 37C. Si sviluppa la lastra in camera oscura ponendola per 2 minuti nella soluzione di sviluppo (vedi Appendice Soluzioni e terreni"), per qualche secondo in acqua e per 2 minuti nella soluzione di fissaggio (vedi Appendice Soluzioni e terreni"). Si confrontano sulla lastra ottenuta il DNA di controllo con il DNA marcato e si stabilisce la quantit di sonda ottenuta. 2.4.6. - Preibridazione e ibridazione La membrana, su cui stato trasferito il DNA digerito, viene incubata a 65C con un soluzione di preibridazione (vedi Appendice Soluzioni e terreni), per un tempo variabile da 2 a 6 ore secondo la dimensione della membrana. La membrana viene successivamente posta in una soluzione di ibridazione (vedi Appendice Soluzioni e terreni). La sonda viene fatta bollire per 10 minuti e subito posta in ghiaccio per 5 minuti per evitare la rinaturazione, quindi unita alla quantit di liquido di preibridazione necessaria e sufficiente a ricoprire la membrana, aggiungendo un 1 l di sonda (stock 20ng/l) per ogni mI di liquido di preibridazione. Viene eseguita una ibridazione per tutta la notte. 2.4.7. - Sviluppo della chemioluminescenza Si recupera il liquido di preibridazione con la sonda, che pu essere congelata e riutilizzata pi volte per un periodo di circa sei mesi. La membrana viene sottoposta a 4 lavaggi (due con la soluzione 1 e due con la soluzione 2: vedi Soluzioni e Terreni) di 10 minuti ciascuno a 65C, pre-riscaldando le soluzioni a 65C. La membrana viene poi posta su carta bibula sterile e mantenuta a basculare per 5 minuti nella soluzione di lavaggio (vedi Soluzioni e Terreni). Si incuba poi in BR 1% filtrato tramite della carta bibula, a 37C in agitazione per 1 ora. Viene sostituito il BR 1% con una soluzione di BR 1% pi mAb antidigossigenina (Roche) in proporzioni di 1:10000, Incubando per 1 ora a temperatura ambiente in agitazione. La membrana vie poi trattata con la soluzione di lavaggio per quattro volte, ciascuna di 10 minuti, in agitazione a
76
II
temperatura ambiente. La membrana trasferita in una vaschetta pulita e lasciata basculare per 5 minuti in tampone AP. Viene poi incubata in una soluzione contenente lumigeno CSPD (Roche) diluito 1:100 in tampone AP, in agitazione per 5 minuti al buio. Si preparano due lucidi e si pone tra loro la membrana, poi si mette il tutto a contatto con una lastra autoradiografica (Kodak) e si effettua una esposizione di 1 ora a 37C. Si sviluppa la lastra in camera oscura ponendola per 2 minuti nella soluzione di sviluppo (vedi Appendice .Soluzioni e terreni"), per qualche secondo in acqua e per 2 minuti nella soluzione di fissaggio (vedi Appendice Soluzioni e terreni"). 2.5. - Reazione di pcr inversa (IPCR) 2.5.1. - Digestione del DNA genomico e precipitazione Si digerisce una aliquota di DNA genomico pari a 5 g con 20 U di Bgl II (BioLabs), alla presenza di tampone Sure cut buffer (BioLabs), in un volume finale di reazione di 100 l. Si incuba a 37C per tutta la notte. Si precipita il DNA digerito aggiungendo 1/10 di volume di 3M sodio acetato pH 5.2 (vedi Soluzioni e Terreni) e poi 2 volumi di etanolo 96%, pre-refrigerato a 20 C. Le microprovette vengono mantenute a 80 C per 30 minuti e poi centrifugate a 13000 r.p.m. per 15 minuti a 4 C. Viene effettuato un lavaggio del pellet con 1 ml di etanolo 70% e di nuovo centrifugate a 13000 r.p.m. per 5 minuti a 4 C. Viene aspirato il surnatante e il pellet viene seccato allaria per qualche minuto. Il DNA viene risospeso in un volume di 10 l di acqua milliQ sterile. 2.5.2. - Autocircolarizzazione dei DNA digeriti Si procede alla circolarizzazione dei singoli frammenti di digestione su loro stessi. Per facilitare la auto ligazione ed evitare la formazione di concatameri, si esegue la reazione in un volume relativamente ampio pari a 100 l e a bassa temperatura. Si utilizza tutto il DNA precedentemente digerito aggiungendo 1 U di T4 DNA ligasi (Roche) nel tampone di reazione specifico per lenzima. Si incuba la reazione per tutta la notte a 4C. Il DNA viene poi precipitato con 1/2
77
II
volume di ammonio acetato 7.5 M e 3 volumi di etanolo 96%, lavato e centrifugato con le modalit precedentemente descritte. Si risospende il campione in 10 l di acqua milliQ sterile. 2.5.3. - Reazione di IPCR Si utilizzano come DNA stampo una quantit variabile tra 100 ng e i 500 ng di DNA circolarizzato. Vengono preparate due miscele di reazione. La prima contiene: 350 nm di ciascun nucleotide (Roche), 500 nm di sequenze oligonucleotidiche IMP1 con IMP2 disegnati sulle porzioni terminali di impala, diretti verso lesterno e utilizzati per isolare le regioni fiancheggianti il trasposone (Figura 1), 5 l di DNA e acqua fino ad un volume di 25 l. La seconda contiene: 5 l di tampone di reazione e 0.75 l di Taq (predisposti dal kit: Expand long template PCR system, Roche). Viene aliquotata la prima miscela in un tubo di reazione a parete sottile (Thin walled PCR tube, Roche), vengono aggiunti 50 l di olio minerale (Sigma) sterile. Il tubo viene posto poi nel blocchetto del termociclatore Perkin Elmer Cetus 9600 (Emeryville, CA). Quando la temperatura raggiunge i 94 C si aggiungono 25 l della seconda miscela di reazione (Chow et al., 92). Le reazioni sono soggette a 35 cicli di denaturazione a 94C per 10 secondi, all'appaiamento dei primer sulle regioni omologhe con un passaggio di temperatura da 68C a 60C (2 cicli a 68C, 2 cicli a 65C, 1 ciclo a 63C e 30 cicli a 60C) per 1,5 minuti, alI'estensione a 68C per 2 minuti ed un autoestensione di 10 secondi per i restanti 30 cicli. Le reazioni sono completate con un'incubazione a 68C per 7 minuti. I prodotti di amplificazione vengono controllati tramite elettroforesi su gel di agaroso (Sea Kem). Qualora l'amplificazione non dia un risultato apprezzabile su gel si esegue una PCR annidata con gli oligonucleotidi IMP5 e IMP6 posizionati pi esternamente su impala rispetto a IMP1 e IMP2 (Figura 1).
78
II
2.6. - Clonaggio dei prodotti dell'IPCR 2.6.1. - Separazione delle bande su gel L'intero volume di reazione della PCR inversa viene caricato su un gel di agaroso all'1%. Le bande di interesse vengono tagliate con un bisturi, ponendo il gel sul transilluminatore a raggi U.V. (LKB). 2.6.2. - Estrazione del DNA da gel Si esegue una purificazione da gel di agaroso mediante il kit Jet-quick (Genomed). Si pesa il frammento tagliato dal gel cui si aggiungono 300 l di soluzione L1 (predisposta dal kit) per ogni 100 mg di banda e si pongono le microprovette eppendorf in un bagno ad acqua calda a 50C per almeno 15 minuti, agitandole ogni 3 minuti, cos da favorire la solubilizzazione del gel. Si trasferiscono 700 l di tale sospensione sulla colonnina fornita dal kit e si centrifuga immediatamente a 13000 r.p.m. per 1 minuto. Si aggiungono 700 l di soluzione L2 (predisposta dal kit) e si lascia riposare per 5 minuti. Si eseguono due centrifugate a 13000 r.p.m. di 1 minuto ciascuna e si trasferisce la colonnina su un tubo sterile da 1,5 mI; Si aggiungono 50 l di acqua milliQ sterile preriscaldata a 50C, quindi si centrifuga per 2 minuti. Il DNA cos ottenuto controllato su un gel di agaroso all'1 % mediante elettroforesi. 2.6.3. - Ligazione del DNA nel vettore Il DNA purificato da gel stato successivamente clonato nel vettore plasmidico pGEM-T (Promega). L'enzima Taq polimerasi (Expand long template PCR system, Roche) utilizzato per la PCR inversa aggiunge, una volta su due, alle estremit di ogni amplimero una coda di adenina (tail). Si utilizzato, per il clonaggio, pGEM-T (Fig. 2), un vettore avente una lunghezza di 3 Kb (50 ng/l). Tale plasmide viene venduto gi linearizzato e provvisto di una timina pendula all'estremit 5' e al 3'. Il sito di clonaggio stato generato, all'interno del gene codificante per il peptide del cistrone lacZ, mediante un taglio con l'enzima
79
II
EcoRV e successiva aggiunta di una coda di timina (tailing). La frequenza di ligazione del frammento di IPCR stata cos notevolmente aumentata. La reazione di ligazione avviene in un volume di reazione di 10 l. Si prepara una miscela contenente: 5 l di tampone 2X della T4 DNA ligasi, 1 l di T4 DNA ligasi, 1 l di vettore pGEM-T (reagenti predisposti dal kit), 1 l di prodotto di IPCR. La reazione viene fatta avvenire a 4 C per tutta la notte, cos da evitare la richiusura su se stesso del plasmide e quindi massimizzare, dopo la trasformazione, il numero di trasformanti positivi. 2.6.4. - Trasformazione Il prodotto della reazione di ligazione viene utilizzato, senza ulteriori manipolazioni, per trasformare un ceppo di E. coli competenti JM109 (JM109 High Efficiency Competent Cells, Promega). Tali cellule sono state manipolato in maniera da contenere l'episoma F', che reca i geni proAB, complementanti la auxotrofia per la prolina di tale ceppo. Lo stesso plasmide porta la porzione lacIqZM15, richiesta per la selezione "blu/bianco" delle colonie trasformate. Si fanno scongelare le cellule competenti in ghiaccio (circa 5 minuti). Si centrifuga per pochi secondi a 13000 r.p.m. il prodotto di ligazione, per raccoglierlo sul fondo del tubo. Si trasferiscono 2 l della miscela in una nuova microprovetta pre-raffreddata in un bagno di ghiaccio. Ad essi si aggiungono 50 l di cellule e si agita gentilmente. Si incuba in ghiaccio per 20 minuti. Si esegue uno shock termico a 42 C per 45-50 secondi e si rimette la provetta in ghiaccio per 2 minuti. Si aggiunge successivamente un volume di 950 l di terreno di coltura SOC (vedi Soluzioni e Terreni) e si incuba a 37C, in agitazione, per 90 minuti. Si piastra un volume di 100 l di reazione di trasformazione su terreno LBA supplementato con ampicillina (Merck), con 30 l, rispettivamente, di IPTG 100 mM (indolilpropil-tio-beta-galattoside, BioRad) e di X-gal 50 mg/ml (5bromo-4-cloro-3-indolil-beta-galattoside, Promega) (vedi Soluzioni e Terreni). L'IPTG un induttore stabile che si lega al repressore dell'operone lattosio, determinando una attiva trascrizione dei geni Z, Y e A. L'X-GAL un analogo sintetico del lattosio che viene metabolizzato dalla galattosidasi, dando come
80
II
prodotto di reazione un pigmento blu. La selezione viene fatta sfruttando il fenomeno della mancata complementazione del lacZ con il lac operon. Le cellule trasformate con il vettore auto richiuso daranno colonie blu, per la capacit di complementare l'operone. Le cellule trasformate con il plasmide e l'inserto appariranno bianche, dal momento che l'inserto lede la funzionalit del lacZ, che incapace di dare complementazione produrr un enzima inattivo incapace di metabolizzare l'X-GAL. Vengono prelevate le colonie bianche mediante uno stuzzicadenti sterile e vengono fatte crescere in 3 ml di terreno LB con ampicillina (vedi Soluzioni e Terreni), in agitazione a 37C per tutta la notte. 2.6.5. - Protocollo per estrazione dl DNA plasmidico dalle cellule batteriche trasformate Le colonie batteriche accresciutesi vengono utilizzate per l'estrazione del DNA plasmidico, cos da verificare l'avvenuta trasformazione. Si trasferisce in una eppendorf sterile un volume pari a circa 1.5 ml di coltura batterica, lo si centrifuga per 30 secondi a 13000 r.p.m. e si elimina il surnatante. Vengono aggiunti 100 l di soluzione R (vedi Soluzioni e Terreni), si risospende il pellet batterico aiutandosi con un vortex (VELP Scientifica), si aggiungono 100 l di soluzione L di lisi (vedi Soluzioni e Terreni), si agita con il vortex, indi si aggiungono 400 l di soluzione N (vedi Soluzioni e Terreni), per precipitare la parete batterica, agitando nuovamente. Si centrifuga per 5 minuti a 13000 r p.m., si recupera il surnatante in un eppendorf sterile da 1,5 mI e si aggiungono 600 l di etanolo 96 %, miscelando per inversione. Si centrifuga per 10 minuti a 13000 r. p.m., si aspira il surnatante e si aggiunge 1 mI di etanolo 75%, miscelando per inversione. Si centrifuga per altri 10 secondi, si elimina il surnatante e si lascia asciugare all'aria il pellet di DNA. Si risospende il pellet con 20 l di TE (pH 8) (vedi Soluzioni e Terreni).
81
II
2.6.6. - Digestioni di controllo Per verificare che i cloni batterici, derivati una colonia bianca, presentino l'inserto di interesse si eseguono delle digestioni differenziali del DNA plasmidico estratto. Si utilizzano gli enzimi di restrizione Nco I e Pst I che sono in grado di riconoscere siti unici di restrizione sul polylinker di pGEM-T, rispettivamente a sinistra e a destra del punto di clonaggio dell'inserto. Si prepara una miscela di reazione, in un volume di 20 l, contenente: 5 l di DNA plasmidico estratto, 1 U di Nco I e Pst I, 2 l di tampone specifico per gli enzimi e acqua milliQ sterile. Si incuba a 37C per 1 ora in un bagnetto termostatico. Si evidenziano i prodotti di digestione su un gel di agaroso all'1% mediante elettroforesi in tamponeTAE 1X (vedi Soluzioni e Terreni). 2.6.7. - Protocollo di estrazione di DNA plasmidico da cellule batteriche in media scala Per la estrazione plasmidica stato utilizzato un protocollo predisposto dalla ditta fornitrice il kit di purificazione Nucleobond AX100 (Macherey-Nagel). Per le soluzioni utilizzate consultare l'Appendice "Soluzioni e terreni". Viene inoculata una colonia batterica in 100 ml di terreno LB (vedi Soluzioni e Terreni) con ampicillina in una beuta da 250 ml. Si mantiene in agitazione a 37C per tutta la notte fino al raggiungimento di una densit di coltura adeguata. Per conservare i cloni si preparano degli strisci su piastra di LBA pi ampicillina e si preparano anche delle provette con glicerolo: si miscelano 400 l di glicerolo sterile con 600 l di coltura batterica (Sworder et al., 1976). La restante coltura viene centrifugata a 5000 r.p.m. a 4C per 5 minuti, in contenitori (Sorvall) sterili. Il surnatante viene eliminato ed il pellet risospeso in 4 mI di tampone S1 (vedi Soluzioni e Terreni), cui viene preventivamente addizionata100 g/ml di RNAsi A (Roche). La sospensione batterica viene trasferita in tubi pi piccoli (Beckman) resistenti alla centrifugazione. Si aggiunge un volume di 4 ml di tampone S2 (vedi Soluzioni e Terreni), quindi si miscela con cautela e si incuba a temperatura ambiente per 5 minuti. Si
82
II
aggiungono, poi, 4 mI di tampone S3 (vedi Soluzioni e Terreni), si agita delicatamente, mediante inversione dei tubi per 6-8 volte, al fine di ottenere una sospensione omogenea. Si pongono, successivamente i tubi in un bagno di ghiaccio per 5 minuti e si centrifugano a 9500 r.p.m. a 4C per 38 minuti. Nel frattempo si equilibra la colonnina NUCLEOBOND AX 100 (Macherey-Nagel) con 2 mI di tampone N2 a pH 6.3 (vedi Soluzioni e Terreni). Terminata la centrifugazione, si rimuove il surnatante con cura evitando di aspirare il precipitato bianco, lo si filtra con una garza sterile e lo si carica sulla colonnina. Si lava la colonnina per 2 volte con 4 mI di tampone N3 a pH 6.3 (vedi Soluzioni e Terreni). Il DNA plasmidico viene eluito con 3 mI di tampone N5 a pH 8.5 (vedi Soluzioni e Terreni), trasferendo la colonnina su un nuovo tubo. Il DNA plasmidico viene poi precipitato aggiungendo 0,7 volumi ( con riferimento al volume di eluizione) di isopropanolo, preequilibrato a temperatura ambiente. La soluzione centrifugata a 9500 r.p.m. per 15 minuti a 4C. Viene eliminato il surnatante e il DNA viene lavato con una adeguata quantit di etanolo 70%, girando il tubo un paio di volte. Dopo aver nuovamente eliminato il surnatante, il pellet risospeso con 200 l di TE (vedi Appendice .Soluzioni e terreni"). Vengono successivamente compiuti dei controlli, mediante restrizione con enzimi, per confermare la positivit del clone. 2.7. - Sequenziamento del DNA Il sequenziamento dei DNA stato fatto dal Servizio di Sequenziamento del Dipartimento di Biologia-LIMA presso il BIOINDUSTRY PARK CANAVESE S.r.l. (Colleretto Giacosa). La reazione di sequenza viene ottenuta utilizzando termociclatori PCR PTC-100 MJ Research e PCR Express Thermal Cycler della Hybaid. Il sequenziamento viene effettuato su CEQ 2000 Analysis System (Beckman Coulter). Il sistema CEQ 2000 si basa su un processo di elettroforesi capillare completamente automatizzato, ideato per determinare la sequenza in basi di DNA mediante l'uso di un kit specifico "4 color-labeled". Il rilevamento avviene tramite fluorescenza laser-indotta in quattro canali spettrali distinti. I dati grezzi sono archiviati in formati compatibili con le applicazioni pi diffuse.
83
II
Le sequenze sono state analizzate con il programma Chromas 1.45 (Mc Carthy). Le sequenze delle regioni fiancheggianti impala, identificate nei cloni ACQ1 e ACQ2 sono state depositate presso la GenBank (numeri di accesso: DA REGISTRARE, rispettivamente). 2.8. - Reazione di PCR 2.8.1. - Amplificazione RAPD-PCR L'analisi molecolare tramite RAPD stata condotta con 7 ceppi di F. oxysporum antagonisti isolati da terreni repressivi liguri, (Tabella 1), per valutare l'omogeneit dei profili di amplificazione, utilizzando il DNA estratto da micelio liofilizzato e da rappresentanti di ff.spp. di Fusarium oxysporum. Le amplificazioni sono state realizzate in 15 l di miscela di reazione, contenenti 10 mM Tris-HCl, pH 9,0, 1,5 mM MgCl2, 50 mM KCl, 0,1% Triton X-100, 0,01% (w/v) gelatina, 60 M di ciascun dATP, dCTP, dGTP e dTTP (Promega), 5 pM di primer, circa 0,1 ng di DNA bersaglio e 0,75 U di SuperTaq DNA polimerasi (HT Biotechnology). Trenta oligonucleotidi di 10 basi ognuno (Operon Technologies Inc.; Tabella 3 ) sono stati provati come primer. Tutti i reagenti (tranne il DNA) sono stati preventivamente miscelati e le aliquote distribuite in tubi Eppendorf di reazione da 0,5 ml, prima dell'aggiunta del DNA bersaglio e dell'olio minerale sterile per PCR (Sigma), per ridurre il rischio di contaminazione. La quantit di DNA stata valutata attentamente per migliorare la specificit e ripetibilit della reazione RAPD secondo i criteri suggeriti da Davin-Regli et al. (1995) La reazione di amplificazione stata effettuata nel Cetus Gene Amp PCR System 9600 (Perkin-Elmer). E' stato usato un programma di amplificazione di 45 cicli che prevedeva un ciclo di denaturazione di 2 minuti e 30 secondi seguito da 30 secondi di denaturazione a 94C, 1 minuto a 36C e 2 minuti a 72C.
84
II
2.8.2. - Utilizzo di primers specifici per il ceppo antagonista 233/2 Gli oligonucleotidi specifici sono stati disegnati sulle sequenze fiancheggianti utilizzando il programma Input3 (prodotto da Richard Resnick, http://wwwgenome.wi.mit.edu/cgi-bin/primer/primer3_www.cgi). Per le reazioni di PCR si utilizzato DNA genomico fungino purificato da micelio liofilizzato, in quantit da 1 l a 5 l corrispondenti a 100 ng- 500 ng. Per ciascuna coppia di primer sono tate approntate reazioni di PCR in condizioni diverse: (Tabella 5). E' stata messa a punto una reazione di PCR "hot start", per fare in modo che l'enzima entri in contatto con gli altri reagenti solo alla temperatura adeguata. Per ci vengono preparate due miscele di reazione. La prima contiene: 350 nm di ciascun nucleotide (Fynzymes), 500 nm di sequenze oligonucleotidiche disegnate, rispettivamente sulla sequenza fiancheggiante di ACQ1 e di ACQ2 (Tabella 4) e sulla porzione terminale di impala, diretti verso lesterno acqua fino ad un volume di 11,5 l. La seconda contenente: 2,5 l di tampone di reazione (vedi Appendice Soluzioni e terreni") e 0,75 l di Taq Polimerasi (vedi Appendice Soluzioni e terreni") e acqua fino ad un volume di 11,5 l. . Viene aliquotata la prima miscela in un tubo di reazione, viene aggiunto il DNA, indi vengono aggiunti 25 l di olio minerale (Sigma) sterile. Il tubo viene posto poi nel blocchetto del termociclatore Perkin Elmer Cetus 9600 (Emeryville, CA). Quando la temperatura raggiunge i 94 C si aggiungono 12,5 l della seconda miscela di reazione (Chow et al., 92). Le reazioni sono soggette ad un primo ciclo di denaturazione di 5 minuti a 94C e successivamente a 30 cicli di denaturazione a 94C per 10 secondi, di appaiamento dei primer sulle regioni omologhe con un passaggio di temperatura da 70C a 64C (2 cicli a 70C, 2 cicli a 68C, 2 cicli a 66C e 30 cicli a 64C) per 30 secondi, alI'estensione a 64C per 1 minuto. Le reazioni sono completate con un'incubazione a 68C per 5 minuti. I prodotti di amplificazione sono controllati dopo l'amplificazione caricando 10 l delle reazioni su un gel di agaroso (Sea Kem) al 2%, mediante separazione elettroforetica e acquisizione dell'immagine mediante il Gel Doc 1000 Molecular Analyst (Bio-Rad).
85
II
2.9. - "SOLUZIONI E TERRENI" 2.9.1. - Coltura degli isolati fungini ldrolizzato di caseina (HC): Pesare 1 g estratto di lievito (Merck), 2 g di idrolizzato di caseina, 1,5 g di fosfato di potassio monobasico (Merck), 1 g di solfato di magnesio (Sigma), 15 g di glucosio (Merck), 10 ml di A-Z soluzione. Solubilizzare le polveri in 1 litro di acqua distillata in un becher agitando con un ancoretta magnetica, su un agitatore magnetico. Aggiungere un volume di circa 70 ml in beute da 200 mI. Autoclavare, per 10 minuti a 121C a due atmosfere di pressione, la beute tappate con tappi di cotone. Soluzione elementi A-Z in tracce: Disciogliere in 3 litri di acqua distillata: 0,93 g di cloruro di litio (Merck), 0,166 g di solfato di rame pentaidrato (Sigma), 0,166 g di solfato di zinco, 1,83 g di acido borico (Sigma), 0,166 g di solfato di alluminio, 0,083 g di cloruro di stagno diidrato, 1,17 g di cloruro di manganese tetraidrato, 0,166 g di solfato di nichel esaidrato, 0,166 g di nitrato di cobalto esaidrato, 0,166 g di biossido di titanio, 0,083 g di ioduro di potassio (Sigma), 0,083 g di bromuro di potassio (Sigma) . Potato dextrose agar (PDA, Merck): Sciogliere 39 g di polvere in un litro di acqua distillata. Sterilizzare in autoclave, per 10 minuti a 121C a due atmosfere di pressione, ed aliquotare con dosatore sterile, 9 mI per capsula Petri. Azoto liquido: fornito da AIR LIQUIDE ltalia Srl-Grugliasco-Torino
86
II
2.9.2. - Estrazione di DNA genomico 2.9.2.a. - Estrazione di DNA genomico da micelio liofilizzato Soluzione di lisi: 50 mM EDTA, 0,2% sodio dodecil solfato (Merck), 100 g/ml di proteinasi K. EDTA 0,5 M: Sciogliere in un becher 186,1 g di disodioetilendiaminoacetato diidrato (Sigma) in 800 mI di acqua distillata, agitare con ancoretta magnetica e rettificare il pH a 8-8.5 con pastiglie di idrossido di sodio (BDH); portare al volume di 1 litro. Dispensare in aliquote di 50 ml e sterilizzare in autoclave. SDS 10%: Sciogliere in un becher 100 g di SDS (Merck) in 900 mI di acqua distillata sterile, riscaldando a 68C. Rettificare il pH a 7,2 con qualche goccia di acido cloridrico fumante (Merck) e portare il volume a 1 litro; Dispensare in aliquote e conservare a temperatura ambiente. Proteinasi K: Sciogliere 100 mg di proteinasi K (Roche) in 10 ml di acqua distillata sterile; dispensare in aliquote da 1 ml (10 mg/ml). Potassio acetato 5M: Sciogliere, in un becker, 490,66 g di potassio acetato (Merck) in 1 litro di acqua milliQ; Facilitare la dissoluzione con l'aiuto di un'ancoretta magnetica, su un agitatore magnetico. Sterilizzare in autoclave. Fenolo cloroformio isoamilalcool (Sigma): disponibile nella formulazione 25:24:1. Ammonio acetato 7,5 M:
87
II
Sciogliere, in un becker, 578,1 g di ammonio acetato (Merck) in 1 litro di acqua distillata; facilitare la dissoluzione con l'aiuto di un'ancoretta magnetica, su un agitatore magnetico. Sterilizzare mediante filtrazione, con membrana porosa 0.22 m (Millipore). lsopropanolo: (Merck) Etanolo 96%:(Merck) Etanolo 75%: Preparare una soluzione con 75 ml di etanolo 96% (Merck) e 25 ml di acqua milliQ sterile. TE: Preparare una soluzione acquosa contenente 10 mM trisidrossimetilaminometano (Merck) e 1 mM EDTA pH 8 (Sigma). 2.9.2.b Metodo rapido di estrazione del DNA per PCR Soluzione L: Preparare una soluzione acquosa contenente 0,1 mM EDTA (Sigma), 10 mM Tris-HCI pH 8. Tris 1M: Sciogliere, in un becker, 121,1 g Tris base (Bio Rad) in 1 litro di acqua milliQ sterile; facilitare la dissoluzione con l'aiuto di un'ancoretta magnetica, su un agitatore magnetico, rettificando il pH con acido cloridrico fumante (Merck).
88
II
2.9.3. - Southern blot 2.9.3.a. - Trasferimento su membrana dei DNA genomici digeriti. Soluzione di depurinazione: Preparare una soluzione 0,25 M di acido cloridrico fumante (Merck). Soluzione di denaturazione: Preparare una soluzione 0,5 M di idrossido di sodio (BDH), 1,5 M di cloruro di sodio (Merck). Soluzione di neutralizzazione: Preparare una soluzione 1,5 M di cloruro di sodio (Merck), 0,5 M di Tris-HCI pH 7.5, e 1 mM EDTA (Sigma). SSC 20X: Preparare una soluzione sciogliendo 175,3 g/I di cloruro di sodio (Merck), 88,2 g/I di sodio citrato (Merck) in acqua milliQ sterile; portare a pH 7 con qualche goccia di acido cloridrico fumante (Merck).Dispensare in aliquote e autoclavare. SSC 2X Preparare una soluzione diluita 10 volte della soluzione madre SSC 20X 2.9.3.b Marcatura delle sonde a DNA con digossigenina. DIG DNA Labeling Kit (Roche): Sistema costituito da: esanucleotidi, dNTPs, enzima Klenow 2 U/l 2.9.3.c. - Controllo del DNA marcato Il kit di controllo DIG DNA Labeling Kit (Roche) contiene: DNA di controllo: marcato con digossigenina (20ng/l)
89
II
Tampone di diluizione: contenente 50g/ml di DNA di sperma di aringa in 10mM Tris-HCl e 1mM EDTA a pH 8. Anti-DIG-AP conjugate: Frammenti Fab di anticorpi policlonale prodotti in pecora (150 U/l), coniugati con lenzima fosfatasi alcalina. Blocking reagent (BR) 10%: Sciogliere 50 g di BR in 500 ml di MBS 1X Blocking reagent (BR) 1%: Preparare una soluzione diluita 10 volte in MBS 1X di BR 10%; filtrare su carta bibula. MBS 1X: Preparare una soluzione 0,1 M acido maleico (Sigma), 0,15 M cloruro di sodio (Merck); rettificare con idrossido di sodio (BDH) a pH 7.5. Tampone AP: Preparare una soluzione 0,1 M di Tris-HCI pH 9,5 e 0,1 M di cloruro di sodio (Merck) in acqua milliQ sterile. Lumigeno: Preparare una soluzione acquosa con CSPD 25 mM (Roche). Soluzione di sviluppo: GBX Developer (Sigma) concentrato da diluire in acqua milliQ (103 mI in 370 mI di acqua).
90
II
Soluzione di fissaggio: GBX Fixer (Sigma) concentrato da diluire in acqua milliQ. 2.9.3.d. - Preibridazione e ibridazione Liquido di preibridazione: Preparare una soluzione con blocking reagent 1%, 0,1% N-laurylsarcosine (Sigma), 2% sodium dodecil solfato (Merck). 2.9.3.e. - Sviluppo chemioluminescenza Soluzione 1: Preparare una soluzione 2X SSC, 0,1% sodio dodecil solfato (Merck). Soluzione 2: Preparare una soluzione 0,1% di SSC, 0,1% sodio dodecil solfato (Merck). Soluzione di lavaggio: Preparare una soluzione 0,3% Tween-20 (Merck) in MBS 1X. Per il Blocking reagent 1%,lanticorpo, lAP buffer, il lumigeno, e le soluzioni di sviluppo (vedi il paragrafo: Controllo del DNA marcato). 2.9.4. - Reazione di IPCR 2.9.4.a. - Digestione del DNA genomico e precipitazione Enzima utilizzato: BglII: 10U/ml (BioLabs) Sodio acetato 3 M: Sciogliere, in un becker 408,1 g sodio acetato triidrato (Merck) in 800 mI di acqua milliQ, rettificando il pH a 5,2 con acido acetico glaciale (Merck) e portando a volume di 1 litro. Dispensare in aliquote e autoclavare.
91
II
Etanolo 96% (Merck): Conservato alla temperatura di 20C. Etanolo 75%: Preparare una soluzione con 75 ml di etanolo 96% (Merck) e 25 ml di acqua milliQ sterile. Conservare a 20C. 2.9.4.b. - Autocircolarizzazione del DNA digeriti Tampone di ligazione: 66 mM Tris-HCI pH 7.5, 5 mM cloruro di magnesio (Carlo Erba), 1 mM ditiotritolo (Sigma), 1 mM ATP (Roche). Cloruro di magnesio 1 M: Sciogliere, in un becher, 203,3 g di cloruro di magnesio esaidrato (Carlo Erba) in 1 litro di acqua milliQ; sterilizzare in autoclave. Ditiotritolo (DTT) 1 M: Sciogliere 3,09 g di DDT in 20 mI di 0,01M di sodio acetato (Merck) a pH 5,2; sterilizzare per filtrazione, dispensare in aliquote da 1 mI e conservare a 20C. ATP 0,1 M: Sciogliere 60 mg di ATP (Roche) in 0,8 mI di acqua milliQ sterile, portando a pH 7 con 0,1 M di idrossido di sodio (BDH); portare al volume finale di 1 mI con acqua milliQ sterile. Dispensare in aliquote e conservare a -70C. Ligasi T4: 1 U/l (Roche)
92
II
2.9.4.c. - Reazione dl IPCR dNTP: predisposti dal kit: Expand Long Template PCR System (Roche, Mannheim), usati alla concentrazione di 350 M per ogni nucleotide. Oligonucleotidi IMP1,IMP2, IMP5, IMP6 (Life technologies): usare 300 nm di ogni sequenza primer. TamponeTaq: (buffer 3) tampone predisposto dal kit Expand Long Template PCR System (Roche, Mannheim), con 22.5 nM MgCl2 Taq: miscela enzimatica contenente un enzima Taq termostabile e la DNA polimerasi Pwo: usare 0,75 l per o gni reazione di amplificazione. 2.9.5. - Clonaggio dei prodotti di PCR 2.9.5.a. - Separazione elettroforetica del DNA Gel agaroso (Sea Kem) 0.8 %: Sciogliere 0.4 g di agaroso in 50 ml di tampone TAE 1X TAE 50X: Sciogliere 242g Tris (Bio Rad), in acqua milliQ sterile, aggiungere 57,1 mI di acido acetico glaciale (Merck), 100 mI di 1,5 M EDTA (Sigma) pH 8; portare ad un volume finale di 1 litro di acqua distillata e autoclavare. Marcatore del peso molecolare (Gibco BRL): Sono caricati su gel 10 l del marcatore avente concentrazione 1 g/l: la banda a 1.6 kb quantificata dalla ditta 100 ng (10% della massa totale).
93
II
Tampone di carica TIPO Il: Preparare una soluzione contenente 0.25% blu di bromofenolo (Sigma), 0,25% xilene cianolo, 15% Ficoll tipo 100, in acqua milliQ sterile. Conservare a temperatura ambiente. 2.9.5.b. - Reazione di ligazione del DNA nel vettore Tampone ligasi:. 10X concentrato (Roche). Ligasi T4: 1U/ l (Roche) 2.9.5.c. - Trasformazione LBA (Luria-Bertani addizionato di agar): Sciogliere in una becher 10 g/I di triptone (Difco), 5 g/I di estratto di lievito (Merck), 10 g/I di cloruro di sodio (Merck), aggiungere 10 mM di cloruro di magnesio (Carlo Erba); portare a pH 7.5 aggiungendo idrossido di sodio (BDH) 10 N; versare un volume di di 500 ml in una bottiglia da 1 litro e aggiungere 15 g/I agar (Merck). Autoclavare. Attendere che il substrato, ancora liquido si sia raffreddato, aggiungere eventuali molecole termolabili (ampicillina, IPTG, XGAL, etc.) e aliquotare in capsule petri. Ampicillina sale sodico Preparare una soluzione, in acqua sterile milliQ, alla concentrazione di 100 mg/ml (Merck) lavorando in ambiente sterile. lsopropiltiogalactoside (IPTG): Preparare una soluzione, in acqua sterile milliQ, alla concentrazione di 100 mM di IPTG (BioRad) .
94
II
5-bromo 4-cloro-3-indolil-galattoside (Xgal): Preparare una soluzione, in acqua sterile milliQ, alla concentrazione di 50 mg/ml di X-GAL (Promega) 2.9.5.d. - Protocollo per lestrazione di DNA plasmidico da cellule batteriche trasformate Soluzione R: Preparare una soluzione 20 mM Tris-HCI pH 8, 5, 1M NaEDTA (Sigma) pH 8, 10g/ml RNAsi (Roche). Soluzione L: Preparare una soluzione all1% di SDS (Merck) e 0,2 M di idrossido di sodio (BDH). Soluzione N: Preparare una soluzione 4 M cloruro di sodio (Merck) e 100mM acido acetico (Merck). 2.9.5.e. - Protocollo di estrazione di DNA plasmidico da cellule batteriche su larga scala (MIDI preparation) Tampone S1: Preparare una soluzione 50 mM Tris-HCI pH 8, 10 mM EDTA (Sigma), 100 g/ml RNAsiA (Roche) . Tampone S2: Preparare una soluzione 200 mM idrossido di sodio (BDH), 1% SDS (Merck). Tampone S3: Preparare una soluzione 2,8 M potassio acetato (Merck) pH 5,2 .
95
II
Tampone N2: Preparare una soluzione 100 mM Tris (Bio Rad), 15% etanolo (Merck), 900 mM cloruro di potassio (Carlo Erba), rettificato con acido fosforico (Merck) a pH 6,3. Tampone N3: Preparare una soluzione 100 mM Tris (Bio Rad), 15% etanolo (Merck), 1150 mM cloruro di potassio (Carlo Erba), rettificato con acido fosforico (Merck) a pH 6,3 . Tampone N5: Preparare una soluzione 100 mM Tris (Bio Rad), 15% etanolo (Merck), 1000 mM cloruro di potassio (Carlo Erba), rettificato con acido fosforico (Merck) a pH 8,5 2.9.6. - Reazione di PCR Tampone: 10 mM Tris-HCI pH 8,8, 1,5 mM cloruro di magnesio (Carlo Erba), 50 mM cloruro di potassio (Carlo Erba), 0,1% Triton X-100 (Merck), 0.01% (w/v) gelatina. dNTP (Finzymes): rispettivamente 200 mM di dATP,dCTP, dGTP, dCTP. Oligonucleotidi: 0,05 mM di ciascuno. Taq polimerasi: preparata seguendo il protocollo di Desai e Pfaffle (1995).
96
II
2.10. - Reazioni di RAPD PCR Tampone: 10 mM Tris-HCI pH 8,8, 1,5 mM cloruro di magnesio (Carlo Erba), 50 mM cloruro di potassio (Carlo Erba), 0,1% Triton X-100 (Merck), 0,01% (w/v) gelatina. dNTP (Finzymes): rispettivamente 200 mM di dATP,dCTP, dGTP, dCTP. Oligonucleotidi: 0,05 mM di ciascuno.
97
III
CAPITOLO 3
1. - RISULTATI 1.1. - Analisi dei profili RAPD-PCR Per valutare il polimorfismo dei Fusaria antagonisti sono stati fatte 25 analisi RAPD, utilizzando 25 primer (Tabella 3). Sono stati ottenuti degli amplificati contenenti da uno a cinque frammenti; le dimensioni delle bande, variabili da isolato a isolato, sono comprese tra 0.3 Kb e 3 Kb. I diversi isolati di Fusarium antagonisti sono stati confrontati con diverse formae speciales di Fusarium oxysporum (Tabella 1). Per lanalisi dei dati stato usato il programma Statistica (Starsoft); si scelto come algoritmo, per costruire il dendrogramma, lUPGMA (unweighted pairgroup method using arithmetic averages). Le distanze tra i diversi raggruppamenti sono state valutate in distanze euclidee per evidenziare meglio le differenze. Lanalisi ha evidenziato una sostanziale differenza tra le forme speciali e tra queste ultime e gli antagonisti. Si sono riscontrate similitudini tra gli isolati 233/2, 245 wt e MSA 32 che formano un gruppo a se' stante rispetto alle forme speciali e agli isolati 152, 257 e 251, e MSA 35 (Figure 1A e 1B). 1.2. - Analisi dei polimorfismi dell' elemento trasponibile impala Per analizzare il polimorfismo del trasposone impala stato eseguito un Southern blot dei medesimi isolati utilizzati per lanalisi RAPD (Tabella 1). Il DNA genomico estratto da ciascun campione stato digerito con lenzima di restrizione BglII, che non riconosce siti di taglio nella sequenza di impala. Tali frammenti sono stati separati tramite elettroforesi, trasferiti su una membrana di nylon e quindi fissati tramite irraggiamento con raggi UV. La membrana stata ibridata con le sonde impala R1 e impala 245. impala R1 stata ottenuta amplificando il trasposone da un isolato di Fusarium oxysporum f. sp. dianthi razza 1, mediante PCR e successiva marcatura con digossigenina mentre
98
III
impala 245 stata ottenuta dallisolato 245 wt con le medesime procedure. Lanalisi con le due diverse sonde ha evidenziato dei profili diversi: il profilo ottenuto con la sonda impala 245 poco polimorfico, ma sovrapponibile interamente a quello, altamente polimorfico, ottenuto con la sonda impala R1 (figure 2A e 2B) Il Southern blot ha permesso di evidenziare dei polimorfismi di inserzione dellelemento trasponibile impala. Utilizzando impala R1 si ottengono profili delle forme speciali molto diversi tra loro e polimorfici rispetto agli antagonisti in generale. Utilizzando la sonda impala 245 si ottiene, in tutti gli isolati, eccetto che in 245 wt, 233/2 e MSA32, un doppietto delle dimensione di 3.6 kb e 4 kb (Figura 2A). Con entrambe le sonde gli isolati 233/2, 245 wt e presentano un profilo unico analogo formato da inserzioni del trasposone a 2.8 kb, a 3.3 kb, un doppietto a 4.3-4.5 kb e a 12 kb mentre MSA 32 ha lo stesso profilo eccetto per la banda a 3.3 kb (corsia 14, 15, 18, Figure 2A e 2B). 1.3. - Isolamento delle regioni fiancheggianti (giunzioni) del trasposone impala nellisolato 233/2 Per isolare le giunzione del trasposone ci si avvalsi della tecnica della PCR inversa (Chiocchetti et al., 1999). Con i primer IMP1 e IMP 2 stato ottenuto un amplimero di circa 3.8 kb, corrispondente all'inserzione a 4.3 kb sul Southern blot. Si risale alla dimensione della banda sul Southern aggiungendo 567 bp al frammento di IPCR ottenuto, dal momento che i primer cosi disegnati (Figura 3) su impala, escludono una zona di 567 bp. E stata provata sul primo ciclo di amplificazione con IMP1 e IMP2 un secondo ciclo con i primer IMP5 e IMP6 disegnati pi esternamente (Figura 3). Questa PCR annidata non ha mai dato origine ad alcun amplificato quando si partiva da un DNA stampo derivato dal primo ciclo. Al contrario si ottenuto un prodotto di 2.2 kb quando sono stati utilizzati direttamente IMP5 e IMP6 sul prodotto di ligazione. Tale frammento corrisponde ad una banda sul Southern blot di 3.2 kb. In realt la sequenza di questo prodotto di PCR ha evidenziato un
99
III
trasposone tronco alle estremit; Lamplimero a 2.2 kb dovrebbe corrispondere sul Southern blot alla banda a 2.8 kb. I prodotti di PCR inversa sono stati chiamati ACQ1 (3.8 kb) e ACQ2 (2.2kb) 1.4. - Sequenziamento dei cloni ACQ1 e ACQ2 Sono state ottenute le sequenze parziali (circa 500 bp per lato) delle regioni fiancheggianti impala, identificate nei cloni ACQ1 e ACQ2. Di seguito vengono riportate le sequenze: ACQ1 sp6
AATCCTATAGAGAATCTGTGGGCGTTGATGAAGGCAGAGATCTGCGACTCGCTGAGGTTCTCTATCTTGTAACGTT ATAGCGTTGTTAACGAGAGCAGGCCAGCGGCTGAGCCATTTATCGAGACTGGTGTTGCTAAGGCCCTTCTTAAGCT TTTCAAACTCAGTCCTAACATTCTGCTTCTGGTCTTTCGCGGTGGGTTGGATGCTATCAGCCAGCGCGATGAGTTAC CGCGGACGTCATGGATTCCGAGGTAGTGCTGCTTGAACTCCCTGACACACTGTGAGGTCAATGCGATCAGAGACC TTGTCGTAT
ACQ1 T7
GCGGATCGGTTATGACGGTATCATAAATCTGATCCCGTTGCTCTTCTGTAAGCTTACGTGGCGCGCCTGATCGCGG CAGGGATATGTTATCGGCTCCACGTTGTGCCTCGCGGCGTAGGGTCGTTATTGATCGTGCCGAGAGGGATATCAG GGAAATGCTTCTTGATCTGTGAGTGCGTAAGCCCTTGCCTACGTAATTCACATATACGAGACCGTAATGACGGCGA TAATTCCTTGCCTCGAGGCATCGTCATGGTGTNGTGATGTGTGTTGTGGGGAACAGGGGCTGCCGTCAGATCAATC AAATCAGATTAGGGCGTAAATCTGAACTGAACTGATACTTTACCCTCTGCATCTGATCTGATTGATTGATTGATAGAT ATCTGATATATAATAGGTCACGTGGAGCTATNCCCAGCCCTCACGATTCTGCCCCGTGGGATCTGACTGCCCTTGT TNGCTTAAGCNTACGC
ACQ2 sp6
GTGAGACTATGCCGAACCGTGTTACTGCTGTAATTACTGCAGAAGGTTGGTATACAAAGTACTGAAGCTGTTCTAAT TGACCTTGTTAGGTCATAAATTACAATATCACAGCAGCAGCATAACAGCTGCCCGTTTCGGCTAGCCTCGATCGTC GGTACCTCTTGGTGTCTTCAAACTTATTGCACCCCACTGTATGTCCATCGTATTCGCCTTGAGCTTAGGTTAAGACG GAGAGGCAAGCCGGTCTCGGCTTGCGCATAGAGCCTTAGAGGAACGAAGTTTAGAGGTCGGAGGCAGATGGGTT GCACGTTCTCTAGAGTAAAGTGGCATTTCCNTTTTATAGCTAGATAATACGGCTAATTTGGTCNGGGTTNACTNTTTT CCCACNCTGAGANNTATTTGCGGACTGATAA
ACQ2-T7
TTGTGTTGTGTGTGTGGAGGAGGAAGAAAGAGCGGCATCGAAACGCGGTCCCGCTCGGGTTAGGCCGGACCGAG ATACCTGCAGATGTATTCAAACTTATTGCACCCCACTGTATTACGATATATCCTGTAGCACGTGCCGAGAAATGTCA GTGTCCACGGATAAGCGGCACAATATGACCTTACGTCATTTTCCTACGACCCCAATAATATCCCGAAACTCTGTTAC CCGGCTTATGAACACAAAGTATCGGTACCGCTTCCGGTCGTCGGGTGAACGGCTTCCGAACGGCTTCAAGAACGG
100
III
CTTTACTGATCCGAAAGTGTGTCGTAGTGTGTGTATACAGTATTGAGCAAGCAGCGATGCTTATAAATACTGACAAA TCATATGCTTACCANGTAGAGCCATGTACCCNAAAGGTCTCCACCCCTAGACTTTACAAAAGAGCATG
1.5. - Amplificazione dei ceppi antagonisti 233/2 e 245 wt Per ottenere degli amplimeri corrispondenti ad ogni sequenza fiancheggiante clonata, sono state eseguite quattro diverse reazioni di PCR con quattro coppie di primer. Ogni reazione prevedeva luso di un oligonucleotide disegnato sulla sequenza fiancheggiante e laltro su un estremit del trasposone impala (Figura 3). Le reazioni sono state eseguite analizzando gli antagonisti e le diverse forme speciali di F. oxysporum. Come stampo stato utilizzato DNA genomico estratto da micelio liofilizzato. Le coppie A2I/IMP5 e A2F/IMP6 hanno dato lamplificazione, rispettivamente, di un amplimero di 313 bp e di 305 bp in tutti gli antagonisti, seppure con intensit diverse e amplificati di diversa taglia molecolare per le forme speciali. La coppia A1I/IMP1 ha dato un amplimero di 439 bp nei ceppi 233/2, 245 wt. MSA32, MSA35 e dei prodotti a diverse taglie molecolari per gli altri ceppi. La coppia A1F/IMP2A non ha dato amplimeri.
2. - DISCUSSIONE I ceppi antagonisti di Fusarium, isolati da terreni repressivi liguri rappresentano un valido strumento di lotta biologica alle malattie causate da F. oxysporum patogeni. La loro immissione nellambiente andrebbe controllata in tempi diversi per evidenziare lentit della popolazione, la sopravvivenza nel terreno e la dispersione nel suolo. Le strade perseguite in passato sono state la mutagenesi, la fusione di protoplasti e la trasformazione genetica. Studi di valutazione dellimpatto ambientale riguardanti ceppi di Fusarium dotati di capacit antagonistica erano gi stati compiuti utilizzando ceppi opportunamente trasformati con il gene per la resistenza alligromicina (Migheli et al.,1996), mutanti colorati ottenuti per mutagenesi e ceppi ibridi derivati dalla fusione di protoplasti (Gullino et al.,1995).
101
III
Nel corso di questa tesi, si cercato di sviluppare un mezzo diagnostico esente da restrizioni di tipo normativo. Lobiettivo del lavoro era quello di sviluppare dei marcatori molecolari, utilizzando i trasposoni naturalmente presenti nel genoma, al fine di etichettare gli isolati 233/2 e 245 wt senza ricorrere a sistemi di trasformazione genica n di mutagenesi. La modificazione genetica avrebbe fatto rientrare i ceppi antagonisti 233/2 e 245 wt nel gruppo degli OGM e ci avrebbe, di fatto, rallentato il processo di registrazione a causa del grande numero di prove richiesto dalla direttiva 90/219. La mutagenesi avrebbe invece potuto pregiudicare la funzionalit dei funghi come agenti di contenimento biologico e creare della variabilit non prevedibile a priori. A tale scopo stato studiato, prima, il profilo RAPD degli antagonisti per ampliare i dati sul polimorfismo presente tra gli antagonisti e la distribuzione dellelemento trasponibile impala nel genoma degli isolati 233/2 e 245 wt confrontandolo con altri isolati di Fusarium saprofiti e con diverse forme speciali. Dallanalisi mediante Southern blot emersa una grande diversit di distribuzione di impala tra 233/2 e 245wt rispetto a tutti gli altri isolati patogeni e saprofiti. Lisolato MSA32 ha mostrato un profilo simile ma non identico a 233/2 e 245 wt, mancando della banda a 3.3 kb e la banda alta del doppietto 4.3-4.5 kb. Ipotizzando che la giunzione di una copia di impala inserita in una posizione unica nel genoma sia anchessa unica, si pensato di poterla amplificare mediante una reazione di PCR che utilizzasse un primer disegnato sul trasposone, diretto verso lesterno e un primer disegnato sulla regione fiancheggiante diretto verso il trasposone (Figura 3). Per rendere pi semplice il clonaggio delle regioni fiancheggianti Fot1 o impala, senza bisogno di costruire una libreria genomica per gli isolati di F. oxysporum 233/2 e 245 wt, stata adottata la tecnica di PCR inversa (IPCR; Ochman et al., 1988; Silver e Keerikatte, 1989; Triglia et al., 1988). Essa prevede una preliminare digestione del DNA con un enzima che non tagli allinterno della sequenza desiderata, una auto-ligazione dei frammenti e lamplificazione mediante PCR utilizzando due primer inversi che fossero disegnati orientati verso lesterno delle sequenza di appaiamento.
102
III
Tale amplificazione d origine potenzialmente ad un insieme di amplimeri pari al numero di inserzioni del trasposone nel genoma. Esiste per un limite tecnico rappresentato dalla capacit della Taq polimerasi di generare frammenti non troppo lunghi. Lenzima utilizzato nella IPCR era in realt una miscela di Taq polimerasi e Pwo DNA polimerasi che garantiva caratteristiche di efficienza e fedelt molto elevate. Sono stati ottenuti amplimeri di 3.8 kb e 2.2 kb corrispondenti a inserzioni specifiche nel genoma di 233/2 e 245 wt. Lutilizzo di elementi trasponibili come marcatori molecolari ha in se' il pericolo dello salto del trasposone e come conseguenza la perdita del marcatore nella posizione originale; ci potrebbe portare a dati non costanti nel tempo e quindi non pi attendibili. Da analisi mediante Southern blot, ripetute in tempi diversi, su isolati patogeni e saprofiti di F. oxysporum stata riscontrata una pressoch nulla mobilit dellelemento impala. Per ovviare, comunque, a questo potenziale inconveniente possibile studiare le copie del trasposone, troncate allestremit e quindi prive delle sequenze ITR, necessarie per la trasposizione (Fernandez et al., 1999). Nonostante le sequenze clonate coincidessero le giunzioni caratteristiche degli isolati 233/2 e 245 wt gli esperimenti di PCR messi a punto per amplificare le giunzioni hanno mostrato una assenza di specificit assoluta. Le amplificazioni hanno fornito amplimeri delle dimensioni attese in 233/2 e 245 wt ma contemporaneamente anche in altri isolati e amplimeri di taglie molecolari diverse in Fusarium oxysporum patogeni (Figura 4). Lanalisi mediante Southern blot, compiuta utilizzando come sonde gli amplimeri derivati dallIPCR su impala, ha dato origine a profili di ibridazione molto complessi, dimostrando che questo elemento inserito in regioni ripetute nel genoma di F. oxysporum. Questo dato era gi stato messo in evidenza per i differenti patotipi di F. oxysporum f.sp. dianthi (Chiocchetti et al.,1999). Dati ottenuti, con lo stesso approccio, su un nuovo isolato di Fusarium agente di tracheofusariosi nella margherita confermano tale tendenza anche per il trasposone Fot1 (Pasquali et al., in preparazione). La tendenza a trasporsi di questi elementi trasponibili in zone ripetute e quindi non attive dal punto di vista trascrizionale, potrebbe essere spiegata
103
III
ipotizzando che un salto in tali regioni genomiche non apporti alcuna mutazione letale o selezionante in senso negativo rendendo levento di trasposizione compatibile con la sopravvivenza del fungo. Alcune copie potrebbero essersi accumulate in tali siti genomici e rappresentare una fonte di variabilit di cui tali funghi dispongono per ladattamento nellambiente. Si potrebbe dimostrare linserzione preferenziale in regioni ripetute mediante lisolamento di tutte le regioni fiancheggianti e con lanalisi di regioni fiancheggianti pi estese. Libridazione con sonde ricavate da regioni fiancheggianti su cariotipi ottenuti mediante elettroforesi su campo pulsato e libridazione in situ su cromosomi purificati potrebbe fare comprendere la localizzazione preferenziale di questi elementi trasponibili nei diversi cromosomi.
3. - CONCLUSIONI La tecnica della PCR inversa, che fino ad oggi ha dato risultati abbastanza soddisfacenti, non si dimostrata applicabile, in modo efficace, al campo dei funghi antagonisti. Non si riuscito, infatti, a discernere tra i diversi isolati di Fusarium antagonisti in maniera univoca. Cio' deve portare a considerare questo approccio in modo pi realistico e a concludere che la tecnica della PCR inversa potenzialmente utile, seppure non deve essere sopravvalutata, e che lutilizzo di tale metodo applicato ai trasposoni presenta dei pericoli intrinseci legati alla distribuzione e localizzazione degli elementi trasponibili nei diversi genomi.
104
IV
BIBLIOGRAFIA Adams P. B. (1990). The potential of mycoparasites for biological control of Plant Diseases. Annual Review of Phytopathology, 28: 59-72. Adouvette-Panvier A., Davies J. E., Grits L. R. e Littlewood B. A. (1980). Studies of ribosomal proteins of yeast species and their hybrids by gel electrophoresis and immunochemical cross-reaction. Molecular and General Genetics, 179: 273-282. Alabouvette C., Tramier R., Grouet D. (1980). Recherches sur la rsistance des sols aux maladies. VIIII. Perspectives dutilization de la rsistance des sols pour lutter contre les fusarioses vasculaires. Annales de Phytopathologie, 12: 83-93. Alberts B., Bray D., Lewis J., Raff M., Roberts K. e Watson J. D. (1995). Biologia molecolare della cellula, 3a Edizione. Zanichelli, Bologna. Altieri M. A., Letourneau D. K. (1982). Vegetation menagement and biological control in agroecosystems. Crop Protection, 1: 405-430. Appel D. J. e Gordon T. R. (1996). Relationship among pathogenic and nonpathogenic isolates of Fusarium oxysporum based on the partial sequence of the intergenic spacer region of the ribosomal DNA. Molecular Plant-Microbe Interactions, 9: 125-138. Armstrong G. M. e Armstrong J. K. (1981). Formae speciales and races of Fusarium oxysporum causing wilt diseases. In: "Fusarium: Diseases, Biology and Taxonomy", Nelson, P.E., Toussoun, T.A. e Cook, R.J. (coord.), The Pennsylvania University Press, University Park, pp.: 391-399. Assigbetse K. B., Fernandez D., Dubois M. P. e Geiger J. P. (1994). Differentiation of Fusarium oxysporum f. sp. vasinfectum races on cotton by random amplified polymorphic DNA (RAPD) analysis. Phytopathology, 84: 622626. Avery O. T., MacLeod C. M., McCarthy M. (1944). Studies on the chemical Nature, of the subtance inducing transformation of pneumococcal types. Journal of Experimental Medicine, 79: 137-158. Ayers W. A. e Adams P. B. (1983). Improved media for growth and sporulation of Sporidesmium sclerotivorum. Canadian Journal of Microbiology, 29: 325-330.
105
IV
Baayen R. P., Van Dreven F., Krijger M. C. e Waalwijk C. (1997). Genetic diversity in Fusarium oxysporum f. sp. dianthi and Fusarium redolens f. sp. dianthi. European Journal of Plant Pathology, 103: 395-408. Baidyaroy D., Huber D. H., Fulbright D. W. e Bertrand H. (2000). Transmissible mitochondrial hypovirulence in a natural population of Cryphonectria parasitica. Molecular Plant-Microbe Interactions, 13(1): 88-95. Baker K. F. e Cook R. J. (1974). Biological control of plant pathogens. Freeman & Co. San Francisco, pp. 433. Barker I. e Pitt D. (1987). Detection of the leaf curl pathogen of anemones in corms by enzyme-linked immunosorbent assay (ELISA). Plant Pathology, 37: 417-422. Beckman C. H. (1966). Cell irritability and localization of vascular infection in plants. Phytopathology, 56: 821-824. Beckman C. M. (1969). The mechanics of gel formation by swelling of simulated plant cell wall membranes and perforation plates of banana root vessel. Phytopathology, 59: 827-843. Beckman C. M. e Zaroogian G. E. (1967). Origin and composition of vascular gel in banana roots. Phytopathology, 57: 11-13. Behrens U., Fedoroff N. V., Laird A., Muellerneumann M., Starlinger P e Yoder J. I. (1984). Cloning of Zea mays controlling element Ac from the wx-n7 allele. Molecular and General Genetics, 194: 346-347. Berg D. E., Howe M. M. (1989). Mobile DNA. American Society for Microbiology. Beyer W., Hoxter H., Miedaner T., Sander E. e Geiger H. H. (1993). Indirect ELISA for quantitative assessment of Fusarium spp. in rye. Journal of Plant Disease and Protection, 100: 278-284. Bilai V. I. (1955). The Fusaria (Biology and Systematics). Doklady Akademii Nauk Ukrainskoj SSR, Kiev. Biles C. L. e Martyn R. D. (1988). Isozyme analysis of Fusarium oxysporum f. sp. niveum races and selected Fusarium sp. (Abstract). Phytopathology, 78: 625.
106
IV
Bingham P. M. e Zachar Z. (1989). Retrotransposons and the FB transposon from Drosophila melanogaster. In Mobile DNA, Berg, D.E. e Howe, M.M. (coord.), American Society for Microbiology, Washington DC, pp. 485-502. Bok S. H., Lee H. W., Choi D. e Kim S. U. (1996). Bioencapsulated biopesticides, in T. Wenhua. R. J. Cook and Rovira (Eds). Advances in Biological Control of Plant Diseases. China Agricultural University Press, Beijing, pp.: 303-309. Bonnet G., Tyagi S., Libchaber A. e Kramer F. R. (1999). Thermodynamic basis of the enhanced specificity of structured DNA probes. Proceedings of the National Academy of Sciences USA, 96: 6171-6176. Booth C. (1971). The Genus Fusarium. Commonwealth Mycological Institute, Kew, Surrey, UK. Bosland P. W. e Williams P. H. (1987). An evaluation of Fusarium oxysporum from crucifers based on pathogenicity, isozyme polymorphism, vegetative compatibility, and geographic origin. Canadian Journal of Botany, 65: 20672073. Bragard H., Singer E., Alizadeh A., Vauterin L. e Maraite H. (1997). Xanthomonas translucens from small grains: diversity and phytopathological relevance. Phytopathology, 87: 1111-1117. Brandes E. W. (1919). Banana wilt. Phytopathology, 9: 339-390. Brayton P. R. e Colwel R. R. (1987). Fluorescent antibody staining method for enumeration of viable environmental Vibrio cholerae 01. Journal of Microbiological Methods, 6: 309-314. Bruns T. D., White T. J. e Taylor J. W. (1991). Fungal molecular systematics. Annual Review of Ecology and Systematics, 22: 525-564. Burdon J. J. e Marshall D. R. (1983). The use of isozymes in Plant Disease research. In: "Isozymes in plant genetics and breeding, Part A", Tanskey, S.D. e Orton, T.J. (coord.), Elsevier Science Publishers, Amsterdam, pp. 401-412. Bureau T. E. e Wessler S. R. (1992). Tourist: a large family of small inverted repeat elements frequently associated with maize genes. Plant Cell, 4: 12831294.
107
IV
Bureau T. E., Ronald P. C. e Wessler S. R. (1996). A computer-based systematic survey reveals the predominance of small inverted-repeat elements in the wild-type rice gene. Proceedings of the National Academy of Sciences USA 93: 8524-8529. Butcher A. C., Croft J. H. e Grindle M. (1972). Use of genotype environmental interaction analysis in the study of natural population of Aspergillus nidulans. Heredity, 29: 263-283. Caetano-Anolls G., Bassam B. J. e Gresshoff P. M. (1995). DNA amplification fingerprinting using very short arbitrary oligonucleotides primers. Bio/Technology, 9: 553-557. Calvi B. R., Hong T. J., Findley S. D. e Gelbart W. M. (1991). Evidence for a common evolutionary origin of inverted repeat transposons in Drosophila and plants: hobo, Activator and Tam3. Cell, 66: 465-471. Capy P., Bazin C., Higuet D. e Langin T. (1998). Dynamics and evolution of transposable elements. Molecular Biology Intelligence Unit. Springer-Verlag ed., Landes Bioscience. Heidelberg. Carle G. F. e Olson M. V. (1985). An electrophoretic karyotype for yeast. Proceedings of the National Academy of Sciences of USA, 82: 3756-3760. Carle G . F., Frank M. e Olson M. V. (1986). Electrophoretic separation of large DNA molecules by periodic inversion of the electric field. Science, 232: 65-68. Chen B., Nuss D. L. (1999). Infectious cDNA clone of hypovirus CHV1-Euro7: a comparative virology approach to investigate virus-mediated hypovirulence of the chestnut blight fungus Cryphonectria parasitica. Journal of Virology, 73(2): 985-992. Chet I. (1987). Trichoderma. Applications, mode of action and potential as a biocontrol agent of soil-borne pathogenic fungi. In: Innovative approaches to plant disease control (Chet, I. coord.). Wiley, New York, pp. 137-160. Chiocchetti A., Ghignone S., Minuto A., Gullino M. L., Garibaldi A., Migheli Q. (1999). Identification of Fusarium oxysporum f. sp. basilici isolated from soil, basil seed, and plants by RAPD analysis. Plant Disease, 83: 576-581.
108
IV
Chiocchetti A., Bernardo I., Daboussi M. J., Garibaldi A., Gullino M. L., Langin T. e Migheli Q. (1999). Detection of Fusarium oxysporum f. sp. dianthi in carnation tissue by PCR amplification of transposon insertions. Phytopathology, 89: 1169-1175. Chou Q., Russel M., Birch D. E., Raymond J. e Bloch W. (1992). Nucleic Acids Research, 20: 1717-1723. Chu G., Vollrath D. e Davis R. W. (1986). Separation of large DNA molecules by contour-clamped homogeneous electric field. Science, 234: 1582-1585. Chun S. C., Schneider R. W., Groth D. E. e Giles C. G. (1997). Effect of different carbon sources on efficacy of biological control of seedling disease in water-seeded rice. Phytopathology, 87: S19. Clare B. G. (1963). Starch-gel electrophoresis of proteins as an aid in identifying fungi. Nature, 200: 803-804. Clare B. G. e Zentmyer G. A. (1966). Starch gel electrophoresis of proteins from species of Phytophthora. Phytopathology, 59: 1334-1335. Clark C. A., Chen C., Ward-Rayner N. e Pettis G. S. (1998). Diversity within Streptomyces ipomoeae based on inhibitory interaction, rep-PCR, and plasmid profiles. Phytopathology, 88: 1179-1186. Clerc A., Manceau C. e Nesme X. (1998). Comparison of random amplified polymorphic DNA with amplified fragment length polymorphism to access genetic 1187. Connick W. J., Jr., Boyette C. D. e McAlpine J. R. (1991). Formulation of mycoherbicide using a pasta-like process. Biological Control, 1: 281-287. Connick W. J., Jr., Daigle D. J., Boyette C. D., Williams K. S. Vinyard B. T. e Quimby P. C., Jr. (1996). Water activity and other factors that affect the viability of Colletotrichum truncatum conidia in in wheat flour-kaolin granules (Pesta). Biocontrol Science and Technology, 6: 277-284. Cook R. J. (1981). The influence of rotation crop on take-all decline phenomenon. Phytopathology, 71: 189-192. diversity and genetic relatedness within genospecies III of Pseudomonas syringae. Applied and Environmental Microbiology, 64: 1180-
109
IV
Correll J. C. (1991). The relationship between formae speciales, races and vegetative compatibility groups in Fusarium oxysporum. Phytopathology, 81: 1061-1063. Correll J. C., Puhalla J. E., e Schneider R. W. (1986). Vegetative compatibility groups among nonpathogenic root-colonizing strains of Fusarium oxysporum. Canadian Journal of Botany, 64: 2358-2361. Correll J. C., Rhoads D. D. e Guerber J. C. (1993). Examination of mitochondrial DNA restriction fragments polymorphism, DNA fingerprints, and randomly Crowhurst amplified polymorphic DNA of Colletotrichum orbiculare. Phytopathology, 83: 1199-1204. R. N., Hawthorne B. T., Rikkerink H. A. e Templeton M. D. (1991). Differentiation of Fusarium solani f. sp. cucurbitae race 1 and 2 by random amplification of polymorphic DNA. Current Genetics, 20: 938-946. Daboussi M. J., Langin T. e Brygoo Y. (1992). Fot1, a new family of fungal transposable elements. Molecular and General Genetics, 232: 12-16. Deacon J. W. e Berry L. A. (1992). Modes of action of mycoparasites in relation to biocontrol of soilborne plant pathogens. In: Biological Control of Plant Diseases (E. C. Tjamos, G. C. Papavizas R. J. Cook, coord.). Plenum Press, New York, 157-167. Dean P. D. G., Johnson W. S. e Middle F. A. (1985). Affinity Chromatography: A practical Approach. IRL Press, Arlington, VA. DeBruijn F. J. (1992). Use of repetitive (repetitive extragenic palindromic and enterobacterial repetitive intergenic consensus). sequences and the polymerase chain reaction to fingerprint the genomes of Rhizobium meliloti isolates and other soil bacteria. Applied and Environmental Microbiology, 58: 2180-2187. Deese D. C. e Stahmann M. A. (1962). Pectic enzymes and cellulose formation by F. oxysporum f. cubense on steam tissues from resistant and susceptible banana plants. Phytopathology, 52: 247-255. Deese D. C. e Stahmann M. A. (1962). Pectic enzymes in Fusarium-infected susceptible and resistant tomato plants. Phytopathology, 52: 255-260.
110
IV
Deininger P. L. (1989). SINEs: Short Interspersed Repeated DNA elements in higher eucaryotes. In Mobile DNA Berg, D.E. e Howe, M.M. (coord.), American Society for Microbiology, Washington DC, pp. 619-636. Del Sorbo G., Scala F., Capparelli R., Iannelli D. e Noviello C. (1993). Differentiation between two formae speciales of Fusarium oxysporum by antisera produced in mice immunologically tolerized at birth through lactation. Phytopathology,83: 1178-1182. Delaunay J., Creusot F. e Schapira G. (1973). Evolution of ribosomal proteins. European Journal of Biology 39: 305-312. Desai U. J., Pfaffle P. K. (1995). Single step purification of a thermostable DNA polymerase expressed in Escherichia coli. BioTechniques, 19: 780-784 Dewey F. M. (1992). Detection of plant-invading fungi by monoclonal antibodies. In: "Techniques for rapid detection of plant pathogens", Duncan, J.M. e Torrance, L. (coord.), Blackwell Scientific Publications, Oxford, pp. 47-62. Dewey F. M. e Thornton C. R. (1995). Fungal immunodiagnostics in plant agriculture. In: "New diagnostics in crop science; biotechnology in agriculture, No. 13", Skerritt, J.H. e Appels, R. (coord.), CAB International, pp. 151-170. Dewey F. M. e Thornton C. R. (1995). Fungal immunodiagnostics in plant agriculture. In: "New diagnostics in crop science; biotechnology in agriculture, No. 13", Skerritt, J.H. e Appels, R. (coord.), CAB International, pp. 151-170. Dick M. (1998). Impact of molecular methods on fungal systematics. Mycologist 12: 116-117. Droby S. e Chalutz E. (1994). Successful biocontrol of postharvest pathogens of fruits and vegetables. Proc. Brighton Crop Protection Conference, 3: 12651272. Dubos B. (1997). Fungal antagonism in aerial agrobiocenoses. In: Innovative Approaches to Plant Disease Control, Chet, I. (coord.), Wiley & Sons, New York, 107-135. Edel V., Steinberg C., Avelange I., Laguerre G. e Alabouvette C. (1995). Comparison of three molecular methods for the characterization of Fusarium oxysporum strains. Phytopathology, 85: 579-585.
111
IV
Edel V., Steinberg C., Gautheron N. e Alabouvette C. (1996). Evaluation of restriction analysis of polymerase chain reaction (PCR)-amplified ribosomal DNA for the identification of Fusarium species. Mycological Research 101: 179187. Edel V., Steinberg C., Gautheron N. e Alabouvette C. (1997). Population of nonpathogenic Fusarium oxysporum associated with roots of four plant species compared to soilborne populations. Phytopathology, 87: 693-697. Elad Y., Chet I. e Katan J. (1980). Trichoderma harzianum: A biocontrol agent effective against Sclerotium rolfsii and Rhizoctonia solani. Phytopathology, 70: 119-121. Elad Y., e Hadar Y. (1981). Biological control of Rhizoctonia solani by Trichoderma harzianum in carnation. Plant Disease, 65: 675-677. Elias K. S. e Schneider R. W. (1991). Vegetative compatibility groups in Fusarium oxysporum f. sp. lycopersici. Phytopathology, 81: 159-162. Emmons S. W., Yesner L., Ruan K. S. e Katzenberg D. (1983). Evidence for a transposon in Caenorhabditis elegans. Cell, 32: 55-65. Engels W. (1989).P element in Drosophila melanogaster. In Mobile DNA , Berg, D.E. e Howe, M.M. (coord.), American Society for Microbiology, Washington, DC, pp. 437-484. Fayet O., Ramond P., Polard P., Prere M. F. e Chandler M. (1990). Functional similarities between retroviruses and the IS3 family of bacterial insertion sequences. Molecular Microbiology, 4: 1771-1777. Fernandez D., Ouinten M., Tantaoui A., Geiger J. P. e Langin T. (1999). Fot1 insertions in the Fusarium oxysporum f. sp. albedinis genome provide diagnostic PCR target for detection of the date palm pathogen. Applied and Environmental Microbiology 64: 633-636. Finnegan D. J. (1989). Eukaryotic transposable elements and genome evolution. Trends in Genetics, 5: 103-107. Finnegan D. J. (1997). Transposable elements: how non-LTR retrotransposons do it. Current Biology, 7: 245-248. Fravel D. R. (1988). Role of the the antibiosis in the biocontrol of plant disease. Annual Review of Phytopathology, 26: 75-91.
112
IV
Fravel D. R., Lewis J. A. e Chittams J. L. (1995). Alginate prill formulation of Talaromyces flavus with organic carriers for biocontrol of Verticillium dahliae. Phytopathology, 85: 165-168. Fravel D. R., Marois J. J., Lumsden R. D. e Connick W. J., Jr. (1985). Encapsulation of potential biocontrol agents in an alginate-clay matrix. Phytopathology, 75: 774-777. Fry W. E. (1982). Priciples of plant disease management. Academic Press, pp. 1-12. Galas D. J. e Chandler M. (1989). Bacterial insertion sequences. In Mobile DNA, Berg, D.E. e Howe, M.M. (coord.), American Society for Microbiology, Washington, DC, pp. 109-162. Gardiner K., Laas W. e Patterson D. (1986). Fractionation of large mammalian DNA restriction fragments using vertical pulsed-field gradient gel electrophoresis. Somatic Cell and Molecular Genetics, 12: 185-195. Garibaldi A., Brunatti F., Allocchio A. (1985). Terreni repressivi verso Fusarium oxysporum f. sp. dianthi: isolamento di microrganismi e loro attivit antagonistica in vaso. La difesa delle piante, 2: 101-106. Garibaldi A., Gullino M. L. (1989). La lotta biologica contro i funghi fitopatogeni: terreni repressivi. Informatore Fitopatologico, 39: 9-17. Garibaldi A., Pergola G., Dalla Guda C. (1980). Sulla presenza in Italia di terreni repressivi nei riguardi della fusariosi del garofano. ATTI Giornate Fitopatologiche: 458-464. Garibaldi A., Piglionica V., Gullino M. L. (1986). Fungicidi sistemici in Italia: impiego, problemi e prospettive. La difesa delle piante, 9: 85-132. Garibaldi A. e Gullino M. L. (1995). Focus on critical issues in soil solarization and substrate disinfestation toward the year 2000. Acta Horticulturae, 382: 2136. Gerik J. S., Lommel S. A. e Huisman O. C. (1987). A specific serological staining procedure for Verticillium dahliae in cotton root tissue. Phytopathology, 77: 261-266.
113
IV
Gerlach W. e Nirenberg H. (1982). The genus Fusarium - A pictorial atlas. Mitteilungen ous der Biologischen Bundesanstalt fr Land und Forstwirtschaft Berlin-Dahlem, Heft 209, Paul Parey, Berlin. Gibson N. J. (1997). A homogeneous method for genotyping with fluorescence polarization. Clinical Chemistry, 43: 1336-1341. Gierl A. (1996). The En/Spm transposable element of maize. Current Topics in Microbiology and Immunology, 204: 145-159. Gilbert M. T. (1987). High Performance Liquid Chromatography. John WrightPSG, Littleton, MA. Gillings M. e Holley M. (1997). Repetitive elements PCR fingerprinting (repPCR). using enterobacterial repetitive intergenic consensus (ERIC). primers is not necessarily directed at ERIC elements. Letters in Applied Microbiology, 25: 17-21. Gleason M. L., Ghabrial S. A., e Ferris R. S (1987). Serological detection of Phomopsis longicolla in soybean seeds. Phytopathology, 77: 371-375. Goidanich G. 1964. Manuale di patologia vegetale. Vol. 2. Edizioni agricole, Bologna, pp. 1202. Gordon T. R. e Okamoto D. (1992). Population structure and the relationship between pathogenic and nonpathogenic strains of Fusarium oxysporum. Phytopathology, 82: 73-77. Gordon T. R. e Martyn R. D. (1997). The evolutionary biology of Fusarium oxysporum. Annual Review of Phytopathology, 35: 111-128. Gordon W. L. (1952). The occurrence of Fusarium species in Canada. II. Prevalence and taxonomy of Fusarium species in cereal seed. Canadian Journal of Botany, 30: 209-251. Gothoskar S. S., Scheffer R. P., Walker J. C. e Stahmann M. A. (1953). The role of pectic enzymes in Fusarium wilt of tomato. Phytopathology, 43: 535-536. Gothoskar S. S., Scheffer R. P., Walker J. C. e Stahmann M. A. (1955). The role of enzymes in the development Fusarium wilt of tomato. Phytopathology, 45: 381-387. Gotz H. e Arnold C.-G. (1980). Comparative electrophoretic study of ribosomal proteins from algae. Planta, 49: 19-26.
114
IV
Grajl-Martn M. J., Simon C. J. e Muehlbauer F. J. (1993). Use of random amplified polymorphic DNA (RAPD) to characterize race 2 of Fusarium oxysporum f. sp. pisi. Phytopathology, 83: 612-614. Granada E. G. de Amezquita M. O. e Arbelez G. (1999). Using aryl esterase electrophoresis techniques to distinguish between Fusarium oxysporum f. sp. dianthi races. In: Proceedingss of the International Symposium on Cut Flowers in the Tropics. 14-17 October 1997, Bogot, Colombia. Acta Horticulturae, 482: 133-137. Gresshoff P. M. (1995). The interface between RFLP techniques, DNA amplification and plant breeding. In: "New diagnostics in crop science; biotechnology in agriculture, No. 13", Skerritt, J.H. e Appels, R. (coord.), CAB International, pp. 101-125. Gueldner R. C., Reilly C. C., Pusey P. L., Costello C. E., Arendale R. F., Cox R. H.,Himmelsbach O. S., Crumley R. C., Cutler H. G. (1990). Isolation and identification of iturins as antifungal peptides in biological control of peach brown rot with Bacillus subtilis. Journal of Agricultural and Food Chemistry, 36: 366-370. Gullino M. L. e Garibaldi A. (1987). Fungicide resistance in Botrytis cinerea and ways to cope with it. 7th Congress of the Mediterranean Phytopathological Union-Granada (Spain) Gullino M. L. (1995). Use of biocontrol agents against fungal disease. Proceding Congress on Microbial Agents in Sustainable Agriculture-Saint Vincent; pp. 50-59. Gullino M. L., Migheli Q. (1999). Risk analysis in the release of biological control agents: antagonistic Fusarium oxysporum as a case study. Proceedingss of the International Symposium on Cut Flowers in the Tropics. Eds. G. Fischer, A. Angarita. Acta Horticulturae.,482: 145-151. Gullino M. L., Migheli Q., Mezzalama M. (1995). Risk analysis in the release of biological control agents: antagonistic Fusarium oxysporum as a case study. Plant Disease, 79: 1193-1201. Gullino M. L. (1987). La resistenza ai fungicidi. Informatore Fitopatologico, 37: 9-15.
115
IV
Gundersen D. E. e Lee I.-M. (1996). Ultrasensitive detection of phytoplasmas by nested-PCR assay using two universal primer pairs. Phytopathologia Mediterranea, 35: 144-151. Gutterson N. I., Layton T. J., Ziegle J. S., Warren G. J. (1986). Molecular cloning of genetic determinants of fungal growth by a fluorescent pseudomonad. Journal of Bacteriology, 165: 696-703. Haas D., Keel C., Laville J., Maurhofer M., Oberhansli T. et al. (1991). Secondary metabolites of Pseudomonas fluorescens strain CHA0 involved in suppression of root disease. In: Advance in Molecular Genetics of PlantMicrobe Interactions, H. Hennecke, H., Verma, D. P. S. (coord.), 1: 450-456. Amsterdam: Kluwer Academic. Hall R., Zentmyer G. A, Erwin D. C. (1969). Approach to taxonomy of Phytophthora through acrylamide gel-electrophoresis of proteins. Phytopathology, 59: 770-774. Hames B. D. e Rickwood D. (1981). Gel electrophoresis of proteins: A practical approach. IRL Press, Oxford-Washington DC. Hawkworth D. L. (1988). Identification. In: "Living resources for biotechnology. Filamentous fungi", Hawkworth, D.L. e Kirsop, B.E. (coord.), Cambridge University Press, Cambridge, pp. 100-114. Hebbar K. P., Lumsden R. D., Poch S. M. e Lewis J. A. (1996). Agricultural by producs as subtrates for growth, conidiation and chlamidospore formation by a potential mycoherbicide, Fusarium oxysporum strain EN-4. Biocontrol Science and Technology, 6: 263-275. Hebbar K. P., Lumsden R. D., Poch S. M. e Lewis J. A. (1997). Liquid fermentation to produce biomass of mycohrbicidal strains of Fusarium oxysporum. Applied Microbiology and Biotehnology, 48: 714-719. Henson J. M. e French R. (1993). The polymerase chain reaction and plant disease diagnosis. Annual Review of Phytopathology, 31: 81-109. Holland P. M., Abramson R. D., Watson R. e Gelfand D. H. (1991). Detection of specific polymerase chain reaction product by utilizing the 5'-3' exonuclease activity of Thermus aquaticus DNA polymerase. Proceedings of the National Academy of Sciences USA, 88: 7276-7280.
116
IV
Houck C. M., Rinehart F. P. e Schmid C. W. (1979). A ubiquitous family of repeated DNA sequences in the human genome. Journal of Molecular Biology, 132: 289-306. Howell C. R., Stipanovic R. D. (1983). Gliovirin, a new antibiotic from Gliocladium virens, and its role in the biological control of Pythium ultimum. Canadian Journal of Microbiology, 29: 321-324. Howie W., Suslov T., (1986). Effect of antifungal compound biosythesis on cotton root colonization and Pythium suppression by a strain of Pseudomonas fluorescens and its antifungal minus isogenic mutant. Phytopathology, 76: 1069. Howie W., Suslov T., (1991). Role of antibiotic biosynthesis in the inhibition of Pythium ultimum in the cotton spermosphere and rhizosphere by Pseudomonas fluorescens. Molecular Plant-Microbe Interactions, 4: 393-399. Hua-Van A., Hericourt F., Capy P., Daboussi M. J. e Langin T. (1998). Three highly divergent subfamilies of the impala transposable element coexist in the genome of the fungus Fusarium oxysporum. Molecular and General Genetics, 259: 354-362. Hutchinson C. A. I., Hardies S. C., Loeb D. D., Shehee W. R. e Edgell M. H. (1989). LINEs and related retroposons; Long Interspersed Repeated sequences in the eucaryotic genome. In Mobile DNA, Berg, D.E. e Howe, M.M. (coord.), American Society for Microbiology, Washington, DC, pp. 593-617. Iannelli D., Capparelli R., Marziano F. Scala F. e Noviello C. (1983). Production of hybridomas secreting monoclonal antibodies to the genus Fusarium. Mycotaxon, 17: 1289-1293. Jacobson D. J. e Gordon T. R. (1990). Variability of mitochondrial DNA an indicator of relationship between populations of Fusarium oxysporum f. sp. melonis. Mycological Research, 94: 734-744. Jacobson J. W., Medhora M. M. e Hartl D. L. (1986). Molecular structure of a somatically unstable transposable element in Drosophila. Proceedings of the National Academy of Sciences USA, 83: 8684-8688. Janssen P., Coopman R., Huys G., Swings J. e Bleeker M. (1996). Evaluation of the DNA fingerprinting method AFLP as a new tool in bacterial taxonomy. Microbiology, 142: 1881-1893.
117
IV
Jarvik T. e Lark K. G. (1998). Characterization of Soymar1, a mariner element in soybean. Genetics, 149: 1569-1574. Jeffreys A. J., Wilson V. e Thein S. L. (1985). Hypervariable "minisatellite" regions in human DNA. Nature, 314: 67-73. Joffe A. Z. (1974). A modern system of Fusarium taxonomy. Mycopathologia et Mycologia Applicata, 53: 201-228. Johnson R. (1992). Past, present and future opportunities in breeding for disease resistance. In: Breeding for disease resistance (Johnson R., Jellis G.J. coord.). Kluwer Academic Plubishers, Dordrecht, pp. 3-22. Jones M. J. e Dunkle L. D. (1993). Analysis of Cochliobolus carbonum races by PCR amplification with arbitrary and gene-specific primers. Phytopathology, 83: 366-370. Julien J., Poirier H. S. e Brygoo Y. (1992).Foret1, a reverse transcriptaselike sequence in the filamentous fungi Fusarium oxysporum. Nucleic Acids Research, 20: 3933-3937. Kasahara S., Wang P. e Nuss D. (2000). Identification of bdm-1, a gene involved in G protein beta-subunit function and alfa-subunit accumulation. Microbiology, 97: 412-417. Kasuga T. e Mitchelson K. R. (1994). The development of a rapid identification of Heterobasidiom annosum intersterility groups by ribosomal RNA gene polymorphism and by minisatellite allele polymorphism. In: "Modern assays for plant pathogenic fungi: Identification, detection and quantification", Schots, A., Dewey, F.M. e Oliver, R. (coord.), CAB International, Wallingford, UK, pp. 117126. Katan J. (1987). Soil solarization. In: Innovative Approaches to Plant Disease Control (Chet I. coord.). Wiley and Sons, New York, pp. 77-105. Keel C., Schnider U., Maurhofer M., Voisard C., Laville J. (1992). Suppression of root disease by Pseudomonas fluorescens CHA0: importance of the bacterial secondary metabolite 2,4-diacetylphloroglucinol. Molecular PlantMicrobe Interactions, 5: 4-13. Kelly A., Alcal-Jimnez A. R., Bainbridge B. W., Heale J. B., Prez-Arts E. e Jimnez-Diaz R. M. (1994). Use of genetic fingerprinting and random
118
IV
amplified polymorphic DNA to characterize pathotypes of Fusarium oxysporum f. sp. ciceris infecting chickpea. Phytopathology, 84: 1293-1298. Kempken F. e Kck U. (1996). Restless, an active Ac-like transposon from the fungus Tolypocladium inflatum: structure, expression and alternative RNA splicing. Molecular and Cellular Biology, 16: 6563-6572. Kerr A. (1980). Biological control through production of Agrocin 84. Plant Disease, 64: 25-30. Kerr A. (1963). The root rot-Fusarium wilt complex of peas. Australian Journal of Biological Sciences, 16: 55-69. Kerssies A., Everink A., Hornstra L. e Van Telgen H. J. (1994). Electrophoretic detection method for Fusarium oxysporum species in cyclamen and carnation by using isoenzymes. In: "Modern assays for plant pathogenic fungi: Identification, detection and quantification", Schots, A., Dewey, F.M. e Oliver, R. (coord.), CAB International, Wallingford, UK, pp. 57-62. Kidwell M. G. (1994). The evolutionary history of the P family of transposable elements. Journal of Heredity, 85: 339-346. Kim K. K., Fravel D. R. e Papavizas G. C. (1988). Identification of a metabolite produced by Talaromyces flavus as glucose oxidase and its role in biocontrol of Verticillium dahliae. Phytopathology, 78, 488-92. Kim D. H., Martyn R. D. e Magill C. W. (1990). Preliminary investigation of geographic isolates and races of Fusarium oxysporum f. sp. niveum using mitochondrial DNA restriction fragment length polymorphisms (Abstract). Phytopathology, 80: 435. Kim D. H., Martyn R. D. e Magill C. W. (1991). Mitochondrial DNA restriction fragment length polymorphisms in Fusarium oxysporum f. sp. niveum. Phytoparasitica, 19: 211-223. Kim D. H., Martyn R. D. e Magill C. W. (1992). Restriction fragment length polymorphism groups and physical map of mithocondrial DNA from Fusarium oxysporum f. sp. niveum. Phytopathology, 82: 346-353. Kim D. H., Martyn R. D. e Magill C. W. (1993). Mitochondrial DNA (mtDNA)relatedness among formae speciales of Fusarium oxysporum in the Cucurbitaceae. Phytopathology, 83: 91-97.
119
IV
Kimura M. (1968). Evolutionary rate at the molecular level. Nature, 217: 624626. Kistler H. C., Bosland P. W., Benny U., Leong S. e Williams P. H. (1987). Relatedness of strains of Fusarium oxysporum from crucifers measured by examination of mitochondrial and ribosomal DNA. Phytopathology, 77: 12891293. Kistler H. C., Momol E. A. e Benny U. (1991). Repetitive genomic sequences for determining relatedness among strains of F. oxysporum. Phytopathology, 81: 331-336. Kitagawa T., Sakamoto Y., Furumi K. e Ogwra H. (1989). Novel enzyme immunoassay for specific detection of Fusarium oxysporum f. sp. cucumerinum and for the general detection of various Fusarium species. Phytopathology, 79: 162-165. Kleckner N. (1989). Transposon Tn10. In Mobile DNA, Berg, D. E. e Howe, M.M. (coord.), American Society for Microbiology, Washington, DC, pp. 227268. Kloepper J. W. e Schroth M. N. (1981). Development of a powder formulation of Rhizobacteria for inoculation of potato seedpieces. Phytopathology, 71: 590592. Kohler G. e Milstein C. (1975). Continuous of cultures fused cells secreting antibody of predefined specifity. Nature, 256: 495-497. Kostrikis L. G, Huang Y., Moore J. P., Wolinsky S. M., Zhang L., Guo Y., Deutsch L., Phair J., Neumann A. U. e Ho D. D. (1998). A chemokine receptor 2 allele delays HIV-1 disease progression. Nature Medicine, 4: 350-353. Kostrikis L. G., Tyagi S., Mhlanga M. M., Ho D. D. e Kramer F. R. (1998). Molecular beacons: spectral genotyping of human alleles. Science, 279: 12281229. Kulkosky J., Jones K. S., Katz R. A., Mack J. P. e Skalka A. M. (1992). Residues critical for retroviral integrative recombination in a region that is highly conserved among retroviral/retrotransposon integrases and bacterial insertion sequence transposases. Molecular and Cellular Biology, 12: 2331-2338.
120
IV
Lanfranco L., Wyss P., Marzach, C. e Bonfante P. (1993). DNA probes for identification of the ectomycorrhizal fungus Tuber magnatum Pico. FEMS Microbiology Letters, 114: 245-252. Langin T., Capy P. e Daboussi M. J. (1995). The transposable element impala, a fungal member of the Tc1-mariner superfamily. Molecular and General Genetics, 246: 19-28. Lee I. M., Hammond R. W., Davis R. E. e Gundersen D. E. (1992). Phylogenetic relationships among plant pathogenic mycoplasmalike organisms (MLOs). based on 16S rRNA sequence analysis (Abstract). Phytopathology, 82: 1094. Lee I.-M., Hammond R. W., Davis R. E. e Gundersen D. E. (1993). Universal amplification and analysis of pathogen 16S rRNA for classification and identification of mycoplasma-like organisms. Phytopathology, 83: 834-842. Lehninger H. L. (1979). Biochimica. Zanichelli, Bologna. Leslie J. F. (1993). Fungal vegetative compatibility. Annual Review of Phytopathology, 31: 127-150. Lewis J. A. e Papavizas G. C. (1985). Characteristics of alginate pellets formulated with Trichoderma and Gliocladium and their effect on the proliferations of the fungi in soils. Plant Pathology, 34: 571-577. Lewis J. A., Fravel D. R., Lumsden R. D. e Shasha B. S. (1995). Application of fungi in granular formulations of pregelatinized starch-four to control damping-off disease caused by Rhizoctonia solani. Biological Control, 5: 397404. Liddell C. M. 1991. Introduction: recent advances in Fusarium systematics. Phytopathology, 81: 1044. Liebermann D., Hoffman L. B., Weinthal J., Childs G., Maxson R., Maeron A., Cohen S. N. e Kedes L. (1983). An unusual transposon with long terminal inverted repeats in the sea urchin Strongylocentrotus purpuratus. Nature, 306: 342-347. Livak K. J., Marmaro J. e Todd J. A. (1995). Toward fully automated genomewide polymorphism screening. Nature Genetics, 9: 341-342.
121
IV
Louws F. J., Bell J., Medina-Mora C. M., Smart C. D. e Opgenorth D. (1998). Rep-PCR mediated genomic fingerprinting: a rapid and effective method to identify Clavibacter michiganensis. Phytopathology, 88: 862-868. Louws F. J., Fulbright D. W., Stephens C. T. e de Bruijn F. J. (1994). Specific genomic fingerprinting of phytopathogenic Xanthomonas and Pseudomonas pathovars and strains generated with repetitive sequences and PCR. Applied and Environmental Microbiology, 60: 2286-2295. Louws F. J., Rademaker J. L. W. e de Bruijn F. J. (1999). The three Ds of PCR-based genomic analysis of phytobacteria: Diversity, detection, and disease diagnosis. Annual Review of Phytopathology, 37: 81-125. Magnano di San Lio G., Scala F. e Migheli Q. (1995). Moderne tecniche diagnostiche in micologia fitopatologica. Petria, 5: 261-298. Manceau C. e Horvais A. (1997). Assessment of genetic diversity among strains of Pseudomonas siringae by PCR-restriction fragment length polymorphism analysis of rRNA operon with special emphasis on P. syringae pv. tomato. Applied and Environmental Microbiology, 63: 498-505. Manicom B. Q., Bar-Joseph M., Rosner A., Vigodsky-Haas H. e Kotze J. M. (1987). Potential applications of random DNA probes and restriction fragment length polymorphism in the taxonomy of Fusaria. Phytopathology, 77: 669-672. Manulis S., Kogan N., Reuven M. e Ben-Jephet Y. (1994). Use of the RAPD technique for identification of Fusarium oxysporum f. sp. dianthi from carnation. Phytopathology, 84: 98-101. Marshall M. R. e Partridge J. E. (1981).Fusarium moniliforme detection corn (Zea mays L.). stalk tissue: identification of specific ribosomal proteins by polyacrilamide gel electrophoresis. Physiological and Molecular Plant Pathology, 18: 133-144. Martin F. N. (1991). Selection of DNA probes useful for isolate identification of two Pythium species. Phytopathology, 81: 742-746. Matsuo T. (1998).In situ visualization of mRNA for basic fibroblast growth factor in living cells. Biochimica et Biophysica Acta, 1379: 178-184. Matta A. (1996). Fondamenti di patologia vegetale. Patron editore: p. 9, 14-15.
122
IV
Matuo T. (1972). Taxonomic studies of phytopathogenic Fusaria in Japan. Review of Plant Protection Research, 5: 34-45. McClintock B. (1956). Controlling elements and the genes. Cold Spring Harbor Symp. Quant. Biol. 21: 197-216. McClure T. T. (1950). Anatomical aspects of the Fusarium wilt of sweet potatoes. Phytopathology, 40: 769-775. McCluskey K. e Mills D. (1990). Identification and characterization of chromosome length polymorphisms among strains representing fourteen races of Ustilago hordei. Molecular Plant-Microbe Interactions, 3: 366-373. McPherson M. J., Oliver R. J. e Gurr S. J. (1992). The polymerase chain reaction. In: "Molecular Plant Pathology: A practical approach, Vol. 1", Gurr, S.J., McPherson, M.J. e Bowles, D.L. (coord.), IRL Oxford University Press, pp. 123-145. Messiaen C. M. e Cassini R. (1981). Taxonomy of Fusarium. In: "Fusarium: Diseases, Biology and Taxonomy", Nelson, P.E., Toussoun, T.A. e Cook, R.J. (coord.), The Pennsylvania University Press, University, pp. 427-445. Micales J. A., Bonde M. R. e Peterson G. L. (1986). The use of isozyme analysis in fungal taxonomy and genetics. Mycotaxon, 27: 405-449. Michelmore R. W. e Hulbert S. H. (1987). Molecular markers for genetic analysis of phytopathogenic fungi. Annual Review of Phytopathology, 25: 383404. Migheli Q., Friard O., Del Tedesco D., Musso M. R. e Gullino M. L. (1996). Stability of transformed antagonistic Fusarium oxysporum strains in vitro and in soil microcosms. Molecular Ecology, 5: 641-649. Migheli Q., Ghignone S., Chiocchetti A., Minuto G., Garibaldi A. (1998). Identificazione di Fusarium oxysporum f. sp. basilici mediante la tecnica RAPDPCR. Colture Protette, 3: 55-59. Migheli Q., Gullino M. L. (1990). La lotta biologica contro funghi fitopatogeni. III. Trichoderma e Gliocladium spp. come mezzi biologici di lotta. Informatore Fitopatologico, 40 (9), 9-18.
123
IV
Migheli Q. e Cavallarin L. (1994). Characterization of antagonistic and pathogenic Fusarium oxysporum isolates by random amplification of polymorphic DNA. Molecular Biotechnology, 2: 197-200. Migheli Q., Briatore E., Andrina M. e Garibaldi A. (1996). Physiological race determination in Fusarium oxysporum f. sp. dianthi by RAPD-PCR. In: Proceedings of the 4th International Symposium of the European Foundation for Plant Pathology, 9-12 September 1996, Bonn, Germany, pp. 203-206. Mills D. e McCluskey K. (1990). Electrophoretic karyotype of fungi: the new cytology. Molecular Plant-Microbe Interactions, 3: 351-357. Milstein C. (1980). Monoclonal antibodies. Scientific American, 243 (4): 66-74. Minuto A., Migheli Q. e Garibaldi A. (1995). Evaluation of antagonistic strains of Fusarium spp. In the biological and integrated control of Fusarium wilt of cyclamen. Crop Protection, 14: 221-226. Minuto A., Minuto G., Migheli Q., Mocioni M. e Gullino M. L. (1997). Effect of antagonistic 16: 765-769. Mohan S. B. (1988). Evaluation of antisera raised against Phytophthora fragariae for detecting the red core disease of strawberries by enzyme-linked immunosorbent assay (ELISA). Plant Pathology, 38: 352-363. Mohan S. B. (1988). Evaluation of antisera raised against Phytophthora fragariae for detecting the red core disease of strawberries by enzyme-linked immunosorbent assay (ELISA). Plant Pathology, 38: 352-363. Morgan G. T. (1995). Identification in the human genome of mobile elements spread by DNA-mediated transposition. Journal of Molecular Biology, 254: 1-5. Motohashi R., Ohtsubo E. e Ohtsubo H. (1996). Identification of Tnr3, a suppressor-mutator/enhancer-like transposable element from rice. Molecular and General Genetics, 250: 148-152. Mouyna I., Renard J. L. e Brygoo Y. (1996). DNA polymorphism among Fusarium oxysporum f. sp. elaeidis populations from oil palm, using repeated and dispersed sequence Palm. Current Genetics, 30: 174-180. Fusarium spp. and of different commercial biofungicide formulations on Fusarium wilt of basil (Ocimum basilicum L.). Crop Protection
124
IV
Murphy E. (1989). Transposable elements in Gram-positive bacteria. In Mobile DNA, Berg, D. E. e Howe, M.M. (coord.), American Society for Microbiology, Washington, DC, pp. 269-288. Nacken W. K., Piotrowiak R., Saedler H. e Sommer H. (1991). The transposable element Tam1 from Antirrhinum majus shows structural homology to the maize transposon En/Spm and has no sequence specificity of insertion. Molecular and General Genetics, 228: 201-208. Nathans D. e Smith H. O. (1975). Restriction endonucleases in the analysis and restructuring of DNA molecules. Annual Review of Biochemistry, 44: 273293. Nelson P. E., Toussoun T. A. e Marasas W. F. O. (1983). Fusarium species: An illustrated manual for identification. The Pennsylvania State University Press, University Park. Nesme X., Vaneechoutte M., Orso S., Hoste B. e Swngs J. (1995). Diversity and genetic relatedness within genera Xanthomonas and Stenotrophomonas using restriction-endonuclease site differences of PCR-amplified 16S ribosomalRNA gene. Systematic and Applied Microbiology, 18: 127-135. Newhouse J. R. e MacDonald W. L. (1991). The ultrastructure of hyphal anastomoses between vegetatively compatible and incompatible virulent and hypovirulent strains of Cyphonectria parasitica. Canadian Journal of Botany, 69: 602-614. Newton A. C. e Reglinski T. (1993). An enzyme-linked immunosorbent assay for quantifying mildew biomass. Journal of Plant Disease and Detection, 100: 176-179. Nicholson P. e Rezanoor H. N. (1994). The use of random amplified polymorphic DNA to identify pathotype and detect variation in Pseudocercosporella herpotrichoides. Mycological Research, 98: 13-21. Nishimura S. (1971). Observation on wilting mechanism of Fusarium-infected watermelon plants. Review of Plant Protection Research, 4: 71-80. Nonomura T., Toyoda H., Matsuda Y. e Ouchi S. (1996). Application of different fluorochrome-labelled probes to simultaneous in situ hybridization
125
IV
detection of formae speciales of Fusarium oxysporum. Annals of the Phytopathological Society of Japan, 62: 576-579. Notermans S. e Kamphuis H. (1990). Detection of mould in food by latex agglutination: a collaborative study. Food and Agriculture Immunology, 2: 37-47. Notermans S., Wieten G., Engel H. W. B., Rombouts R. M., Hoogerhout P. e Van Boom J. H. (1987). Purification and properties of extracellular polysaccharide (EPS). antigens produced by different mould species. Journal of Applied Bacteriology, 62: 157-166. Notermans S., Wieten G., Engel H. W. B., Rombouts R. M., Hoogerhout P. e Van Boom J. H. (1987). Purification and properties of extracellular polysaccharide (EPS). antigens produced by different mould species. Journal of Applied Bacteriology, 62: 157-166. Ochman H., Gerber A. S. e Harte D. L. (1988). Genetic application of an inverse polymerase chain reaction. Genetics, 120: 621-625. O'Donnell K. (1992). Ribosomal DNA internal transcribed spacer are highly divergent in the phytopathogenic ascomycete Fusarium sambucinum (Gibberella pulicaris). Current Genetics, 22: 213-220. Oliver R. (1993). Nucleic acid-based methods for detection and identification. In: "Principles of diagnostic techniques in plant pathology", Fox, R.T.V. (coord.). CAB International, Wallingford, UK, pp. 153-169. Oosumi T. e Belknap W. R. (1997). Characterization of the Sol3 family of nonautonomous transposable elements in tomato and potato. Journal of Molecular Evolution, 45: 137-144. Palti J. (1981). Cultural practices and infectious crop disease. Springer-Verlag, Berlin, pp. 243. Papavizas G. C. (1982). Biological control of selected soilborne plant pathogens with Gliocladium and Trichoderma. In: Biological Control of Plant Diseases (E. C. Tjamos, G. C. Papavizas, R. J. Cook, coord.), Plenum Press, New York, 223-230. Paran I. e Michelmore R. W. (1993). Development of reliable PCR-based markers linked to downy mildew resistance genes in lettuce. Theoretical and Applied Genetics, 85: 985-993.
126
IV
Partridge J. E (1991). Specific gene product of Fusarium: analysis of ribosomal proteins. Phytopathology, 81: 1055-1058. Pennypacker B. W. (1981). Anatomical changes involved in the pathogenesis of plant by Fusarium. In: "Fusarium: Diseases, Biology and Taxonomy", Nelson, P.E., Toussoun, T.A. e Cook, R.J. (coord.), The Pennsylvania University Press, University Park, pp. 399-408. Pennypacker B. W. e Nelson P. E. (1972). Histopathology of carnation infected with F. oxysporum f. sp. dianthi. Phytopathology, 62: 1318-1326. Phipps P. M. e Stipes R. J. (1976). Histopathology of mimosa infected with Fusarium oxysporum f. sp. perniciosum. Phytopathology, 66: 836-843. Piano S., Neyrotti V., Migheli Q., Gullino M. L. (1997). Biocontrol capability of Metschnikowa pulcherrima against Botrytis postharvest rot of apple. Postharvest Biology and Technology, 11: 131-140. Plyler T. R., Simone G. W., Fernandez D. e Kistler H. C. (1999). Rapid detection of the Fusarium oxysporum lineage containing the Canary Island date palm wilt pathogen. Phytopathology, 89: 407-413. Ponsonnet C. e Nesme X. (1994). Identification of Agrobacterium strains by PCR-RFLP analysis of pTi and chromosomal region. Archives of Microbiology, 161: 300-309. Poupard P., Grare S. e Cavelier N. (1991). Using ELISA method for assessment of in vitro development of strains of Pseudocercosporella herpotrichoides, the cereal eyespot fungus. Proceedingss of the Third International Conference of Plant Diseases, 3-5 December 1991, Bordeaux, France, pp. 655-662. Raillo A. (1950). Gryby roda Fusarium. State Publ. Moskva. Gos. Izd-vo selhoz. Lit-ry. Rhoades H. e Linford M. B. (1959). Control of Pythium root rot by nematode Aphelenchus avenae. Plant Disease Reporter, 43: 323-328. Risch S. J. (1987). Agricultural ecology and insect outbreaks. In: Insect outbreaks (Barbosa P. e Schultz J. C. coord.), Academic Press, New York, pp. 217-238.
127
IV
Ristaino J.B., Thomas W. 1997. Agriculture, bromide and the ozone hole: can we fill the gaps? Plant Disease, 81: 964-977. Robertson D. (1978). Characterization of a mutator system in maize. Mutation Research, 51: 21-28. Robertson H. M. (1996). Members of the pogo superfamily of DNA-mediated transposons in the human genome. Molecular and General Genetics, 252: 761766. Roitman J. M.,Mahoney N. E., Janisiewicz W. J. (1990). Production and composition of phenylpyrrole metabolites production by Pseudomonas cepacia. Applied Microbiology & Biotechnology, 34: 381-385. Rosewich U. L., Pettway R. E., Katan T. e Kistler H. C. (1999). Population genetic analysis corroborates dispersal of Fusarium oxysporum f. sp. radicislycopersici from Florida to Europe. Phytopathology, 89: 623-630. Roussos S., Olmos A., Raimbault M., Saucedo-Castaneda Lonsane B. K. (1991). Strategies for large scale inoculum development for solid state fermentation system: Conidiospores of Trichoderma harzianum. Biottechnol. Techn., 5 :415-420. Rovira A., Ryder M., Harris A. (1992). Biological control of root diseases with Pseudomonas. In: Biological Control of Plant Diseases (E. C. Tjamos, G. C. Papavizas, R. J. Cook, coord.), Plenum Press, New York, 175-184. Sadowsky M. J., Kinkel L. L., Bowers J. H. e Schottel J. L. (1996). Use of repetitive intergenic DNA sequences to classify pathogenic and disease suppressive Streptomyces strains. Applied and Environmental Microbiology, 62: 3489-3493. Saiki R. K., Gelfand D. H., Stoffel S., Scharf S. J., Higuci R., Horn G. T., Mullis K. B. e Erlich H. A. (1988). Primer directed enzymatic amplification of DNA with a thermostable DNA-polymerase. Science, 239: 487-491. Sano T., Smith C. L. e Cantor C. R. (1992). Immuno-PCR: a very sensitive antigen detection by means of specific antibody-DNA complexes. Science, 258: 120-122.
128
IV
Sauer K. M., Hulbert S. H. e Tisserat N. A. (1993). Identification of Ophiosphaerella herpotricha by cloned DNA probes. Phytopathology, 83: 97102. Scher F. M., Baker R. (1982). Effects of Pseudomonas putida and a synthetic iron chelator on induction of suppressiveness to Fusarium wilt pathogens. Phytopathology, 72: 1567-1573. Schildkraut C. L., Marmur J. e Doty P. (1961). The formation of hybrid DNA molecules and their use in studies of DNA homologies. Journal of Molecular Biology, 3: 595-617. Schilling A. G., Mller E. M. e Geiger H. H. (1996). Polymerase chain reaction-based assay for specific detection of Fusarium culmorum, F. graminearum, and F. avenaceum. Phytopathology, 86: 515-522. Schneider B., Ahrens U., Kirkpatrick B. C. e Seemuller E. (1993). Classification of plant-pathogenic mycoplasma-like organisms using restrictionsite analysis of PCR-amplified 16S rDNA. Journal of General Microbiology, 139: 519-527. Schofield P., Pell A. N. e Krause D. O. (1997). Molecular beacons: trial of a fluorescence-based solution technique for ecological studies with ruminal bacteria. Applied and Environmental Microbiology, 63: 1143-1147. Schwartz D. C. e Cantor C. R. (1984). Separation of yeast chromosome-sized DNAs by pulsed field gradient gel electrophoresis. Cell, 37: 67-75. Shapiro J. A., Sporn P. (1977). Tn402: a new transposable element determining trimethoprim resistance that inserts in bacteriophage lambda. Journal of Bacteriology, 129(3):1632-1635. Shapiro M. (1992). Use of optical brighteners as radiation protectans for gypsy moth (Lepidoptera: Lymantriidae) nuclear polyhedrosis virus. Journal of Economic Entomology, 85: 1682-1686. Shapiro M. e Robertson J. L. (1992). Ehnancement of gypsy moth (Lepidoptera: Lymantriidae) baculovirus activty by optical brighteners. Journal of Economic Entomology, 85: 1120-1124.
129
IV
Sheratt D. (1989). Tn3 and related transposable elements: site specific recombination and transposition. In Mobile DNA , Berg, D.E. e Howe, M.M. (coord.), American Society for Microbiology, Washington, DC, pp. 163-184. Shirsat A. H. (1988). A transposonlike structure in the 5 flanking sequence of a legumin gene from Pisum sativum. Molecular and General Genetics, 212: 129-133. Silver J. e Keerikatte V. (1989). Novel use of polymerase chain reaction amplify cellular DNA adjacent to an integrated provirus. Journal of Virology, 63: 1924-1928. Simon L., Lalonde M. e Bruns T. D. (1992). Specific amplification of 18S fungal ribosomal genes from vesicular-arbuscular endomycorrhizal fungi colonizing roots. Applied and Environmental Microbiology, 58: 291-295. Skinner D. Z., Budde A. D., e Leong S. A. (1991). Molecular karyotype analysis of fungi. In "More gene manipulation in fungi", Bennet, J.W. e Lasure, L.L. (coord.). Academic Press, San Diego, pp. 86-103. Slininger P. J. e Shea-Wilbur M. A. (1995). Liquid-culture pH, temperature and carbon (not nitrogen). source regulate phenazine productivity of the take all biocontrol agent Pseudomonas fluorescens 2-79. Applied Microbiology & Biotechnology, 43: 794-800. Smit A. F. e Riggs A. D. (1996). Tiggers and DNA transposon fossils in the human genome. Proceedings of the National Academy of Sciences USA, 93: 1443-1448. Smith R. e Walker J. C. (1930). A cytological study of cabbage plants in strains susceptible or resistant to yellows. Journal of Agriculture Research 41: 17-35. Snyder W. C. e Hansen H. N. (1940). The species concept in Fusarium. American Journal of Botany 27: 64-67. Southern E. M. (1975). Detection of specific sequences among DNA fragments separated by gel electrophoresis. Journal of Molecular Biology 98: 503-507. Sundaram S., Plasencia J. e Bantarri E. E. (1991). Enzyme-linked immunosorbent assay for detection of Verticillium spp. using antisera produced to V. dahliae from potato. Phytopathology, 81: 1485-1489.
130
IV
Sworder D. C., Waterpicture A. e Easter M. A. (1976). Decrease of timefreezing of np-glu
-
mutant of Neurospora crassa. New Zealand Journal of
Biological Sciences 3: 126-133. Sztejnberg A., Galper S., Mazar S. e Lisker N. (1989). Ampelomyces quisqualis for biological and integrated control of powdery mildew in Israel. J. Phytopathology, 124: 285-295. Taylor J. W. (1986). Fungal evolutionary biology and mitochondrial DNA. Experimental Mycology 10: 259-269. Thomashow L. S., Weller D. M. (1988). Role of a phenazine antibiotic fro Pseudomonas fluorescens in biological control of Gaeumannomyces graminis f. sp. tritici. Journal of Bacteriology, 170: 3499-3508. Thomashow L. S., Weller D. M., Bonsall R. F., Pierson L. S. III. (1990). Production of the antibiotic phenazin-1-carboxylic acid by Pseudomonas species in the rhizosphere of wheat. Applied Environmental Microbiology, 56: 908-912. Tisdale W. H. (1917). Flaxwilt: a study of the nature, and inheritance of wilt resistance. Journal of Agriculture Research 11: 573-605. Tooley P. W., Therrien C. D. e Ritch D. L. (1989). Mating type, race composition, nuclear DNA content, and isozyme analysis of peruvian isolates of Phytophthora infestans. Phytopathology, 79: 478-481. Triglia T., Peterson M. G. e Kemp D. J. (1988). A procedure for in vitro amplification of DNA segments that lie outside the boundaries of known sequences. Nucleic Acids Research, 16: 81-86. Tu Z. (1997). Three novel families of miniature inverted-repeat transposable elements are associated with genes of the yellow fever mosquito, Aedes aegypti. Proceedings of the National Academy of Sciences USA 94: 7475-7480. Tudor M., Lobocka M., Goodell M., Pettitt J. e OHare K. (1992). The pogo transposable element family of Drosophila melanogaster. Molecular and General Genetics, 232: 126-134. Tyagi S. e Kramer F. R. (1996). Molecular beacons: probes that fluoresce upon hybridization. Nature Biotechnology, 14: 303-308.
131
IV
Tyagi S., Bratu D. P. e Kramer F. R. (1998). Multicolor molecular beacons for allele discrimination. Nature, Biotechnology 16: 49-53. Ullstrup A. J. (1937). Histological studies on wilt of china aster. Phytopathology, 27: 737-748. Van Lenteren J. C., Minks A. K. e De Ponti O. M. B. (1992). Biological control and integrated crop protection: towards enviromentally safer agriculture. Pudoc, Wageningen, pp.239. Vaneechoutte M., Rossau R., De Vos P., Gills M. e Janssens D. (1992). Rapid identification of bacteria of the Commonadaceae with amplified ribosomal DNA-restriction analysis (ARDRA). FEMS Microbiology Letters, 93: 227-234. Versalovic J., Koeuth T. e Lupski J. R. (1991). Distribution of repetitive DNAsequences in eubacteria and application to fingerprinting of bacteria genomes. Nucleic Acids Research, 19: 6823-6831. Vet J. A. M., Majithia A. R., Marras S. A. E., Tyagi S., Dube S., Poiesz B. J. e Kramer F. R. (1999). Multiplex detection of four pathogenic retroviruses using molecular beacons. Proceedings of the National Academy of Sciences of the USA, 96: 6394-6399. Vidhyasekaran P., Sethuramam K., Rajappan K., Vasumathi K. (1997). Powder formulation of Pseudomonas fluorescens to control pigeonpea wilt. Biological Control, 8: 166-171. Vodkin L. O., Rhodes P. R. e Goldberg R. B. (1983). A lectin gene insertion has the structural features of a transposable element. Cell, 34:1023-1031. Vollrath D. e Davis R. W. (1987). Resolution of DNA molecules greater than 5 megabases by contour clamped homogeneous electric fields. Nucleic Acids Research, 15: 7865-7876. Vos P., Hogers R., Bleeker M., Rejans M. e van de Lee T. (1995). AFLP: a new technique for DNA fingerprinting. Nucleic Acids Research, 23: 4407-4414. Vruggink H. e Van Vuurde J. W. L. (1990). Enzyme-linked immunosorbent assay (ELISA). In: "Methods in Phytobacteriology", Klement, Z., Rudolph, K. e Sands, D.C. (coord.), Akadmiai Kiado, Budapest, Hungary, pp. 177-190.
132
IV
Ward D. M., Bateson M. M., Weller R. e Ruff-Roberts A. L. (1993). Ribosomal RNA analysis of microorganisms as they occur in nature. Advances in Microbial Ecology, 12: 219-286. Watson J. D. e Crick F. H. C. (1953). A structure for deoxyribose nucleic acid. Nature, 171: 737-738. Weber A. (1993). Population growtth, agricultural production and food supplies. Crop Production and Crop Protection, 1-71. Weller D. M. (1988). Biological control of soil-borne plant patthogens in the rhozosphere with bacteria. Annual Review of Phytopathology, 26:379-407. Welsh J. e McClelland M. (1990). Fingerprinting genomes using PCR with arbitrary primers. Nucleic Acids Research, 18: 7213-7218. Wetmur J. G. (1991). DNA probes: application of the principle of nucleic acid hybridization. Critical Review of Biochemical and Molecular Biology, 26: 227259. Wetzel T., Candresse T., Macquaire G., Ravelonandro M. e Dunez J. (1992). A high sensitive immunocapture polymerase chain reaction method for plum pox potyvirus detection. Journal of Virological Methods, 39: 27-37. Whipps J. M. e Gerlagh M. (1992). Biology of Coniothyrium minitans and its potential for use in disease biocontrol. Mycological Research, 96: 897-907. White T. J, Bruns T., Lee S. e Taylor J. (1990). Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. In: "PCR protocol: A guide to methods and application", Innis, M., Gelfand, D.H., Sninsky, J.J. e White, T.J. (coord), Academic Press, San Diego, CA, pp. 315-322. Widjojoatmodjo M. N., Fluit A. C., Torensma R., Keller B. H. I. e Verhoef J. (1991). Evaluation of the magnetic immuno PCR assay for rapid detection of Salmonella. European Journal of Clinical Microbiology and Infectious Disease, 10: 935-938. Williams J. G. K., Kubelik A. R., Livak K. J., Rafalski J. A. e Tingey S. V. (1990). DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Research, 18: 6531-6535.
133
IV
Williams J. G. K., Kubelik A. R., Rafalski J. A. e Tingey S. (1991). Genetic analysis with RAPD markers. In: "More gene manipulation in fungi", Bennet, J.W. e Lasure, L.L. (coord.). Academic Press, San Diego, pp. 431-439. Wollenweber H. W. e Reinking O. A. (1935). Die Fusarien, ihre Beschreibung, Schadwirkung und Bekmpfung, Paul Parey, Berlin. Wong W. C., White M. e Wright I. G. (1988). Production of monoclonal antibodies to Fusarium oxysporum f. sp. cubense race 4. Letters in Applied Microbiology 6: 39-42. Yeadon P. J. e Catcheside D. E. (1995). Guest, a 98 bp inverted repeat transposable element in Neurospora crassa. Molecular and General Genetics, 247: 105-109. Young R. J., Francis D. M., StClair D. A. e Taylor B. H. (1994). A dispersed family of repetitive DNA sequences exhibits characteristics of a transposable element in the genus Lycopersicon. Genetics, 137: 581-588. Yuan J. Y., Finney M., Tsung N. e Horvitz H. R. (1991). Tc4, a Caenorhabditis elegans transposable element with an unusual fold-back structure. Proceedings of the National Academy of Sciences USA, 88: 3334-3338.
134
You might also like
- Report Pesticidi Salute 2022Document25 pagesReport Pesticidi Salute 2022Francesco SchioppaNo ratings yet
- Attività Antiossidante, Antimutagena e Antigenotossica dei LattobacilliFrom EverandAttività Antiossidante, Antimutagena e Antigenotossica dei LattobacilliNo ratings yet
- 1.patogenicita' e VirulenzaDocument51 pages1.patogenicita' e VirulenzaFrancesco SchioppaNo ratings yet
- Sostanze Di Origine Naturale Ad Azione Erbicida PDFDocument31 pagesSostanze Di Origine Naturale Ad Azione Erbicida PDFcontadino_impazzitoNo ratings yet
- Balcone FioritoDocument4 pagesBalcone FioritoD3st1nyNo ratings yet
- Agricoltura Sostenibile (Ecocompatibile) Le Filiere AgroalimentariDocument64 pagesAgricoltura Sostenibile (Ecocompatibile) Le Filiere AgroalimentariDomenico Bevilacqua50% (2)
- Gestione Sostenibile Delle Erbe InfestantiDocument34 pagesGestione Sostenibile Delle Erbe InfestantipiripicchioNo ratings yet
- PRO E CONTRO BiotecnologieDocument2 pagesPRO E CONTRO BiotecnologieSonia Putrino75% (4)
- Piante Medicinali in Fitoterapia PDFDocument6 pagesPiante Medicinali in Fitoterapia PDFrodrigue yanNo ratings yet
- Terapia antibiotica e oli essenziali: La soluzione al problema della resistenza acquisita dai batteriFrom EverandTerapia antibiotica e oli essenziali: La soluzione al problema della resistenza acquisita dai batteriNo ratings yet
- Riassunto Patologia VegetaleDocument8 pagesRiassunto Patologia VegetaleGiorgioNo ratings yet
- Anticrittogamici Detti Fungicidi Definitivo Stop!Document52 pagesAnticrittogamici Detti Fungicidi Definitivo Stop!nat copNo ratings yet
- Perspectivas de Control BiologicoDocument44 pagesPerspectivas de Control BiologicoIKER NERINo ratings yet
- Biodiversit e AgrobiodiversitDocument29 pagesBiodiversit e Agrobiodiversitgiada innocentiNo ratings yet
- Appunti Microbiologia Degli AlimentiDocument129 pagesAppunti Microbiologia Degli AlimentiAntonio ScarpatoNo ratings yet
- Micotossine Negli AlimentiDocument57 pagesMicotossine Negli AlimentiMarco MacciNo ratings yet
- Dispensa Corso MicologicoDocument51 pagesDispensa Corso MicologicoWalter LentiniNo ratings yet
- Antecedentes Del TemaDocument4 pagesAntecedentes Del TemaCarla BarriosNo ratings yet
- Trattato Sul GanodermaDocument24 pagesTrattato Sul Ganodermahscappa8519No ratings yet
- Qualità Dei Prodotti AnimaliDocument60 pagesQualità Dei Prodotti AnimaliCaelo0No ratings yet
- Piante Medicinali in FitoterapiaDocument6 pagesPiante Medicinali in FitoterapiaSergioNo ratings yet
- Sicuro Che Mangiare Insetti Non Sia Rischioso - Focus - ItDocument3 pagesSicuro Che Mangiare Insetti Non Sia Rischioso - Focus - ItFrancesco SchioppaNo ratings yet
- Metaboliti e DifesaDocument83 pagesMetaboliti e DifesaalexsseverinoNo ratings yet
- L'agricoltura Biologica Stefano CanaliDocument9 pagesL'agricoltura Biologica Stefano CanaliSamNo ratings yet
- Microbiologia medica I: agenti patogeni e microbioma umanoFrom EverandMicrobiologia medica I: agenti patogeni e microbioma umanoNo ratings yet
- L'Erosione Genetica Di Specie Agrarie in Ambito MediterraneoDocument44 pagesL'Erosione Genetica Di Specie Agrarie in Ambito MediterraneoluzzaneddaNo ratings yet
- Tesi Giovanni Tuccio Per PDCDocument130 pagesTesi Giovanni Tuccio Per PDCjonaNo ratings yet
- MicologiaDocument13 pagesMicologiaMassimiliano PalumboNo ratings yet
- Produzioni Vegetali, Appunti Scienze Dell AlimentazioneDocument33 pagesProduzioni Vegetali, Appunti Scienze Dell AlimentazioneGiorgio SciarraNo ratings yet
- 1c Avversità Biotiche e Possibilità Di Intervento, SostenibilitàDocument110 pages1c Avversità Biotiche e Possibilità Di Intervento, SostenibilitàCarlos GolbanoNo ratings yet
- BiotecnologiaDocument18 pagesBiotecnologiamelissa lopezNo ratings yet
- Natu 16Document1 pageNatu 16piperinoNo ratings yet
- EDUCAZIONEAMBIENTALEDocument157 pagesEDUCAZIONEAMBIENTALEGaiaNo ratings yet
- Bioetica e OgmDocument3 pagesBioetica e OgmEnrico BoattinNo ratings yet
- Introduzione Alla MicrobiologiaDocument42 pagesIntroduzione Alla Microbiologiamaria_ponticelliNo ratings yet
- Biologia VegetaleDocument21 pagesBiologia VegetaleUomo RagnoNo ratings yet
- Avverità Biotiche e Difesa Delle Piante - 01:12Document10 pagesAvverità Biotiche e Difesa Delle Piante - 01:12EnricoCicinelliNo ratings yet
- Totale MicrobiologiaDocument214 pagesTotale MicrobiologiaguyNo ratings yet
- EM Libro HigaDocument11 pagesEM Libro Higaascionefrancesco85100% (1)
- 001introduzione MicrobiologiaDocument50 pages001introduzione MicrobiologiaAlessandra Patricia LopesNo ratings yet
- Il Brusone Del RisoDocument13 pagesIl Brusone Del RisoabyutzaNo ratings yet
- Ambiente e SaluteDocument201 pagesAmbiente e SalutegioelNo ratings yet
- Antivirali Naturali-EstrattoDocument9 pagesAntivirali Naturali-EstrattoKantiaeNo ratings yet
- Coltivare Funghi Commestibili: Guida Pratica con 20 Capitoli DettagliatiFrom EverandColtivare Funghi Commestibili: Guida Pratica con 20 Capitoli DettagliatiNo ratings yet
- 4° Microbiologia UFSB - Solo Parte IIIDocument2 pages4° Microbiologia UFSB - Solo Parte IIIJonatas Oliveira MeloNo ratings yet
- Ingenieria Genetica VegetalDocument12 pagesIngenieria Genetica VegetalJuan Camilo PabonNo ratings yet
- Appunti Patologia VegetaleDocument28 pagesAppunti Patologia Vegetalepietro90100% (1)
- Predazione, Pascolamento e MalattieDocument91 pagesPredazione, Pascolamento e MalattieMarta PerciballiNo ratings yet
- Orto Domestico: Manuale Pratico e Completo con Tecniche per Principianti e EspertiFrom EverandOrto Domestico: Manuale Pratico e Completo con Tecniche per Principianti e EspertiNo ratings yet
- DisbiosiDocument10 pagesDisbiosiandreaNo ratings yet
- Funghi curativi: Una guida pratica e culinaria Guida all'uso dei funghi per la salute di tutto il corpoFrom EverandFunghi curativi: Una guida pratica e culinaria Guida all'uso dei funghi per la salute di tutto il corpoNo ratings yet
- 04 TeiDocument18 pages04 TeimemoliaNo ratings yet
- BatteriologiaDocument69 pagesBatteriologiaAndrea ColomboNo ratings yet
- PatogeniDocument194 pagesPatogeniralapubsNo ratings yet
- Appunti Microbiologia Generale e Enologica PDFDocument87 pagesAppunti Microbiologia Generale e Enologica PDFMarco Lami100% (1)
- Giacchetti - Microbiologia - 3 Lezione TFPC - Patogenesi Batterica e Risposta OspiteDocument43 pagesGiacchetti - Microbiologia - 3 Lezione TFPC - Patogenesi Batterica e Risposta OspiteFrancesco SchioppaNo ratings yet
- Sbob Igiene 1 (27 - 04)Document14 pagesSbob Igiene 1 (27 - 04)Alessandro PalmaNo ratings yet
- Benessere Intestinale PDFDocument61 pagesBenessere Intestinale PDFmaria maggioNo ratings yet
- L ' Erboristeria VeterinariaDocument16 pagesL ' Erboristeria VeterinariapippoNo ratings yet
- Malattie Dei Cereali A PagliaDocument0 pagesMalattie Dei Cereali A Pagliapedro41No ratings yet
- Pound ScrittiEconomiciDocument123 pagesPound ScrittiEconomicipedro41No ratings yet
- GIORNATA Sulle Strategie Bioecologiche Di Lotta Contro Gli Organismi NociviDocument212 pagesGIORNATA Sulle Strategie Bioecologiche Di Lotta Contro Gli Organismi Nocivipedro41No ratings yet
- Ricette Umbre - AntipastiDocument3 pagesRicette Umbre - Antipastipedro41No ratings yet
- Lo Stress Ossidativo Nel Cavallo AtletaDocument24 pagesLo Stress Ossidativo Nel Cavallo Atletapedro41No ratings yet
- (Ebook - Architettura - ITA - PDF) Storia Dell'Architettura ContemporaneaDocument46 pages(Ebook - Architettura - ITA - PDF) Storia Dell'Architettura Contemporaneapedro41No ratings yet
- La Canapa in ToscanaDocument43 pagesLa Canapa in Toscanapedro41No ratings yet
- La Canapa in ToscanaDocument43 pagesLa Canapa in Toscanapedro41No ratings yet
- Il Recupero e La Gestione Delle Acque Di Vegetazione Dei Frantoi OleariDocument17 pagesIl Recupero e La Gestione Delle Acque Di Vegetazione Dei Frantoi Olearipedro41No ratings yet
- (Ebook-Manuale-ITA) - Emule - It - La Più Completa Guida in Italiano Ad EmuleDocument78 pages(Ebook-Manuale-ITA) - Emule - It - La Più Completa Guida in Italiano Ad Emulepedro41No ratings yet
- (ARCHITETTURA Ebook) Storia Dell'Arte Einaudi - Rudolf Wittkower - Idea e Immagine, Studi Sul Rinascimento ItalianoDocument198 pages(ARCHITETTURA Ebook) Storia Dell'Arte Einaudi - Rudolf Wittkower - Idea e Immagine, Studi Sul Rinascimento Italianopedro41100% (1)
- (Ebook-Manuale-ITA) - Emule - It - La Più Completa Guida in Italiano Ad EmuleDocument78 pages(Ebook-Manuale-ITA) - Emule - It - La Più Completa Guida in Italiano Ad Emulepedro41No ratings yet
- (Fai Da Te - Architettura) Gazebo E Casette in Legno Per Il GiardinoDocument6 pages(Fai Da Te - Architettura) Gazebo E Casette in Legno Per Il Giardinopedro41No ratings yet
- (Architecture Ebook - Ita) Architettura Ed Energia - La Progettazione Dell'illuminazione NaturaleDocument54 pages(Architecture Ebook - Ita) Architettura Ed Energia - La Progettazione Dell'illuminazione Naturalepedro41No ratings yet
- (Ebook - Ita - Cucina) Federazione Italiana Cuochi Sez. Di Rapallo - Raccolta Di Ricette IllustrateDocument76 pages(Ebook - Ita - Cucina) Federazione Italiana Cuochi Sez. Di Rapallo - Raccolta Di Ricette Illustratepedro41No ratings yet
- Architettura in Pietra Delle Barbagie, Dell'Ogliastra Del Nuorese e Delle Baronie PDFDocument4 pagesArchitettura in Pietra Delle Barbagie, Dell'Ogliastra Del Nuorese e Delle Baronie PDFpedro41No ratings yet
- (Ebook - Ita - Cucina) - Le Cento Migliori Ricette Per Le Cene in PiediDocument32 pages(Ebook - Ita - Cucina) - Le Cento Migliori Ricette Per Le Cene in Piedipedro41No ratings yet
- (Ebook - Ita - Botanica) Guida Illustrata Alla Potatura Delle Piante Da Giardino PDFDocument23 pages(Ebook - Ita - Botanica) Guida Illustrata Alla Potatura Delle Piante Da Giardino PDFpedro41No ratings yet
- Architetti I Giardini Zen e L Architettura Dello SpiritoDocument517 pagesArchitetti I Giardini Zen e L Architettura Dello SpiritoJardines OrientalesNo ratings yet
- (Ebook - Ita - Cucina) - Le Cento Migliori Ricette Per Le Cene in PiediDocument32 pages(Ebook - Ita - Cucina) - Le Cento Migliori Ricette Per Le Cene in Piedipedro41No ratings yet
- Grammatica Ungherese Ad Uso Degl ItalianiDocument268 pagesGrammatica Ungherese Ad Uso Degl Italianiitaloungherese100% (1)
- (Ebook - Ita - Botanica) Guida Illustrata Alla Potatura Delle Piante Da Giardino PDFDocument23 pages(Ebook - Ita - Botanica) Guida Illustrata Alla Potatura Delle Piante Da Giardino PDFpedro41No ratings yet
- (Ebook - ITA - MATEMATICA) Formulario Matematica GeneraleDocument15 pages(Ebook - ITA - MATEMATICA) Formulario Matematica GeneralePedro TorresNo ratings yet
- (Ebook - Ita - Cucina) - Le Cento Migliori Ricette Per Le Cene in PiediDocument32 pages(Ebook - Ita - Cucina) - Le Cento Migliori Ricette Per Le Cene in Piedipedro41No ratings yet
- (Ebook - Ita - Cucina) Cucina Siciliana Antiche RicetteDocument17 pages(Ebook - Ita - Cucina) Cucina Siciliana Antiche RicettebobisseNo ratings yet
- 07010000-PI Biotech ItaliaDocument68 pages07010000-PI Biotech Italiapedro41No ratings yet
- (Ebook - Ita - Cucina) - Le Cento Migliori Ricette Per Le Cene in PiediDocument32 pages(Ebook - Ita - Cucina) - Le Cento Migliori Ricette Per Le Cene in Piedipedro41No ratings yet