Professional Documents
Culture Documents
Metabolismo de RNA
Uploaded by
Cesar ParionaCopyright
Available Formats
Share this document
Did you find this document useful?
Is this content inappropriate?
Report this DocumentCopyright:
Available Formats
Metabolismo de RNA
Uploaded by
Cesar ParionaCopyright:
Available Formats
METABOLISMO DEL RNA
Introduccin.-
Expresin de la informacin gentica:
DNA (templado)
transcripcin
RNA.
RNA ~ DNA; OH 2 y U x T, hebra simple.
RNA hebra simple se pliega sobre si misma con ms diversidad estructural
que el DNA
Interviene en almacenamiento, transmisin de la informacin y catlisis.
Excepto algunos genomas virales, todos los RNAs derivan
de la informacin permanentemente almacenada en DNAs.
En la transcripcin, un sistema de enzimas transfiere la informacin
contenida en la doble hebra del DNA a una hebra de RNA con secuencia
complementaria a una de las hebras.
Tres RNAs:
mRNA codifica la secuencia de AA de uno ms polipptidos especificado
por uno un set de genes.
tRNA lee la informacin codificada en el mRNA y transfiere el AA indicado
rRNA constituyente de los ribosomas.
Otros RNAs tienen funciones catalticas o regulatorias o son precursores de
otros RNAs.
La transcripcin es ms selectiva (en la replicacin se copia todo el
cromosoma), genes particulares o grupos de genes en un determinado tiempo,
Regulacin
Algunas porciones del genoma, nunca son transcritas.
La clula restringe la expresin gentica a la formacin de productos necesarios
en un momento particular.
Secuencias regulatorias especficas marcan el inicio y el final del segmento
de DNA que se transcribe y designan que hebra de DNA va a ser usada como
templado.
Revisaremos:
Sntesis de RNA de un DNA templado
Procesamiento post-sinttico y turnover de RNA
RNA como templado de DNA
Sntesis de RNA dependiente de DNA.-
Transcripcin Vs. Replicacin:
Similitudes : mecanismo qumico, polaridad, uso de templado, fases de: iniciacin
elongacin y terminacin.
Diferencias: no requiere primer, limitada a segmentos de una molcula de DNA.
El RNA es sntetizado por la RNA polimerasa.-
Se le descubre por el 1960, la RNA polimerasa dependiente de DNA.
Requiere de un DNA templado, 4 Nu (A,T,G,U) y Mg
++
, une Zn
++
.
El mecanismo ~ DNApol, RNA pol elonga una hebra de RNA por adicin de Nu a
un extremo OH 3, en sentido 5-3.El nuclefilo OH 3, ataca el P, del NuTP que
se va a incorporar, liberandose PPi
(NMP)n + NTP
RNA polimerasa
(NMP)n+1 + PPi
La transcripcin ocurre por apareamiento de bases en una burbuja de
de DNA abierto
La RNA pol require DNA y es ms activa cuando se une a DNA de doble hebra,
Solo una hebra sirve como templado que es copiado en sentido 3- 5,
( antiparalela a la nueva hebra de RNA).
C/Nu es incorporado segn la regla de apareamiento de W yC.
Se inserta U, que se aparea con A del templado, la geometra del
apareamiento de bases determina la seleccin de bases.
La RNA pol no requiere primer, se une al promotor, El grupo tri-P de primer Nu
no es hidrlizado y se mantiene durante la transcripcin.
Durante la elongacin el extremo en crecimiento del RNA en formacin se
Mantiene forma temporal como un hibrido DNA -RNA de doble helix (8 bp )
Para permitir la accin de la RNA pol, el DNA duplex debe desenrrollarse sobre
una corta distancia formando una burbuja de transcripcin (en E. coli 17 bp, un
corto hibrido DNA-RNA en la regin desenrrollada, la v = 50 a 90 Nu/seg.
Como el DNA es una doble helix, el movimiento de la burbuja de transcripcin,
requiere de la rotacin de la molcula, la rotacin esta restricta por la unin de
protenas al DNA, como resultado el movimiento de la RNA pol genera ondas
de superenrrollamiento positivo al lado de la burbuja de transcripcin y
superenrrollamientos negativos por detrs. Los problemas topolgicos son
aliviados por accin de las topoisomerasas.
Las dos hebras complementarias en el DNA cumplen diferentes roles en la
Transcripcin, la hebra que sirve como templado se llama hebra templado,
La hebra complementaria al templado es la hebra no templado o codante que
es idntica en secuencia al RNA con U en lugar de T.
La hebra que codifica para un gen particular, puede estar localizada en
cualquiera de las dos hebras de un cromosoma dado.
Las secuencias regulatorias que controlan la transcripcin estn por convencin
designadas en la hebra no templada.
Genoma de adenovirus
La RNA pol de E. coli, es un complejo enzimtico grande con un core de
5 subunidades (
2
; Mr 390,000) y una subunidad .
se une al core y dirige la enzima a sitios especficos en el DNA, las 6
subunidades constituyen la holoenzima de la RNA pol.
La holoenzima de E. coli existe en varias formas dependiendo del tipo de ,
la ms comn es
70
(Mr 70,000).
RNA pol carece de actividad proofreading exonuclesa 3 - 5 pudiendose
generar un error por cada 10
4
10
5
Nu.
Generalmente se producen muchas copias de RNA de un simple gen y son
degradadas y remplazadas por lo que un error en el RNA no tiene tanta
consecuencia como un error en el DNA.
Holoenzima de la bacteria Thermus aquaticus
La sntesis de RNA se inicia en los promotores.-
La RNA pol se une a una secuencia especfica en el DNA llamada
promotor, que dirige la transcripcin de segmentos de DNA (genes)
adyacentes.
Las secuencias de los promotores son poco variables.
En E. coli la regin del promotor se extiende de -70 a +30.del sitio de
inicio
Secuencias de promotores reconocidos por
70
tienen similitudes en
dos secuencias cortas ubicadas en las posiciones -10 y -35, estas
secuencias son sitios de interaccin importantes para la subunidad 70,
aunque las secuencias no son idnticas en todos los promotores
bacteriales, ciertos Nu mantienen una posicin formando una secuencia
de consenso, (recordar en E. coli secuencias de consenso ori C ).
La secuencia de consenso en -10 es 5 TATAAT 3 y en -35 es 5 TTGACA 3
Un tercer elemento de reconocimiento rico en AT es llamado elemento UP
(up stream promotor) se presenta en promotores de ciertos genes que son
muy expresados, ubicado entre las posiciones 40 y 60, se le une la
subunidad
70
.
La eficiencia de la unin RNA pol con un promotor para iniciar la transcripcin
esta determinada por estas secuencias, el espaciamiento entre ellas y su
distancia del sitio de inicio de la transcripcin.
Mutaciones que afectan la funcin de un promotor a menudo involucran un bp
de las regiones -30 -10, variaciones en la regiones de consenso afectan la
Unin de la RNA pol y la iniciacin de la transcripcin.
El cambio en un solo bp, disminuye el grado de unin en varios ordenes de
magnitud.
La secuencia del promotor establece el nivel basal de expresin para genes
particulares en E. coli, que pueden variar grandemente de un gen al siguiente.
El inicio de la transcripcin consiste de dos etapas principales (con mltiples
pasos).
1 RNA pol se une al promotor formando sucesivamente un complejo cerrado
(el DNA unido est intacto) y un complejo abierto ( en el cual el DNA se
desenrrolla).
2 Se inicia la transcripcin en el complejo que conduce a un cambio
conformacional que convierte el complejo a la forma de elongacin,
seguido por el movimiento del complejo de transcripcin fuera del promotor
(promotor clearence), todos estos pasos pueden ser afectados por la
secuencias especfica de las secuencias del promotor.
E. coli tiene otros promotores que se unen con la holoenzima de la RNA pol, a
travs de diferentes factores . Ejm. Heat shock protenas. Los productos
de este set genes se producen a elevados niveles cuando las clulas son
agredidas, tal como un incremento de T. La RNA pol se une a los
promotores de estos genes cuando
70
es reemplazado con
32
(Mr 32,000),
Que es especfica para promotores heat shock proteins.
A travs del uso de diferentes factores ,
la clula puede coordinar la expresin de
genes que permitan mayores cambios en
Funcin a su estado fisiolgico.
La transcripcin es regulada.-
El requerimiento del producto de un gen vara con la condicin celular o
el estado de desarrollo, y la transcripcin es regulada para dar productos
solo en proporciones necesarias.
La regulacin puede producirse en cualquiera de los tres pasos, iniciacin
elongacin o terminacin, principalmente en los pasos de unin de la
Polimerasa e iniciacin de la transcripcin.
Las diferencias en las secuencias de promotores son uno de los varios
niveles de control.
La unin de protenas a secuencias cerca o distantes del promotor pueden
afectar los niveles de expresin del gen.
La unin de protenas pueden activar la transcripcin facilitando la unin de la
RNA pol, o pasos posteriores a lo largo del proceso de iniciacin.
Tambin pueden reprimir la transcripcin bloqueando la actividad de la
Polimerasa.
En E. coli La protena receptor cAMP (CRP) activa la transcripcin,
de genes que codifican enzimas que metabolizan azucares diferentes de
la glucosa.
Represores son protenas que bloquean la sntesis de RNA en genes
Especficos . Represor Lac, la transcripcin de genes para enzima que
metabolizan lactosa, estn bloqueados cuando no se dispone de lactosa.
La regulacin de la sntesis de protenas en bacterias y eucariotes, est
dirigida por la transcripcin sobre todo en sus primeros pasos.
Secuencias especficas sealan la terminacin de la transcripcin
La RNA pol tiene alta procesividad, si libera un transcrito prematuramente no
puede reasumir su sntesis.
Al encontrarse con ciertas secuencias en el DNA detiene la sntesis y en
algunas termina la transcripcin.
En eucariotes el proceso de terminacin no es muy conocido, en E coli , 2
tipos de seales:
a) Terminadores independientes de rho, en ellos el RNA transcrito tiene
secuencias autocomplementarias que forman un hairpin entre 15 a 20 Nu
antes del extremo. Adems una corta secuencia de A en el templado que
son transcritos como U en el extremo 3, cuando la polimerasa llega a un
sitio terminal con esta estructura, se detiene, el hairpin distorsiona las
interacciones entre RNA y RNA pol, disociandose el transcrito.
b) Los terminadores dependientes de rho, carecen de los A repetidos, pero
normalmente incluyen una secuencia rica en CA llamada elemento rut
(utilizacin de rho) la protena rho se asocia con el RNA en un sitio de unin
especfico y migra en direccin 5-3, hasta que el alcanza al complejo de
transcripcin que esta detenido en un sitio de terminacin, aqu contribuye a
la liberacin del transcrito.
La protena rho tiene una actividad helicasa dependiente de ATP, que
promueve la translocacin de la protena a lo largo del RNA, el ATP es
hidrolizado por rho durante el proceso de terminacin.
Las clulas eucarioticas tienen tres clases de RNApol nucleares
La maquinaria eucariotica es ms compleja que la bacterial.
Tres complejos distintos, con distinta funcin, que se unen a una secuencia
promotor especfico pero con algunas subunidades en comn:
RNA pol I transcribe solo el RNA preribosomal que contiene los precursores
para rRNAs 18S, 5.8S y 28S. Sus promotores varan mucho su secuencia
de una especie a otra.
RNA pol II transcribe mRNAs y algunos RNAs especiales. Reconoce miles
de promotores que varan mucho en sus secuencias. Muchos de sus
promotores tienen pocas secuencias comunes caja TATA (con la secuencia
de consenso TATAAA) cerca del par de bases -30 y una secuencia iniciador
Inr cerca del sitio de inicio en +1
RNA pol III transcribe tRNAs, el rRNA 5S y algunos RNAs pequeos
especiales. sus promotores estn bien caracterizados, algunas de sus
secuencias regulatorias del inicio estn en el mismo gen, otras
convencionalmente antes del sitio de inicio.
La RNApol II requiere de otros factores proticos para su actividad
Cumple un rol central en eucariotes, ha sido muy estudiada y es mucho
ms compleja que la bacterial, muy conservada estructura , funcin y
mecanismo.
Es una enzima muy grande con 12 subunidades, la subunidad mayor
(RBP1) tiene alto grado de homologa con bacterial , otra subunidad
RBP2 es similar a la subunidad y otras dos RBP3 y RBP11 son
homlogas a las estructuras de las 2 subunidades bacteriales.
Funciona con genomas ms complejos y con DNAs ms empacados
que los de bacteria.
La subunidad mayor (RBP1) tiene un largo tallo C-terminal con
muchas secuencias de consenso repetidas de 7 AA (YSPTSPS), en
enzimas de levaduras hay 27 repeticiones(18 componen el consenso)
y en enzimas de ratones y humanos 52 (21 en consenso), este
Dominio Carboxilo Terminal CTD esta separado del cuerpo principal
de la enzima por una secuencia linker no estructurada, El CTD
cumple roles muy importantes.
RNA pol II requiere de un arreglo de otras protenas, factores de
transcripcin, para formar un complejo de transcripcin activo.
Los factores de transcripcin generales requeridos en cada promotor
de la Pol II se designan como TFII con un identificador adicional, y
son altamente conservados en todos los eucariotes
La transcripcin por las
Pol II, se describe en
las fases de: ensamble,
iniciacin, elongacin y
terminacin c/u
asociado con protenas
caractersticas.
En la clula pueden
estar en grandes
complejos
preensamblados
simplificando las vas
para el ensamble sobre
los promotores.
1
2
3
4 (lleva a 5 a su blanco)
estabiliza
5
6
7
Helicasa -kinasa
(TFIIH y CDK9 - pTEFb)
9
8
TFIIF
10
Ensamble de la RNA pol y Factores de Transcripcin al promotor
La formacin de un complejo cerrado se inicia cuando la protena de
unin a TATA (TBP) se une a la caja TATA (1), TBP esta unido por TFIIB
que tambin une a DNA por todos los lados de TBP (2).
La unin de TFIIA aunque no es esencial puede estabilizar el complejo
TFIIB TBP en el DNA y puede ser importante en promotores sin-
consenso donde la unin de TBP es dbil. (3)
Al complejo TFIIB-TBP se une a otro complejo de TFIIF y RNA pol II.
TFIIF ayuda a llevar a la RNA pol a su blanco, esto es a sus promotores.
Debido a que interacta con TFIIB y por que reducen la unin de la RNA
pol a sitios no especficos en el DNA. (4 y 5)
Finalmente TFIIE y TFIIH se unen para crear un complejo cerrado ( 6 y 7),
TFIIH tiene una actividad de DNA helicasa desenrollado el DNA cerca del
sitio de inicio (requiere Hidrlisis de ATP) creando un complejo abierto, se
requieren ms de 30 polipptidos
Iniciacin de la hebra de RNA y Clearence del promotor
TFIIH tiene otra funcin, una de sus subunidades tiene actividad de kinasa,
que fosforila a la RNApol II en varios sitios del CTD. Otras proten kinasas
incluyendo a CDK9 (Kinasa dependiente de ciclina 9) que es parte del
complejo pTEFb (factor de transcripcin de elongacin positiva b), tambin
fosforila a CTD ; esto produce un cambio conformacional en el complejo
total inicindose la transcripcin. (8)
La fosforilacin es importante en la elongacin y afecta las interacciones
entre el complejo de transcripcin y otras enzimas del procesamiento del
transcrito.
Durante la incorporacin de los primeros 60 70 Nu, TFIIE y luego TFIIH
son liberados comenzndose la fase de elongacin.
Elongacin, Terminacin y Liberacin
TFIIF permanece asociado con RNA pol II a travs de la elongacin, la
actividad de la enzima se ve incrementada por factores de elongacin, (9)
Los factores de elongacin suprimen las pausas durante la transcripcin y
coordinan las interacciones entre los complejos de protenas involucrados
en el procesamiento post transcrpcional de los mRNAs.
completado el transcrito, el proceso termina por mecanismos no conocidos, la
RNA pol II es defosforilada y reciclada para iniciar otro transcrito. (10)
Regulacin de la actividad de la RNA pol II
La regulacin en los promotores, involucra la interaccin de otras protenas
con el complejo de preiniciacin, algunas protenas regulatorias interactan
con los factores de transcripcin otras con la misma Pol II, otras con TFIID, un
complejo de 12 prot incluyen TBP y ciertos factores asociados a TBP o TAFs.
Diversas funciones de TFIIH
La reparacin de DNA es ms eficiente en genes que estn siendo transcritos
y la hebra templado se repara ms eficiente que la no templada, algunas de
las subunidades de TFIIH son componentes esenciales del complejo de
reparacin por excisin de Nu.
Cuando la RNApol se detiene en un sitio de una lesin TFIIH puede
interactuar con la lesin y reclutar el complejo total de reparacin por excisin
de Nu , la perdida genetica de ciertas subunidades de TFIIH.. Xeroderma
pigmentoso, retencin de crecimiento, fotosensibilidad desordenes
neurolgicos.
La RNA polimerasa Dependiente de DNA puede sufrir Inhibicin
selectiva
La elongacin de la transcripcin en bacterias y eucariotes es inhibida por
el antibitico Actinomicina D. la porcin planar de la molcula se intercala
entre pares sucesivos GC deformando el DNA, evitando el movimiento de
la polimerasa a lo largo del templado. Util en para identificar procesos
celulares que dependen de la sntesis de RNA. Acrididina acta similar.
Rifampicina inhibe la sntesis de RNA bacterial, se une a la subunidad de
la RNApol bacterial, evitando el clearence del promotor.
El hongo Amanita phalloides, produce -amanitina bloquea la Pol II, y en
altas concentraciones a Pol III. Ni Pol I, ni la Pol bacterial , ni la Pol II del
hongo son sensibles.
Procesamiento de RNA
Muchos RNAs de bacterias y todos las de eucariotes son procesadas en
algn grado despus de su sntesis. Muchas de las enzimas son RNAs,
Ribozimas.
El RNA recin sintetizado se denomina Transcripto primario, los
procesamientos ms extensivos se dan en mRNAs eucariotes y tRNAs de
bacterias y eucariotes.
El trascripto primario de un mRNA eucariote contiene secuencias que
comprenden todo el gen, aunque las secuencias que codifican el polipptido
no estn contiguas, los fragmentos no codantes que interrumpen los
segmentos codantes son llamados intrones mientras que los segmentos
codantes son los exones.
Un proceso llamado splicing remueve los intrones y los exones se unen para
formar una secuencia continua que especifica un poli pptido funcional
Los mRNAs eucariticos son tambin modificados en los extremos. Se
adiciona residuo modificado 5cap. El extremo 3es clivado y se adicionan
80 a 250 A cola poli A.
Los complejos de protenas que llevan a cabo estos tres eventos muchas
veces no actan en forma independiente sino estn organizadas entre
ellas y con el CTD fosforilado de la Pool II, cada complejo afecta la
accin de los otros.
Otras protenas que transportan el mRNA al citoplasma, tambin estn
asociadas con el mRNA en el ncleo, asi, el procesamiento del
transcripto est asociado con su transporte.
mRNA eucariote conforme es sintetizado, forma un complejo con una
docena de protenas, la composicin del complejo cambia conforme el
transcrito es procesado, transportado al citoplasma y conducido al
ribosoma
Tres eventos del procesamiento
tRNAs de procariotes y eucariotes, se procesan por clivajes en cada
extremo y en pocos casos por splicing, muchos azucares y bases tambin
son modificados,
G NH2
Finalmente, los RNA sufren una completa y regulada degradacin,
la velocidad de recambio de RNAs, es determinante en su St St. Y en la
velocidad en la cual apagan la expresin de un gen cuyo producto ya no
se requiere.
Durante el desarrollo de organismos multicelulares, por ejemplo, ciertas
protenas deben ser expresadas nicamente en un estado, y el mRNA que
es codificado para tal protena debe ser producido y degradado en un
tiempo apropiado.
Los mRNAs eucariotes, adicionan un casco
en el extremo 5.
El cap se forma por condensacin de GTP
con el extremo 3trifosfato del transcripto,
Luego la guanina es metilada en N-7, grupos
adicionales de metilos se adicionan en OH 2
del primer y segundo Nu.
Los Metilos derivan del S-adenosilmetionina,
todas estas reaciones se producen muy
tempranamente en la transcripcin, despus
que los primeros 20 - 30 Nu han sido
adicionados.
Las 3 enzimas capping y a travs de ellas el
extremo 5 del transcripto mismo estn
asociadas con la CTD de la RNA polimerasa II
hasta que el cap es sintetizado. El extremo 5
con su casquete, es liberado de las enzimas
capping y unido a CTD por el complejo que une
el cap (CBC).
You might also like
- Metabolismo Del ArnDocument9 pagesMetabolismo Del ArnNathalie CortezNo ratings yet
- PDF Triptico Tamiz NeonatalDocument2 pagesPDF Triptico Tamiz NeonatalMeliza MeraNo ratings yet
- Investigación y Confirmación de Brotes-2Document17 pagesInvestigación y Confirmación de Brotes-2Leticia ConstantinoNo ratings yet
- Cap. 8 - Psicología y Promoción Salud El Caso de La Alimentación InfantilDocument82 pagesCap. 8 - Psicología y Promoción Salud El Caso de La Alimentación InfantilAnto CabonaNo ratings yet
- Fisiologia FetalDocument13 pagesFisiologia Fetalmanibea100% (1)
- Aminas SimpaticomimeticasDocument13 pagesAminas SimpaticomimeticasCarolina F' RochaNo ratings yet
- Las Paperas o Parotiditis Epidemica PDFDocument7 pagesLas Paperas o Parotiditis Epidemica PDFmanuelNo ratings yet
- Hdne Giardia LambliaDocument2 pagesHdne Giardia LambliaCarlos ChavezNo ratings yet
- Norma 197Document7 pagesNorma 197Karla Andrea JonasNo ratings yet
- 3er Tema Enfermedades Agudas Transmisibles y No Transmisibles.Document25 pages3er Tema Enfermedades Agudas Transmisibles y No Transmisibles.Daiane BorgoNo ratings yet
- Intolerancia A La Lactosa y Azúcares ReductoresDocument21 pagesIntolerancia A La Lactosa y Azúcares ReductoresMarianaRincon0% (1)
- Los Encimas Como Sitios de Acción de Los FármacosDocument5 pagesLos Encimas Como Sitios de Acción de Los FármacosDOminique Venemeti100% (1)
- La Tríada Ecológica ?Document8 pagesLa Tríada Ecológica ?Karen MacuareNo ratings yet
- Historia Natural de La Toxoplasmosis 1Document20 pagesHistoria Natural de La Toxoplasmosis 1AngelNZNo ratings yet
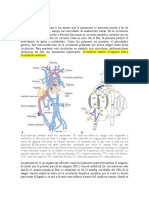
- Circulación Fetal - PediatriaDocument5 pagesCirculación Fetal - Pediatriaomar duvan vargas moralesNo ratings yet
- Sondas VesicalesDocument17 pagesSondas VesicalesAbdelaziz El Aakrout Kaamouchi100% (1)
- Triptico-Necator AmericanusDocument2 pagesTriptico-Necator AmericanusANdriid Lopez ClavijoNo ratings yet
- PulicosisDocument3 pagesPulicosisjackriden100% (1)
- Fagocitosis (Microbiología)Document3 pagesFagocitosis (Microbiología)Andrea Huerta EscobedoNo ratings yet
- ÓRGANO DIANA - Qué Es, Definición y EjemplosDocument7 pagesÓRGANO DIANA - Qué Es, Definición y EjemplosEnrique AlvarezNo ratings yet
- ARO GinecoDocument39 pagesARO GinecoHellen GordilloNo ratings yet
- Ciclo Vital de La Familia y El IndividuoDocument24 pagesCiclo Vital de La Familia y El IndividuoRommicitaNo ratings yet
- Clasificación CatéteresDocument7 pagesClasificación CatéteresDeysisita Ruiz CamposNo ratings yet
- Caso Clínico 2Document5 pagesCaso Clínico 2ErickAlvarezNo ratings yet
- Valoracion de La EmbarazadaDocument14 pagesValoracion de La Embarazadaerika2609No ratings yet
- Signos Vitales POR GRUPO ETAREODocument43 pagesSignos Vitales POR GRUPO ETAREOROSSY VARGASNo ratings yet
- Atencion Inmediata Al Recien NacidoDocument18 pagesAtencion Inmediata Al Recien NacidoEnilce Jessica CruzNo ratings yet
- AnencefaliaDocument13 pagesAnencefaliaGISELA ORTIZNo ratings yet
- Manual de VacunaciónDocument29 pagesManual de VacunaciónVillarreal Galán Andrea Jazmin100% (1)
- PlaceDocument3 pagesPlacejudith velzNo ratings yet
- Desinfección de La Sala QuirúrgicaDocument18 pagesDesinfección de La Sala QuirúrgicaCristhian De Jesus Mendez HernandezNo ratings yet
- Diágrama de FlujoDocument3 pagesDiágrama de FlujoKaren Briseño CabreraNo ratings yet
- Place EclampsiaDocument2 pagesPlace EclampsiaVioleta AlmarazNo ratings yet
- Historia Natural y Social de La ParasitosisDocument5 pagesHistoria Natural y Social de La Parasitosisrene clausi salazar100% (1)
- FAMILIOGRAMADocument32 pagesFAMILIOGRAMAAlfonsoSaldañaChuquizutaNo ratings yet
- De Acuerdo Con La OMSDocument7 pagesDe Acuerdo Con La OMSHeberVelasquezNo ratings yet
- Nom 003 Ssa2 1993Document20 pagesNom 003 Ssa2 1993César Serrano MaciasNo ratings yet
- Historia Natural Embarazo EctópicoDocument3 pagesHistoria Natural Embarazo EctópicoYolanda Paola MuvdiNo ratings yet
- Selección de Datos para La VigilanciaDocument22 pagesSelección de Datos para La VigilanciaKathyMartínezNo ratings yet
- Seminario Metabolismo ADN 2016Document51 pagesSeminario Metabolismo ADN 2016Melanie TassoNo ratings yet
- Llinea Del Tiempo Cuidado Paliativo - OrganizedDocument3 pagesLlinea Del Tiempo Cuidado Paliativo - OrganizedVanesa FloresNo ratings yet
- Mapa ConceptualDocument2 pagesMapa Conceptualapi-472592451No ratings yet
- Museo de Medicina EnsayoDocument4 pagesMuseo de Medicina EnsayoMichelle MarinNo ratings yet
- Unam Practica Gasometria y DialisisDocument14 pagesUnam Practica Gasometria y DialisisJesus CervantesNo ratings yet
- Historia Clinica FORMATO BlancoDocument6 pagesHistoria Clinica FORMATO BlancoNayro Gaym Gaza CortesNo ratings yet
- Tira HorariaDocument15 pagesTira HorariaTriny ValdezNo ratings yet
- Control TermicoDocument22 pagesControl TermicoGabriela Castillo González100% (1)
- Norma Oficial Mexicana NOM-007 - SSA2-2016, para La Atención de La Mujer Durante El Embarazo, Parto y Puerperio, y de La Persona Recién Nacida.Document28 pagesNorma Oficial Mexicana NOM-007 - SSA2-2016, para La Atención de La Mujer Durante El Embarazo, Parto y Puerperio, y de La Persona Recién Nacida.IRMA ADELAIDA ACOSTA CASTRONo ratings yet
- Detección Temprana y Atención Oportuna de Enfermedades CongenitasDocument15 pagesDetección Temprana y Atención Oportuna de Enfermedades CongenitasRendón Magali100% (1)
- Neumococo 13 y 23Document2 pagesNeumococo 13 y 23Carlos VazquezNo ratings yet
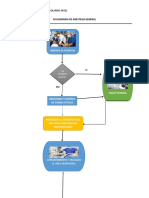
- FLUJOGRAMA DE ANESTESIA GENERAL Univ GUZMAN FLORES ROLANDO JHOEL 3DDocument4 pagesFLUJOGRAMA DE ANESTESIA GENERAL Univ GUZMAN FLORES ROLANDO JHOEL 3DRolando Jhoel Guzman100% (1)
- Depresión PlaceDocument11 pagesDepresión Placekaren jennifer chavez jimenezNo ratings yet
- Cronograma de Actividades EnfemeriaDocument2 pagesCronograma de Actividades Enfemeriaeric jesusNo ratings yet
- Epidemiologia de Herpes GenitalDocument4 pagesEpidemiologia de Herpes GenitalIngrid CanizNo ratings yet
- 2 Cuidados Del Neonato en IncubadoraDocument21 pages2 Cuidados Del Neonato en IncubadoraLuis Palacios0% (1)
- Historia Natural de La ApendicitisDocument8 pagesHistoria Natural de La ApendicitisMaríaFernandaVelascoNo ratings yet
- Cuadro Comparativo MicrobiologiaDocument3 pagesCuadro Comparativo MicrobiologiaMonica PerdomoNo ratings yet
- Introduccion A La PediatriaDocument22 pagesIntroduccion A La PediatriaDaiiana CentenoNo ratings yet
- Proceso de TranscripciónDocument17 pagesProceso de TranscripciónKarina Alejandra Cervantes RoblesNo ratings yet
- ARN y Sus CaracterísticasDocument41 pagesARN y Sus CaracterísticasNazareth EspinozaNo ratings yet
- Cuestionario 05 - Bioca PrácticaDocument5 pagesCuestionario 05 - Bioca PrácticaMarjorie Lizbeth P.No ratings yet
- Taller de Replicacion y TraduccionDocument4 pagesTaller de Replicacion y TraduccionNataliaCharryNo ratings yet
- Resumenes de BBM 2021 Por Denise BourlotDocument270 pagesResumenes de BBM 2021 Por Denise BourlotEmylyn AndradeNo ratings yet
- Las BiomoléculasDocument25 pagesLas BiomoléculasDe Val RickNo ratings yet
- Secuenciación de ProteínasDocument13 pagesSecuenciación de ProteínasManuel A. VelazNo ratings yet
- Resumen Capitulo 4 LehningerDocument4 pagesResumen Capitulo 4 LehningerÉrick JuárezNo ratings yet
- Nucleotidos y NucleoprotreinasDocument38 pagesNucleotidos y NucleoprotreinasNazareth LucianaNo ratings yet
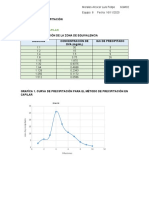
- Informe de Laboratorio Actividad EnzimaticaDocument6 pagesInforme de Laboratorio Actividad EnzimaticaLUIS ANGEL LLANOS ACUÑANo ratings yet
- 20 Tipos de Proteínas y Sus Funciones en El CuerpoDocument1 page20 Tipos de Proteínas y Sus Funciones en El CuerpoGuereca Montelongo Andrea100% (1)
- Examen de BioquimicaDocument2 pagesExamen de BioquimicaYuMi BautistaNo ratings yet
- Factores Físicos y Químicos Que Alteran La Función y Estructura de Las ProteínasDocument3 pagesFactores Físicos y Químicos Que Alteran La Función y Estructura de Las ProteínasCristian Contreras100% (2)
- Bioquimica Pensum IVICDocument1 pageBioquimica Pensum IVICMiguel León RoperoNo ratings yet
- Diagrama de LanzaderasDocument1 pageDiagrama de LanzaderasedipsonNo ratings yet
- Taller Sintesis de Proteinas 2020Document3 pagesTaller Sintesis de Proteinas 2020Luis Blanco Blanco gomez100% (1)
- Metabolismo de Aminoacidos y ProteinasDocument49 pagesMetabolismo de Aminoacidos y Proteinasjuan100% (1)
- 15 Preguntas Acerca de Las ProteínasDocument4 pages15 Preguntas Acerca de Las ProteínasAndresfdezcmc94% (47)
- Reaccines de PrecipitaciónDocument4 pagesReaccines de PrecipitaciónLuis AlcocerNo ratings yet
- Test Diagnóstico de BioquímicaDocument5 pagesTest Diagnóstico de BioquímicaAbril Tames FerrufinoNo ratings yet
- Genética UCSUR - T1 - Bases Moleculares de La HerenciaDocument57 pagesGenética UCSUR - T1 - Bases Moleculares de La HerenciaDiego Cruzado oroscoNo ratings yet
- ADN, Genes y Código GenéticoDocument6 pagesADN, Genes y Código GenéticomoreNo ratings yet
- CASEÍNADocument9 pagesCASEÍNAmjambrinaNo ratings yet
- Programa Diseño de BioproductosDocument3 pagesPrograma Diseño de BioproductosLauraNo ratings yet
- Biosíntesisde Ácidos GrasosDocument88 pagesBiosíntesisde Ácidos GrasosUSMP FN ARCHIVOSNo ratings yet
- Preguntas ProteinasDocument2 pagesPreguntas ProteinasandreaNo ratings yet
- Guia 5 TAP BiologíaDocument9 pagesGuia 5 TAP Biología7ndym5dj8vNo ratings yet
- Química BiológicaDocument35 pagesQuímica BiológicaNeira Osorio100% (1)
- Proteosomas y UbiquitinaDocument23 pagesProteosomas y UbiquitinaGabriela Rodriguez MuñozNo ratings yet
- Biomoleculas David Velasco Tarea 3Document6 pagesBiomoleculas David Velasco Tarea 3David GoyesNo ratings yet
- Anual SM Semana 03 - BiologíaDocument19 pagesAnual SM Semana 03 - BiologíaGeordan SanchezNo ratings yet
- Membrana PlasmáticaDocument5 pagesMembrana PlasmáticaMazuey CroissantsNo ratings yet